ARTÍCULO ORIGINAL
doi: http://dx.doi.org/10.7705/biomedica.v36i0.2957
1 Grupo de Virología, Dirección de Redes en Salud Pública, Subdirección Laboratorio Nacional de Referencia, Instituto Nacional de Salud, Bogotá, D.C., Colombia
2 Facultad de Ciencias, Departamento de Biología, Universidad Nacional de Colombia, Bogotá, D.C., Colombia
Institución en donde se realizó el estudio: Laboratorio de Polio/EV/entéricos, Grupo de Virología, Instituto Nacional de Salud, Bogotá, D.C., Colombia
Contribución de los autores:
Dioselina Peláez: diseño del estudio, análisis e interpretación de los datos, discusión de los resultados
Lady Contreras-Gómez: diseño del estudio
Daniel Martínez-Vargas: ensayos moleculares, análisis filogenético, análisis e interpretación de los datos, discusión de los resultados y redacción del manuscrito
Mariel Palacios-Vivero y Martha Escalante-Mora: ensayos serológicos
Recibido: 03/07/15; aceptado: 03/12/15
Introducción. El virus de la hepatitis E se ha convertido en un problema de salud pública, especialmente en los países en desarrollo. Se conocen cuatro genotipos en mamíferos, de los cuales el G3 se ha encontrado en hepatitis autóctonas en países y regiones con gran población de cerdos, y el G1 se ha asociado a muertes maternas.
Objetivo. Determinar la infección simultánea con el virus de la hepatitis E y sus genotipos circulantes en Colombia en 1.097 sueros utilizando los marcadores serológicos de los virus de las hepatitis A, B y C.
Materiales y métodos. Se seleccionaron 1.097 sueros provenientes de diferentes municipios de Colombia, conservados en el Laboratorio de Virología del Instituto Nacional de Salud. Se determinaron los anticuerpos IgG e IgM anti-hepatitis E. A los positivos se les amplificó el genoma viral mediante reacción en cadena de la polimerasa convencional. Los productos se secuenciaron y analizaron filogenéticamente y se los comparó con las secuencias del ORF2 registradas en el GenBank.
Resultados. Se identificaron 278 sueros positivos para IgG anti-hepatitis E, 62 para IgM y 64 para ambos marcadores. La infección simultánea con los virus de la hepatitis E y la hepatitis A determinada por IgG anti-hepatitis E fue de 33,6 % y por IgM anti-hepatitis E fue de 16,1 %; la infección simultánea por los virus de la hepatitis E y B fue de 23,4 % y 8,1 %, y por los virus de la hepatitis E y C fue de 35,4 % y 5,83 %, respectivamente. De las 52 muestras positivas en la reacción en cadena de la polimerasa convencional, nueve secuencias se agruparon como genotipo 3a de origen porcino, cepa norteamericana.
Conclusiones. La mayor seropositividad se registró para las hepatitis A y E. La frecuencia de la infección simultánea con el virus de la hepatitis E y otros virus hepatótropos indica que este patógeno puede ser más frecuente de lo esperado. La circulación del genotipo 3a implica que esta enfermedad puede presentarse en forma de brote y de zoonosis en Colombia.
Palabras clave: virus de la hepatitis E, hepatitis A, genotipo, coinfección.
doi: http://dx.doi.org/10.7705/biomedica.v36i0.2957
Coinfection of hepatitis E virus and other hepatitis virus in Colombia and its genotypic characterization
Introduction: Hepatitis E virus has emerged as a public health problem, particularly in developing countries. The four genotypes identified in mammals include the G3 found in indigenous hepatitis in countries and regions with high porcine population, and the G1, associated with maternal deaths.
Objective: To determine coinfection by hepatitis E virus and the circulating genotypes in Colombia in 1,097 samples using serological markers for hepatitis A, B and C.
Materials and methods: Serum samples of 1,097 patients from different regions of Colombia stored at the Laboratorio de Virología of the Instituto Nacional de Salud were selected to detect IgG and IgM anti-hepatitis E virus antibodies. The viral genomes of positive samples were amplified by RT-PCR, and the products were sequenced and phylogenetically analyzed by comparing ORF2 sequences deposited in the GenBank.
Results: IgG anti-hepatitis E virus antibodies were found in 278 samples, IgM in 62, and both markers in 64. Hepatitis E virus and hepatitis A virus coinfection determined by IgG anti-hepatitis E virus was 33.6% and 16.1% by IgM; hepatitis E virus and hepatitis B virus coinfection was 23.4% and 8.1%, and hepatitis E virus and hepatitis C virus coinfection was 35.4% and 5.83%, respectively. Among the 52 positive samples by PCR nine were sequenced and grouped within genotype 3A of the American porcine strain.
Conclusions: The highest seropositivity was observed for hepatitis A and E. The incidence of hepatitis E virus coinfection with other hepatotropic viruses indicated that this pathogen is more frequent than expected. The circulation of genotype 3A implies that this disease may occur in outbreaks and as zoonosis in Colombia.
Key words: Hepatitis E virus, hepatitis A, genotype, coinfection.
doi: http://dx.doi.org/10.7705/biomedica.v36i0.2957
El virus de la hepatitis E (HEV) es el agente causal de la hepatitis E, enfermedad que se transmite por vía entérica en muchas regiones del mundo y es responsable de más de 50 % de los casos de hepatitis aguda en los países endémicos (1). El virus se contagia principalmente por la vía fecal-oral y se asocia con infecciones esporádicas en los países en desarrollo, en tanto que en los países industrializados la transmisión es principalmente zoonótica, por su presencia en cerdos; los brotes epidémicos ocurren en áreas con infraestructuras sanitarias deficientes y se asocian con el consumo de agua contaminada (2). La infección durante el embarazo merece especial cuidado, ya que provoca cuadros de hepatitis fulminante con hasta 20 % de letalidad (3).
El VHE pertenece a la familia Hepeviridae, subfamilia Hepevirus, y es el único miembro del género Orthohepevirus ; la especie tipo es Orthohepevirus A , comúnmente conocido como virus de la hepatitis E (4). El virus posee un genoma de ARN de cadena simple y de sentido positivo, su tamaño es de 7,2 kb y cuenta con tres marcos abiertos de lectura (5,6). Se han reportado cuatro genotipos del HEV en mamíferos: los genotipos 1 y 2, exclusivos de los seres humanos, de los cuales el genotipo 1 es la principal causa de hepatitis E esporádica y epidémica en las regiones en desarrollo de Asia y África. En Suramérica se han reportado casos aislados en Venezuela, Uruguay y Argentina (7). El genotipo 2 se ha identificado en pacientes en México, Chad y Nigeria (6,8,9). El genotipo 3 se ha encontrado en casos de hepatitis autóctona en muchas regiones desarrolladas y tiene una alta prevalencia en poblaciones de cerdos en todo el mundo (10). El genotipo 4 se ha identificado en poblaciones humanas y de cerdos en regiones industrializadas de Japón, China, Taiwán e India (11), así como en Europa central (12-14). Aunque se han establecido varios subtipos dentro de estos cuatro genotipos, en algunos análisis recientes se sugiere que no es posible establecer límites concretos para distinguirlos de forma sistemática (15,16), sin embargo, tales categorías pueden llegar a ser útiles en los estudios epidemiológicos (17).
La infección con múltiples virus hepatótropos y en varias combinaciones se presenta en 7 a 24 % de los casos de hepatitis viral aguda esporádica (18). Un estudio en India reportó que la infección simultánea por el HEV y el virus de la hepatitis B (HBV) detectada mediante ELISA llegó a ser de 18 %, y, mediante reacción en cadena de la polimerasa (PCR) convencional, de 6,25 %, lo cual indica que puede ocurrir en varias enfermedades hepáticas (19). La tasa de infección por el HEV y el virus de la hepatitis A (HAV) puede llegar a ser de 11,5 %, lo cual hace necesaria la detección del HEV para la planeación de estrategias de vacunación y para mejorar los programas sanitarios (20).
En Colombia hay poca información sobre la presencia del HEV (21,22) (Rendón J, Navas M, Hoyos M, Cortés F, Correa G, Sepúlveda M, et al . Evidencia serológica y molecular de la circulación del virus de la hepatitis E en Medellín. Infectio. 2010;14(Supl.1):34. Memorias, VII Encuentro Nacional de Investigación en Enfermedades Infecciosas). Por esta razón, el objetivo de este estudio fue detectar serológica y molecularmente la infección simultánea por el HEV y otros virus hepatótropos (HAV, HBV, HCV), en sueros con diagnóstico positivo para HAV mediante la detección de IgM (+), para HBV mediante la detección del antígeno de superficie de la hepatitis B (HBsAg +), y para el HCV mediante ensayo de inmunotransferencia recombinante ( recombinant immunoblot assay , RIBA +), y caracterizar el genotipo del HEV que circula en Colombia.
Materiales y métodos
Tipo de estudio
Se hizo un estudio descriptivo y retrospectivo en sueros de pacientes con diagnóstico positivo para hepatitis virales.
Selección de las muestras
Se seleccionaron muestras a conveniencia con marcadores serológicos que demostraban una infección activa para hepatitis virales: IgM anti-HAV (hepatitis A), HBsAG (hepatitis B) y RIBA para HCV (hepatitis C). Estos sueros provenientes de los 32 departamentos de Colombia, se recolectaron durante el periodo 2004-2014 y se conservaron a -25 °C en el Laboratorio de Virología del Instituto Nacional de Salud. Se excluyeron los sueros lipémicos o con hemólisis, así como las muestras con una cantidad insuficiente de suero.
Pruebas serológicas
Con base en los criterios de inclusión, se analizaron 1.097 muestras de suero para la detección de anticuerpos IgG e IgM anti-HEV mediante ELISA de tercera generación, usando kits comerciales (Dia.Pro, Diagnostic Bioprobes Srl, Milán, Italia) y siguiendo las instrucciones del fabricante.
Extracción del ARN viral
El ARN viral de los sueros positivos para alguno de los marcadores serológicos anti-HEV se extrajo usando el QIAamp Viral RNA Mini Kit ® (Qiagen, Brazil) de acuerdo con las instrucciones del fabricante. El ARN aislado se almacenó a -30 °C hasta su uso posterior.
Transcripción inversa seguida de reacción en cadena de la polimerasa anidada
El ARN del HEV se detectó mediante reacción en cadena de la polimerasa anidada (nRT-PCR), usando dos sets de iniciadores que amplificaban fragmentos de las regiones de los genes ORF1 y ORF2 (23), y un set que amplificaba una región del ORF1 (24).
Los iniciadores para el ORF1 (23), que flanquean las posiciones 56-79 y 473-451 del genoma, corresponden a la región que codifica para la metiltransferasa (MeT) y amplifican un fragmento de 418 pb. Los iniciadores externos (ConsORF1-s1: 5´-CTGGCATYACTACTGCYATTGAGC-3´ y ConsORF1-a1: 5´-CCATCRARRCAGTAAGTGCG GTC-3´) y los internos (ConsORF1-s2: 5´-CTGC CYTKGCGAATGCTGTGG-3´ y ConsORF1-a2: 5´-GGCAGWRTACCARCGCTGAACATC-3´) amplifican un fragmento de 287 pb.
Los iniciadores para el ORF1 (24) amplificaron la región 4254-4560 del genoma viral, correspondiente a la región de la polimerasa (RdRp) de una longitud de 307 pb: ESP 4213–4235 5´-CATGGTAAAGTGGGTCAGGGTAT-3´. ISP 4232–4253 5´-GTATTTCGGCCTGGAGTAAGAC-3´. IAP 4561–4583 5´-TCACCGGAGTGYTTCTTCCAGAA -3´. EAP 4576–4595 5´-AGGGTGCCGGGCTCG CCGGA-3´.
Los iniciadores para el ORF2 corresponden a la posición 6298-6321 y 6494-6470 del prototipo Burma (código de acceso M73218 en el Gen Bank). Los iniciadores externos (ConsORF2-s1: 5´-GACAGAATTRATTTCGTCGGCTGG-3´ y ConsORF2- a1: 5´-CTTGTTCRTGYTGGTTRTCAT AATC-3´) amplificaron un producto de 197 pb en la primera PCR, y los internos (ConsORF2-s2: 5´-GTYGTCTCRGCCAATGGCGAGC-3´ y ConsORF2-a2: 5´-GTTCRTGYTGGTTRTCATAA TCCTG-3´) amplificaron un producto de 145 pb en la segunda PCR.
En la primera ronda de PCR se utilizó Qiagen OneStep RT-PCR Kit ® (Qiagen, Brazil), con un volumen final de reacción de 25 µl: 5 µl de solución tampón 5X RT-PCR, 1 µl de dNTP en concentración de 10 mM, 1 µl de cada iniciador en concentración de 10 µM, 1 µl de la mezcla de enzimas y 3 µl de ARN. En la segunda PCR se obtuvo un volumen final de 25 µl con los siguientes reactivos: 2,5 µl de solución tampón 10X PCR (Invitrogen); 0,5 µl de dNTP en concentración de10 mM (Invitrogen); una unidad de Platinum Taq polymerase (Invitrogen); 1 µl de cada iniciador en concentración de 10 µM; 0,75 µl de MgCl 2 en concentración de 50 mM (Invitrogen) y 2 µl del producto amplificado de la primera PCR.
Las condiciones para la primera PCR fueron las siguientes: un ciclo de transcripción inversa a 50 °C durante 30 minutos seguido por un paso de inactivación/activación a 95 °C durante 15 minutos; 35 ciclos de desnaturalización a 94 °C durante 30 segundos, de anillamiento a 50 °C durante 30 segundos, de extensión a 72 °C durante 30 segundos, y una extensión final a 72 °C durante 6 minutos. La segunda PCR tuvo las siguientes condiciones: una desnaturalización inicial a 94 °C durante 2 minutos, 35 ciclos de desnaturalización a 94 °C durante 30 segundos, de anillamiento a 50 °C durante 30 segundas, de extensión a 72 °C durante 30 segundos, y una extensión final a 72 °C durante 6 minutos.
Los productos se marcaron con SYBR Safe ® (Invitrogen), se separaron mediante electroforesis en gel de agarosa al 2 % y se visualizaron en un fotodocumentador (Bio-Rad, Gel Doc TM XR+ System).
Secuenciación y análisis filogenético
Los productos positivos de PCR para el ARN del VHE se purificaron mediante el QIAquick PCR Purification Kit ® (Qiagen, Brazil), de acuerdo con las instrucciones del fabricante, y se secuenciaron usando el BigDye Terminator ® , v 3.1 Cycle Sequencing Kit (Applied Biosystems, CA, USA), según las instrucciones del fabricante en un secuenciador ABI 3130 (Applied Biosystem).
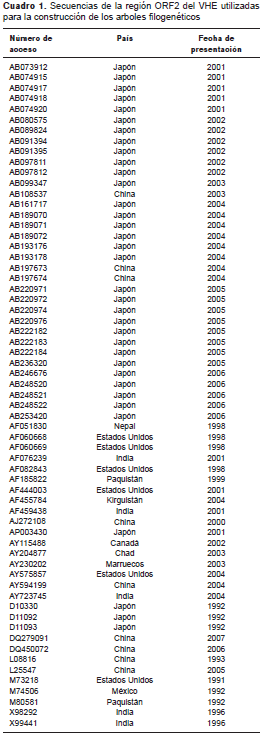
Cada electroferograma se analizó y se editó usando el programa BioEdit (v. 7.2.5) para obtener la secuencia consenso a partir de las secuencias hacia adelante y hacia atrás. De la base de datos del GenBank (http://www.ncbi.nlm.nih.gov/) se seleccionaron 63 secuencias de 141 pb correspondientes a la región ORF2 de VHE de distintos genotipos y diferentes partes del mundo (cuadro 1), y se alinearon con las secuencias obtenidas utilizando el programa Clustal Omega disponible en la página web del European Bioinformatics Institute (EMBL-EBI) (http://www.ebi.ac.uk/Tools/msa/clustalo/). En ninguno de los países de Suramérica se encontraron secuencias del HEV G3 de la región ORF2 amplificada con los iniciadores utilizados en este estudio.

El análisis filogenético se hizo mediante el método estadístico neighbor-joining en el programa MEGA6 (v. 6.06), utilizando un modelo de distancias-p. Se hizo un análisis filogenético bayesiano adicional usando el programa MrBayes (v. 3.2.5) y muestreando a lo largo de todo el espacio general de tiempo reversible ( General Time Reversible , GTR) en un total de diez millones de generaciones.
Resultados
De los 1.097 sueros procesados, 278 fueron positivos para IgG anti-HEV, 62 para IgM anti-HEV y 64 para ambos marcadores. Considerando que 342 sueros fueron positivos para IgG anti-HEV y 126 para IgM anti-HEV, la seropositividad general para IgG anti-HEV fue de 31,2 % y 11,5 % para los anticuerpos IgM anti-HEV. La infección por HEV y HAV determinada por IgG anti-HEV, fue de 33,6 % y de 16,1 % por IgM anti-HEV; para el HBV fue de 23,4 % y 8,1 % y para el HCV, de 35,4 % y 5,8 %, respectivamente (cuadro 2).

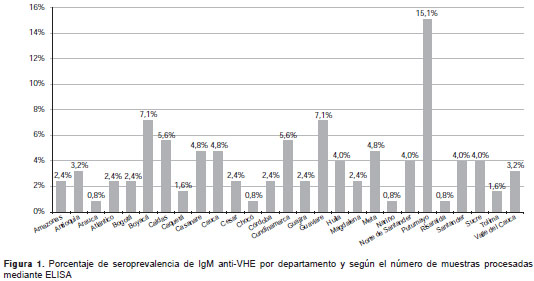
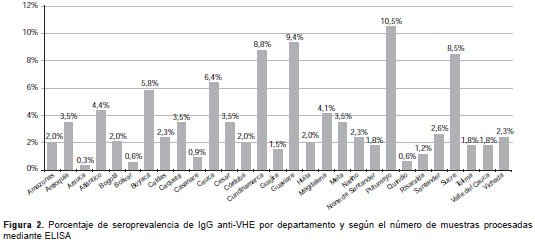
El análisis de la variable de edad mostró que 48 % de la infección simultánea por HAV y HEV, determinada por la presencia de IgM anti-HAV e IgM anti-HEV, se produjo antes de los 16 años de edad, 9 % entre los 16 y los 30 años de edad, y 41 % de los casos no registraba información sobre la edad. La infección simultánea por HBV y HEV, determinada por la presencia del HBsAg y de IgM anti-HEV, mostró que 28 % de los casos se produjo en el grupo de 2 a 15 años de edad, 32 % de 16 a 30 años, 8 % de 46 a 70 años y en 32 % de los casos no había información con respecto a la edad. Los departamentos con mayor presencia de anticuerpos anti-IgG e IgM anti-HEV fueron Putumayo, Guaviare, Boyacá y Cundinamarca (figura 1 y figura 2).
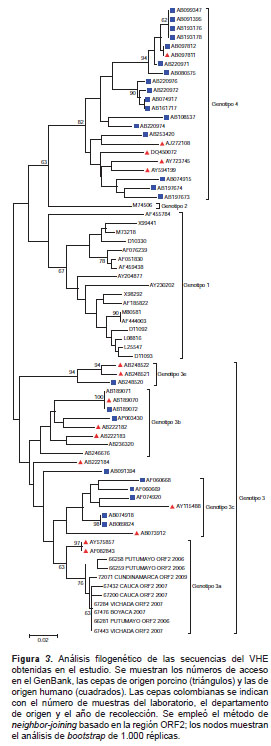
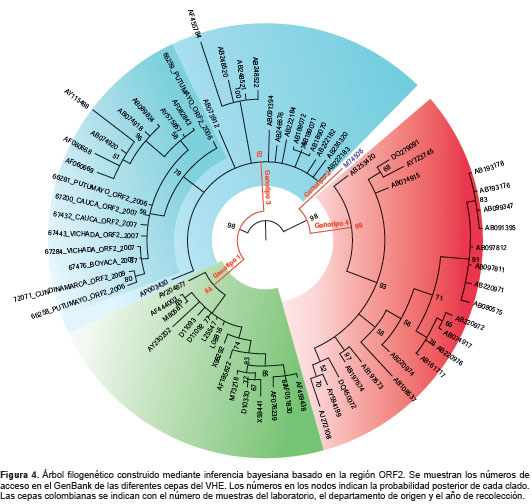
De las 119 muestras positivas para IgG anti-HEV, las tres para IgM anti-HEV y las 59 para ambos marcadores, analizadas mediante PCR convencional para la detección del ARN viral, 52 fueron positivas y, de estas, nueve se secuenciaron y se encontró que correspondían al genotipo 3a de cepas norteamericanas de origen porcino (figura 3 y figura 4). Estos nueve virus caracterizados se detectaron en sueros de pacientes proce-dentes de los departamentos de Boyacá, Cauca, Cundinamarca, Putumayo y Vichada, recolectados en los años 2006, 2007 y 2009.
Discusión
En estudios previos ya se había detectado la circulación del HEV en el departamento de Antioquia (Rendón J, Navas M, Hoyos M, Cortés F, Correa G, Sepúlveda M, et al . Evidencia serológica y molecular de la circulación del virus de la hepatitis E en Medellín. Infectio. 2010;14(Supl.1):34. Memorias, VII Encuentro Nacional de Investigación en Enfermedades Infecciosas) y en otros departamentos de Colombia (22); sin embargo, no existían estudios sobre la infección concomitante del HEV y otros virus hepatótropos en el país. En este estudio se logró detectar la infección simultánea por el HEV y los virus hepatótropos HAV, HBV y HCV mediante la detección de IgM anti-HEV, así como la infección concomitante con dichos virus determinada por la presencia de IgG anti-HEV. Los resultados muestran que uno de los mayores porcentajes de infección concomitante se registró entre el HEV y el HAV, lo cual se explica por la similitud de las características epidemiológicas de ambos virus. Los resultados obtenidos respaldan la hipótesis de un posible origen común de la transmisión por aguas o alimentos contaminados (25).
Resultó sorpresivo el alto porcentaje de infección concomitante por el HEV y los virus HBV (23,5 %) y HCV (35,4 %) (cuadro 1), teniendo en cuenta que las principales vías de transmisión de estos virus son diferentes: entérica para el HEV, contacto con sangre o fluidos corporales para el HBV y parenteral para el HCV. Sin embargo, algunos estudios en India han reportado que la infección simultánea con múltiples virus hepatótropos se presentaba en varias combinaciones en 7 a 24 % de los pacientes con una hepatitis viral aguda (26); en Egipto este porcentaje fue de 52 % (27) y en Italia fue de 27 % (28). A partir de estos resultados pueden plantearse varias hipótesis: la infección por el HEV puede ocurrir en un paciente en recuperación de una infección previa por el HBV; también puede activar una infección crónica por el HBV previa; de igual manera, una infección previa con el HEV puede favorecer una nueva infección con el HCV, o puede darse una transmisión simultánea de ambos virus por transfusión sanguínea. La transmisión del HEV por transfusiones sanguíneas se ha documentado en varios continentes (29-34), por lo que es posible una transmisión parenteral simultánea de HBV y HEV, y de HCV y HEV, lo cual sugiere la transmisión de estos virus por vías similares o coincidentes.
Hasta el momento no se han recabado datos sobre la seroprevalencia del HEV en la población general en Colombia, y solo se ha estimado en poblaciones con características muy definidas, de tal manera que se cuenta con una serie de reportes con datos de seropositividad que varían entre 7,5 y 11,25 % para IgG anti-HEV y entre 1,74 y 46 % para IgM anti-HEV (21,22) (Rendón J, Navas M, Hoyos M, Cortés F, Correa G, Sepúlveda M, et al . Evidencia serológica y molecular de la circulación del virus de la hepatitis E en Medellín. Infectio. 2010;14(Supl.1):34. Memorias, VII Encuentro Nacional de Investigación en Enfermedades Infecciosas). Los resultados de este estudio evidenciaron una seropositividad de 31,2 % para IgG anti-HEV y de 11,5 % para IgM anti-HEV, lo cual indica que la exposición al HEV es alta en la población de estudio y que la infección subclínica por el HEV en Colombia puede llegar a ser más común de lo que se suponía con base en los estudios previos. Con el porcentaje detectado de hepatitis E aguda se puede afirmar que esta enfermedad no se reconoce en nuestro país, probablemente porque no se la contempla en el diagnóstico de pacientes con hepatitis no-A, no-B y no-C.
Según el rango de edad, 48 % de la infección simultánea con HAV y HEV se produjo en el grupo de personas de menos de 16 años de edad y, 9 %, en el grupo de 16 a 30 años de edad; la gran mayoría de estas infecciones se detectó en regiones con mala calidad del agua y prácticas de higiene deficientes, lo cual las convierte en un riesgo potencial de infección a una edad temprana. Se registró que 28 % de los casos de infección simultánea con HBV y HEV se produjo en el grupo de 2 a 15 años de edad, 32 %, en el de 16 a 30 años, y 8 %, en el de 46 a 70 años. La hepatitis B afecta a la población general, sin embargo, es más frecuente en los jóvenes, adultos y grupos poblacionales con factores de riesgo para la enfermedad; según los estudios más recientes, en Colombia se han encontrado prevalencias de HBsAg de 5,66 % (35), lo que concuerda con los grupos de edad que presentan mayor porcentaje de infección simultánea con HBV y HEV en este estudio, y evidencia que, dado que dicha endemia es de moderada a alta, es muy probable que una persona que presente el HBV se pueda infectar también con el HEV (27,36).
La caracterización genotípica del HEV es de gran importancia, ya que la gravedad de la hepatitis E está relacionada directamente con el genotipo del virus (15): las hepatitis fulminantes en mujeres embarazadas y las manifestaciones no hepáticas de la enfermedad, como la pancreatitis, están asociadas con el genotipo 1; la hepatitis crónica en pacientes con inmunodeficiencia se ha observado principalmente en infecciones con el genotipo 3 del HEV (37). Además, todos los casos de transfusión relacionados con la hepatitis E han sido causados por cepas del HEV de los genotipos 3 y 4, lo cual sugiere un potencial de transmisión parenteral de estos genotipos zoonóticos (38).
Por otra parte, en estudios en Colombia se ha demostrado la presencia del genotipo 3 del virus en el agua de consumo humano (Baéz P, Jaramillo CM, Arismendi L, Rendón JC, Cortés-Mancera F, Toro M, et al . Evidencia molecular de virus de la hepatitis E en fuentes de agua en Antioquia. Biomédica. 2015;35(Supl.1):40-1. Memorias, VI Simposio Colombiano de Virología), lo que restaría argumentos para pensar en una posible infección simultánea por el HEV y los virus HBV y HCV por vía parenteral. Sin embargo, es necesario adelantar nuevas investigaciones en el país sobre otras vías de transmisión del HEV diferentes de la entérica, ya que los datos obtenidos en este estudio indicarían que otra posible vía de transmisión es la parenteral, como lo han reportado estudios previos (39,40).
Los departamentos con mayor presencia de anti-cuerpos IgG e IgM anti-HEV fueron Putumayo, Guaviare, Boyacá y Cundinamarca (figura 1 y figura 2). Estas regiones se han caracterizado históricamente por su deficiente suministro de agua potable (41,42), lo cual explicaría la gran seroprevalencia no solo para anticuerpos anti-HEV, sino también, las altas tasas de hepatitis A (43). Tradicionalmente, estos también son departamentos con una gran población porcina (http://www.dane.gov.co/index.php/agropecuario-alias/estadisticas-de-sacrificio-de-ganado-esag), lo cual estaría favoreciendo el ciclo zoonótico de la enfermedad.
En conclusión, se logró determinar la infección concomitante del VHE y otros virus hepatótropos (HAV, HBV y HCV), con porcentajes de infección dual de HBV y HEV, y HCV y HEV sorpresivamente altos si se tiene en cuenta que las principales vías de transmisión de estos virus son muy diferentes (entérica y parenteral). Esto abre la posibilidad de considerar la vía parenteral como una vía de transmisión secundaria del HEV en el país. Los casos de infección simultánea con HAV y HEV pueden llegar a ser más comunes debido a que ambos virus comparten la vía de transmisión entérica, y por las deficiencias en el tratamiento del agua para consumo humano en algunos departamentos del país, donde se ha detectado la presencia de ambos virus en el agua para consumo humano.
La detección del genotipo 3 del HEV en varios departamentos del país, sumada a los hallazgos de otros estudios realizados en Colombia, confirma que este es el genotipo que está circulando, lo que concuerda con el predominante en Suramérica. La identificación del subtipo 3a abre la puerta para futuras investigaciones sobre las posibles implicaciones epidemiológicas asociadas a dicho subtipo en el país. Una vez establecida la circulación del HEV en Colombia, se hace necesario profundizar en el estudio del virus para establecer su incidencia en la población, las vías de transmisión y los efectos de la infección dual con otros virus causantes de hepatitis, con el fin de crear estrategias de control y prevención de la infección.
A la unidad de secuenciación de genomas del Grupo de Virología del Instituto Nacional de Salud y a todas las personas que de alguna manera hicieron posible este estudio.
Los autores declaran no tener conflicto de intereses.
El presente estudio fue financiado por el Instituto Nacional de Salud y por Colciencias (proyecto No. 710 de 2013, código No. 2010456935048).
Correspondencia:
Dioselina Peláez-Carvajal, Grupo de Virología, Dirección de Redes en Salud Pública, Subdirección Laboratorio Nacional de Referencia, Instituto Nacional de Salud, Avenida calle 26 N° 51-20, CAN, Bogotá, D.C., Colombia
Telefax: 220 0770, extensión 1439
1. Pérez-Gracia MT, Suay B, Mateos-Lindemann ML. Hepatitis E: An emerging disease. Infect Genet Evol. 2014;22:40-59. http://dx.doi.org/10.1016/j.meegid.2014.01.002
2. Dalton HR, Bendall R, Ijaz S, Banks M. Hepatitis E: An emerging infection in developed countries. Lancet Infect Dis. 2008;8:698-709. http://dx.doi.org/10.1016/S1473-3099(08)70255-X
3. Navaneethan U, Al Mohajer M, Shata MT. Hepatitis E and pregnancy: Understanding the pathogenesis. Liver Int. 2008;28:1190-9. http://dx.doi.org/10.1111/j.1478-3231.2008.01840.x
4. Smith DB, Simmonds P, Jameel S, Emerson SU, Harrison TJ, Meng XJ, et al. Consensus proposals for classification of the family Hepeviridae. J Gen Virol. 2014;95:2223-32. http://dx.doi.org/10.1099/vir.0.068429-0
5. Purdy MA, Khudyakov YE. Evolutionary history and population dynamics of hepatitis e virus. PLoS One. 2010;5:e14376. http://dx.doi.org/10.1371/journal.pone. 0014376
6. Tam AW, Smith MM, Guerra ME, Huang CC, Bradley DW, Fry KE, et al . Hepatitis E virus (HEV): Molecular cloning and sequencing of the full-length viral genome. Virology. 1991;185:120-31. http://dx.doi.org/10.1016/0042-6822(91)90760-9
7. Echevarría JM, González JE, Lewis-Ximénez LL, Lopes dos Santos DR, Munne MS, Pinto MA, et al . Hepatitis E virus infection in Latin America: A review. J Med Virol. 2013;85:1037-45. http://dx.doi.org/10.1002/jmv.23526
8. Buisson Y, Grandadam M, Nicand E, Cheval P, van Cuyck-Gandre H, Innis B, et al . Identification of a novel hepatitis E virus in Nigeria. J Gen Virol. 2000;81:903-9. http://dx.doi.org/10.1099/0022-1317-81-4-903
9. van Cuyck-Gandre H, Zhang HY, Tsarev SA, Clements NJ, Cohen SJ, Caudill JD, et al . Characterization of hepatitis E virus (HEV) from Algeria and Chad by partial genome sequence. J Med Virol. 1997;53:340-7.
10. Engle RE, Yu C, Emerson SU, Meng XJ, Purcell RH. Hepatitis E virus (HEV) capsid antigens derived from viruses of human and swine origin are equally efficient for detecting anti-HEV by enzyme immunoassay. J Clin Microbiol. 2002;40:4576-80. http://dx.doi.org/10.1128/JCM.40.12.4576-4580.2002
11. Lu L, Li C, Hagedorn CH. Phylogenetic analysis of global hepatitis E virus sequences: Genetic diversity, subtypes and zoonosis. Rev Med Virol. 2006;16:5-36.
12. Colson P, Brunet P, Lano G, Moal V. Hepatitis E virus genotype 4 in Southeastern France: Still around. Liver Int. 2015. http://dx.doi.org/10.1111/liv.12924
13. Monne I, Ceglie L, Di Martino G, Natale A, Zamprogna S, Morreale A, et al . Hepatitis E virus genotype 4 in a pig farm, Italy, 2013. Epidemiol Infect. 2015;143:529-33. http://dx.doi.org/10.1017/S0950268814001150
14. Hakze-van der Honing RW, van Coillie E, Antonis AF, van der Poel WH. First isolation of hepatitis E virus genotype 4 in Europe through swine surveillance in the Netherlands and Belgium. PLoS One. 2011;6:e22673. http://dx.doi.org/10.1371/journal.pone.0022673
15. Okamoto H. Genetic variability and evolution of hepatitis E virus. Virus Res. 2007;127:216-28. http://dx.doi.org/10.1016/j.virusres.2007.02.002
16. Oliveira-Filho EF, Koenig M, Thiel H-J. Genetic variability of HEV isolates: Inconsistencies of current classification. Vet Microbiol. 2013;165:148-54. http://dx.doi.org/10.1016/j.vetmic.2013.01.026
17. Dai X, Dong C, Zhou Z, Liang J, Dong M, Yang Y, et al . Hepatitis E virus genotype 4, Nanjing, China, 2001-2011. Emerg Infect Dis.2013;19:1528-30. http://dx.doi.org/10.3201/eid1909.130013
18. Poddar U, Thapa BR, Prasad A, Singh K. Changing spectrum of sporadic acute viral hepatitis in Indian children. J Trop Pediatr. 2002;48:210-3. http://dx.doi.org/10.1093/tropej/48.4.210
19. Singh A, Singh S, Ansari MA, Irshad M. Co-infectivity of hepatitis B virus and hepatitis E virus. BMC Infect Dis. 2012;12(Supl.1):P1. http://dx.doi.org/10.1186/1471-2334-12-S1-P1
20. Rao P, Shenoy MS, Baliga S, Joon A. Prevalence of HAV and HEV in the patients presenting with acute viral hepatitis. Indian J Med Microbiol. 2015;33(Suppl.):102-5. http://dx.doi.org/10.4103/0255-0857.150908
21. Betancur C, Mejía M, Portillo S. Seroprevalencia de hepatitis E en trabajadores de fincas porcícolas del Valle de Aburrá, 2011-2012. Acta Médica Colombiana. 2013;38: 68-70.
22. Peláez D, Hoyos MC, Rendón JC, Mantilla C, Ospina MC, Cortés-Mancera F, et al . Infección por el virus de la hepatitis E en pacientes con diagnóstico clínico de hepatitis viral en Colombia. Biomédica. 2014;34:354-65. http://dx.doi.org/10.7705/biomedica.v34i3.2236
23. Wang Y, Ling R, Erker JC, Zhang H, Li H, Desai S, et al . A divergent genotype of hepatitis E virus in Chinese patients with acute hepatitis. J Gen Virol. 1999;80:169-77. http://dx.doi.org/10.1099/0022-1317-80-1-169
24. Zhai L, Dai X, Meng J. Hepatitis E virus genotyping based on full-length genome and partial genomic regions. Virus Res. 2006;120:57-69. http://dx.doi.org/10.1016/j.virusres.2006.01.013
25. Rodríguez-Lay L de A, Quintana A, Montalvo-Villalba MC, Leimos G, Corredor MB, Moreno AG, et al . Dual infection with hepatitis A and E viruses in outbreaks and in sporadic clinical cases: Cuba 1998-2003. J Med Virol. 2008;80:798-802. http://dx.doi.org/10.1002/jmv.21147
26. Kumar A, Yachha SK, Poddar U, Singh U, Aggarwal R. Does co-infection with multiple viruses adversely influence the course and outcome of sporadic acute viral hepatitis in children? J Gastroenterol Hepatol. 2006;21:1533-7. http://dx.doi.org/10.1111/j.1440-1746.2006.04509.x
27. Zaki Mel S, Salama OS, Mansour FA, Hossein S. Hepatitis E virus coinfection with hepatotropic viruses in Egyptian children. J Microbiol Immunol Infect. 2008;41:254-8.
28. Pisanti FA, Coppola A, Galli C. Association between hepatitis C and hepatitis E viruses in southern Italy. Lancet. 1994;344:746-7.
29. Arankalle VA, Chobe LP. Retrospective analysis of blood transfusion recipients: Evidence for post-transfusion hepatitis E. Vox Sang. 2000;79:72-4. http://dx.doi.org/10.1159/000031215
30. Boxall E, Herborn A, Kochethu G, Pratt G, Adams D, Ijaz S, et al . Transfusion-transmitted hepatitis E in a ´nonhyperendemic´ country. Transfus Med. 2006;16:79-83. http://dx.doi.org/10.1111/j.1365-3148.2006.00652.x
31. Colson P, Coze C, Gallian P, Henry M, De Micco P, Tamalet C. Transfusion-associated hepatitis E, France. Emerg Infect Dis. 2007;13:648-9. http://dx.doi.org/10.3201/eid1304.061387
32. Haim-Boukobza S, Ferey MP, Vetillard AL, Jeblaoui A, Pelissier E, Pelletier G, et al . Transfusion-transmitted hepatitis E in a misleading context of autoimmunity and drug-induced toxicity. J Hepatol. 2012;57:1374-8. http://dx.doi.org/10.1016/j.jhep.2012.08.001.
33. Matsubayashi K, Nagaoka Y, Sakata H, Sato S, Fukai K, Kato T, et al . Transfusion-transmitted hepatitis E caused by apparently indigenous hepatitis E virus strain in Hokkaido, Japan. Transfusion. 2004;44:934-40. http://dx.doi.org/10.1111/j.1537-2995.2004.03300.x
34. Matsubayashi K, Kang JH, Sakata H, Takahashi K, Shindo M, Kato M, et al . A case of transfusion-transmitted hepatitis E caused by blood from a donor infected with hepatitis E virus via zoonotic food-borne route. Transfusion. 2008;48:1368-75. http://dx.doi.org/10.1111/j.1537-2995.2008.01722.x
35. Tolosa N. Hepatitis B, C y coinfección hepatitis B-Delta. Fecha de consulta: 16 de septiembre de 2015. Disponible en: http://www.ins.gov.co/lineas-de-accion/subdireccion-vigilancia/sivigila/protocolos%20sivigila/pro%20hepatitis%20b%20c%20y%20delta.pdf
36. Ganova-Raeva L, Punkova L, Campo DS, Dimitrova Z, Skums P, Vu NH, et al . Cryptic hepatitis B and E in patients with acute hepatitis of unknown etiology. J Infect Dis. 2015;212:1962-9. http://dx.doi.org/10.1093/infdis/jiv315
37. Kamar N, Dalton HR, Abravanel F, Izopet J. Hepatitis E virus infection. Clin Microbiol Rev. 2014;27:116-38. http://dx.doi.org/10.1128/CMR.00057-13
38. Rodríguez-Frías F, Jardi R, Buti M. Hepatitis E: virología molecular, epidemiología y patogénesis. Enferm Infecc Microbiol Clin. 2015;30:624-34. http://dx.doi.org/10.1016/j.eimc.2012.01.014
39. Pillonel J, Gallian P, Sommen C, Couturier E, Piquet Y, Djoudi R, et al . Assessment of a transfusion emergent risk: The case of HEV. Transfus Clin Biol. 2014;21:162-6. http://dx.doi.org/10.1016/j.tracli.2014.07.004
40. Gallian P, Piquet Y, Assal A, Djoudi R, Chiaroni J, Izopet J, et al . Hepatitis E virus: Blood transfusion implications. Transfus Clin Biol. 2014;21:173-7. http://dx.doi.org/10. 1016/j.tracli.2014.07.007
41. Guzmán B, Nava G, Mejía A, Soler J. Estado de la vigilancia de la calidad del agua para consumo humano en Colombia - 2013. Fecha de consulta: 16 de septiembre de 2015. Disponible en: http://www.ins.gov.co/sivicap/Normatividad/Estado%20de%20la%20vigilancia%20de%20la%20calidad%20del%20agua%202013.pdf
42. Contreras M, González K. El acceso al agua para consumo humano en Colombia. Fecha de consulta: 16 de septiembre de 2015. Disponible en: http://www.economiainstitucional.com/esp/vinculos/pdf/No29/myanez29.pdf
43. Folleco A. Informe final hepatitis A, Colombia, 2013. Fecha de consulta: 16 de septiembre de 2015. Disponible en: http://www.ins.gov.co:81/lineas-de-accion/Subdireccion-Vigilancia/Informe%20de%20Evento%20Epidemiolgico/HEPATITIS%20A%202013.pdf