Biomédica 2016;36:593-602
doi: http://dx.doi.org/10.7705/biomedica.v36i4.3135
ARTÍCULO ORIGINAL
1 Departamento de Genética, Faculdade de Medicina de Ribeirão Preto, Universidad de São Paulo, Ribeirão Preto, São Paulo, Brasil
2 Departamento de Biología, Faculdade de Filosofía, Ciencias y Letras de Ribeirão Preto, Universidade de São Paulo, Ribeirão Preto, São Paulo, Brasil
Contribución de los autores:Andrés Felipe Aristizábal-Pachón: recolección de muestras, datos clínicos y de histopatología, ejecución de experimentos y análisis de datos
Los dos autores participaron en la concepción del estudio, el diseño de los experimentos y en la escritura del artículo.
Recibido: 23/11/15; aceptado: 03/05/16
Introducción. La cadherina E (CDH1) cumple un papel importante en la transición epitelio-mesénquima y está relacionada con la invasión y las metástasis en varios tipos de carcinomas. Sin embargo, el efecto de las mutaciones y ‘epimutaciones’ germinales en la propensión al cáncer de mama no es claro.
Objetivo. Evaluar el polimorfismo rs5030625, los cambios en el patrón de metilación del promotor y la expresión en la transcripción del gen CDH1 en pacientes con cáncer de mama.
Materiales y métodos. Se tomaron muestras de sangre periférica de 102 pacientes con cáncer de mama y 102 mujeres de control. La genotipificación del polimorfismo rs5030625 se hizo mediante reacción en cadena de la polimerasa (PCR) y análisis de polimorfismos de longitud del fragmento de restricción; la PCR y el análisis de disociación de alta resolución sensible a metilación se emplearon para determinar el estado y el nivel de metilación del promotor del CDH1; por último, el nivel de expresión en la transcripción del CDH1 se evaluó mediante PCR cuantitativa con transcripción inversa.
Resultados. Los resultados no evidenciaron asociación entre el polimorfismo rs5030625 y el cáncer de mama. Se encontraron perfiles aberrantes de metilación del promotor del CDH1 en las pacientes con cáncer de mama relacionados con las primeras etapas de desarrollo del cáncer. La disminución de la expresión del CDH1 se asoció con la presencia de metástasis y el estado de metilación del promotor.
Conclusión. Las alteraciones en el CDH1 se asociaron con la invasión y las metástasis en el cáncer de mama. Se proporcionó evidencia adicional sobre la relevancia del CDH1 en el desarrollo y la progresión del cáncer de mama.
Palabras clave: neoplasias de la mama/genética; cadherinas; reacción en cadena de la polimerasa; polimorfismo de longitud del fragmento de restricción; reacción en cadena de la polimerasa de transcriptasa inversa.
doi: http://dx.doi.org/10.7705/biomedica.v36i4.3135
Effect of genetics, epigenetics and variations in the transcriptional expression of cadherin–E in breast cancer susceptibility
Introduction: Cadherin-E (CDH1) is an important regulator of epithelial-mesenchymal transition, invasion and metastasis in many carcinomas. However, germinal epimutations and mutations effect in breast cancer susceptibility is not clear.
Objective: To evaluate rs334558 polymorphism, promoter methylation status and CDH1 expression profile in breast cancer patients.
Materials and methods: We collected peripheral blood samples from 102 breast cancer patients and 102 healthy subjects. The identification of rs334558 polymorphism was performed using PCR-RFLP, while methylation-specific PCR (MSP) and methylation-sensitive high-resolution melting (MS-HRM) were used to explore CDH1 methylation status; finally, CDH1 transcriptional expression profile was evaluated using RT-qPCR.
Results: We found no association between rs334558 polymorphism and breast cancer. Aberrant promoter methylation profile was found in breast cancer patients and it was related with early cancer stages. CDH1 down-regulation was significantly associated with metastasis and promoter methylation.
Conclusion: CDH1 alterations were associated with invasion and metastasis in breast cancer. Our results offer further evidence of CDH1 relevance in breast cancer development and progression.
Key words: Breast neoplasms/genetics; cadherins; polymerase chain reaction; restriction fragment length polymorphism; reverse transcriptase polymerase chain reaction.
doi: http://dx.doi.org/10.7705/biomedica.v36i4.3135
La cadherina E es una glucoproteína de transmembrana codificada por el gen de la cadherina (CDH1) localizado en el cromosoma 16q22.1; es la proteína de adhesión más importante del epitelio y hace parte del tipo I de las cadherinas “clásicas”. Es responsable por las uniones celulares dependientes de Ca2+, de la vía de adhesión celular homofílica, del desarrollo de la polaridad normal del epitelio y de la arquitectura del tejido (1,2). Además, une el entorno extracelular con el citoesqueleto y, mediante su dominio intracelular, hace parte de las vías de señalización celular con las cateninas, particularmente la p120, la alfa y la beta (3,4).
La cadherina E contiene un solo dominio transmembrana, cinco dominios extracelulares, de los cuales el más próximo a la membrana se caracteriza por presentar cuatro cisteínas conservadas que estabilizan la formación de un fuerte contacto celular mediante enlaces disulfuro, y un dominio citoplásmico que puede dividirse en dos subdominios: uno conservado proximal, generalmente denominado dominio yuxtamembrana, y el otro, el dominio de unión a beta catenina (5). El dominio citoplásmico se une a las cateninas (6). La catenina alfa es una proteína homóloga a la vinculina que se une a la actina. Las interacciones de la cadherina E y la catenina alfa forman la región adherente; estas interacciones son mediadas por la catenina beta, la cual actúa como un adaptador entre ellas (7).
La pérdida de la función adhesiva de la cadherina E es un paso crítico en la progresión de las células epiteliales hacia un fenotipo más agresivo. La cadherina E se reconoce ampliamente como un gen supresor de factores de invasión, ya que su inactivación o la diminución de su función debido a mutaciones, deleciones alélicas o alteraciones epigenéticas (por ejemplo, la hipermetilación del promotor), se asocian con la transición epitelio-mesénquima, lo que conlleva una mayor agresividad del tumor y un mayor potencial metastásico. La función de la cadherina E se pierde frecuentemente durante el desarrollo del cáncer epitelial humano (8); además, se asocia con la diferenciación y la invasión del tumor, y la metástasis en ganglios linfáticos (2,9). Las alteraciones genéticas y epigenéticas, y los cambios en el nivel de transcripción de la cadherina E, se han asociado con el desarrollo del cáncer de mama (10-13), pero dichos resultados no son concluyentes y, en algunos casos han sido contradictorios.
El cáncer de mama se cuenta entre las enfermedades más comunes en las mujeres de todo el mundo; aproximadamente, 23 % de todos los casos diagnosticados de cáncer se relacionan con la mama. En 2012, su incidencia anual era de 1.677 millones de casos nuevos y 522.000 muertes (14,15). En Brasil, el cáncer de mama ha sido una de las principales causas de muerte desde 1979 (16,17), con 57.120 casos nuevos en el 2014, y un riesgo estimado de 56,1 casos por cada 100.000 mujeres; es el más común en la población femenina y representa el 20,8 % de todos los tipos de cáncer diagnosticados en las mujeres. Este cáncer es la séptima causa de muerte en Brasil, después de las enfermedades cerebrovasculares y la diabetes, supera los otros tipos de cáncer y fue la causa de muerte de 13.345 pacientes en el 2012 (17).
En este contexto, la importancia de una mejor compresión de los mecanismos moleculares involucrados en el desarrollo y la progresión del cáncer de mama es evidente, ya que contribuye a mejorar el diagnóstico y el pronóstico en términos de tiempo y seguridad.
El objetivo del presente estudio fue evaluar la asociación de las alteraciones genéticas y epigenéticas, y los cambios en la expresión en la transcripción del CDH1, con el desarrollo del cáncer de mama en un grupo de casos y un grupo de control.
Materiales y métodos
Población de estudio
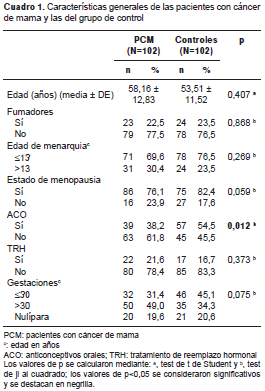
Se tomaron muestras de sangre periférica de 204 mujeres de los hospitales Santa Casa y Hospital de las Clínicas de la ciudad de Ribeirão Preto (SP), Brasil. Del total, 102 correspondieron a pacientes con cáncer de mama sin importar su edad o la etapa de la enfermedad. La información sobre el estado de los receptores hormonales, el tipo histológico, la edad en el momento del diagnóstico y el estadio según el método de estadificación TNM se obtuvieron de la historia clínica de cada paciente (cuadro 1). El grupo de control estuvo conformado por 102 mujeres sanas, sin historia familiar de cáncer. La información sobre el número de gestaciones, la edad de la menopausia y la menarquia, el tabaquismo, el uso de anticonceptivos y el tratamiento de reemplazo hormonal, se recopiló mediante un cuestionario específico (cuadro 2).


Extracción de ADN
Se extrajo el ADN genómico de cada muestra con el kit de purificación Wizard Genomic DNA (Promega, Madison, WI, USA), siguiendo las instrucciones del proveedor. Cada muestra extraída se cuantificó por espectrofotometría en el equipo NanoVue (GE, Fairfield, CT, USA). Se evaluaron la pureza con las razones 260/230 y 260/280, y la integridad mediante electroforesis en gel de agarosa al 1 %.
Genotipificación
La región promotora del gen CDH1 presenta el polimorfismo de un solo nucleótido (Single Nucleotide Polymorphism, SNP) rs5030625, el cual se evaluó mediante reacción en cadena de la polimerasa (PCR) y análisis de polimorfismos de longitud del fragmento de restricción (Restriction Fragment Length Polymorphism, RFLP), usando iniciadores que amplifican regiones específicas del promotor del CDH1.
Las secuencias de los iniciadores se presentan en el cuadro 3. La PCR se hizo en un volumen total de 20 μl que contenían 100 ng de ADN genómico, 10 pmol de cada iniciador, 4 nmol de cada dNTP (GE, Fairfield, CT, USA), 2 mM de MgCl2 y 1 U de Taq polimerasa Platinum en solución tampón 1X (Invitrogen, Carlsbad, CA, USA). Las condiciones de la amplificación fueron las siguientes: un ciclo a 95 °C durante dos minutos, seguido de 35 ciclos a 94 °C durante un minuto, a 64 °C durante un minuto y a 72 °C durante un minuto, finalizando con uno a 72 °C durante cinco minutos.

El producto amplificado fue de 447 pb y su digestión se hizo con la enzima de restricción BanII (Fermentas, Waltham, MA, USA), la cual hace cortes en dos posiciones en ausencia del polimorfismo, generando tres fragmentos de 263, 116 y 68 pb, y un corte en presencia del polimorfismo, generando dos fragmentos de 331 y 116 pb. Los productos de la PCR y del análisis de RFLP se visualizaron en gel de agarosa al 2,5 %.
Conversión con bisulfuro
El ADN extraído se sometió a conversión de las citosinas no metiladas en uracilos con el estuche Cells-to-CpG™ Bisulfite Conversion Kit (Applied Biosystems, Carlsbad, CA, USA), aplicando el siguiente protocolo. El ADN se sometió a desnaturalización y se mezcló con bisulfito de sodio, y después, a ciclos a temperaturas de 95 °C y 65 °C durante dos horas, lo cual permitió que el grupo amino (-NH2) de todas las citosinas se convirtiera en grupo cetona (=O) y, así, las primeras se convirtieran en uracilos; solo las 5’metilcitosinas no se convirtieron por interferencia electrónica del grupo metilo. Por último, la muestra de ADN se purificó en columnas de separación para retirar la sal y el bisulfito. Como control positivo de la conversión, se amplificó un fragmento de 133 pb del gen de la beta actina usando un conjunto de iniciadores específicos para ADN modificado, los cuales amplifican independientemente del estado de metilación de la muestra.PCR específica para metilación (MSP)
Para evaluar el estado de la metilación del gen CDH1, se analizaron cinco islas CpG de la región promotora en las posiciones 913, 852, 846, 838 y 836. Esta región específica se amplificó dos veces mediante PCR convencional usando iniciadores específicos para las islas CpG metiladas y las no metiladas, según descripción previa (18).Las secuencias de los iniciadores se presentan en el cuadro 3. La PCR se hizo en un volumen total de 20 μl que contenían 100 ng de ADN convertido con bisulfito, 0,5 μM de cada iniciador, 0,2 mM de la mezcla de los dNTP (GE, Fairfield, CT, USA), 2 mM de cloruro de magnesio y 1 U de Taq polimerasa Platinum en solución tampón 1X (Invitrogen, Carlsbad, CA, USA). Como control positivo de la amplificación del estado metilado y del no metilado, se hizo una reacción usando el estuche Cells-to-CpG™ Methylated & Unmethylated gDNA Control Kit (Applied Biosystems, Carlsbad, CA, USA) para cada conjunto de iniciadores, en las siguientes condiciones: 95 °C durante 10 minutos, seguido de 40 ciclos a 94 °C durante 30 segundos, 57 °C y 53 °C (metilado y no metilado, respectivamente) durante 30 segundos y 72 °C durante 30 segundos, finalizando con 72 °C durante 10 minutos.
Análisis de la disociación de alta resolución sensible a la metilación
La disociación de alta resolución sensible a la metilación (Methylation-Sensitive High Resolution Melting, MS-HRM) se hizo para semicuantificar el grado de metilación del promotor del CDH1. Se diseñaron iniciadores específicos para la región promotora del CDH1, usando el programa Methyl Primer Express™ (Applied Biosystems, Carlsbad, CA, USA), cuyas secuencias se presentan en el cuadro 3. La disociación se hizo en un volumen final de 20 μl, usando la mezcla MeltDoctor™ HRM Master Mix 1X (Applied Biosystems, Carlsbad, CA, USA), 0,5 μM de cada iniciador y 80 ng de ADN convertido con bisulfito en las siguientes condiciones: un ciclo de 10 minutos a 95 °C, 40 ciclos de 15 segundos a 95 °C y uno de un minuto a 60 °C. Por último, el análisis de disociación de alta resolución se hizo a partir de 60 °C durante un minuto, con aumento progresivo de temperatura de 0,3 % cada 15 segundos hasta alcanzar los 95 °C. Se utilizaron diluciones del estuche Cells-to-CpG™ Methylated & Unmethylated gDNA Control Kit (Applied Biosystems, Carlsbad, CA, USA) para elaborar las curvas de los patrones de metilación a 100, 10, 5, 2, 1 y 0 %.Todos los experimentos se hicieron en el equipo Applied Biosystems Step One plus Real-Time PCR System. Con la comparación de las curvas de los patrones de metilación mediante el programa HRMTM v3.0.1 (Applied Biosystems, Carlsbad, CA, USA), se determinó el porcentaje de metilación de las regiones promotoras del gen CDH1 en el grupo de estudio.
Extracción de ARN
En cada muestra de sangre se separaron las células mononucleares (Peripheral Blood Mono nuclear Cell, PBMC) con el medio Histopaque 1077 (St. Louis, MO, USA). El ARN total se extrajo usando TRIzol (Invitrogen, Carlsbad, CA, USA), de acuerdo con las recomendaciones del fabricante. Las muestras de ARN se cuantificaron en el equipo NanoVue (GE, Fairfield, CT, USA). Las razones 260 a 230 y 260 a 280 sirvieron para determinar la pureza, y la integridad de las moléculas se evaluó con la inmunoprecipitación de cromatina (ChIP) del estuche RNA 6000 Nano Kit en un bioanalizador 2100 (Agilent, Santa Clara, CA, USA).PCR cuantitativa en tiempo real
El ARN fue convertido en ADN complementario usando ADNasa I y transcriptasa inversa (SuperSript III Transcriptase Reverse, Invitrogen, Carlsbad, CA, USA). La expresión génica se evaluó cuantitativamente usando la mezcla Taqman Universal PCR Master Mix y las sondas Taqman prediseñadas del CDH1 (Hs00254446_m1), y del GAPDH (Hs02758991_g1) usado como gen constitutivo, (Applied Biosystems, Carlsbad, CA, USA), según el protocolo del proveedor.Todos los experimentos se hicieron en el equipo Applied Biosystems 7500 Real-Time PCR System y se analizaron con el programa DataAssist v3.01TM. El nivel de expresión de la transcripción (Fold change, FC) se calculó con la ecuación FC=2-ΔCt según recomendación de Schmittgen, et al. (19), comparando el grupo de casos con el de control. Cada experimento se hizo por triplicado con cada muestra.
Análisis estadístico
Los datos se analizaron con el programa estadístico SPSS®, versión 22 para Windows (Chicago, IL, USA). Las pruebas t de Student y U de Mann-Whitney se usaron para comparar los datos de expresión génica entre los grupos. La regresión logística incondicional se usó para calcular la razón de probabilidades (odds ratio, OR), con un intervalo de confianza de 95 % (IC95%), de la relación de los genotipos con el cáncer de mama.Para evaluar la relación de los genotipos y los perfiles de metilación con las características histopatológicas, se hicieron la prueba de ji al cuadrado y la exacta de Fisher, en tanto que, con el análisis de varianza (ANOVA) y la prueba de Kruskal-Wallis, se hizo el análisis multivariado. Los valores de probabilidad (p) menores de 0,05 se consideraron estadísticamente significativos.
Para el análisis de los resultados de expresión génica, el valor de p se ajustó mediante el método de Benjamini-Hochberg para establecer la tasa de falsos descubiertos.
Consideraciones éticas
Todos los procedimientos de este estudio fueron aprobados por el Comitê Nacional de ética em Pesquisa, bajo el número de aprobación CAAE: 01779812.9.0000.5440, y por el Comité de ética en Investigación del Hospital das Clínicas de la Facultad de Medicina de la Universidad de Sao Paulo. El consentimiento informado fue firmado por todos los participantes en el estudio.Resultados
Asociación del polimorfismo rs5030625 y el riesgo de cáncer de mama
La variante rs5030625 del gen CDH1 se analizó en 102 pacientes con cáncer de mama y en 102 mujeres sanas que hacían parte del grupo de control. Las frecuencias genotípicas se calcularon y se comprobó si se mantenía el equilibrio de Hardy-Weimberg (p>0,05), con lo cual se evidenció la falta de estratificación poblacional. No se encontraron diferencias estadísticamente significativas en la distribución de los genotipos G/G, G/GA y GA/GA entre el grupo de pacientes y el grupo de control (c2=0,329; gl=2; p=0,848). El análisis de asociación hecho mediante regresión logística incondicional para evaluar el posible efecto de la variante en el riesgo de desarrollar cáncer de mama, indicó que no hubo asociación significativa entre la presencia del polimorfismo y el desarrollo de la enfermedad. En cuanto a las otras variables y factores de riesgo analizados en este estudio, no se encontraron diferencias significativas en la distribución de las frecuencias genotípicas entre el grupo de casos y el de control.
Estado de la metilación de las islas CpG en las regiones promotoras del CDH1 y el desarrollo del cáncer de mama
El estado de la metilación de las islas CpG de la región promotora del gen CDH1 se evaluó mediante PCR específica para metilación (Methylation-Specific PCR, MSP) y MS-HRM. Los resultados obtenidos evidenciaron la existencia de perfiles de metilación aberrantes en el promotor del CDH1. Según el análisis por MSP del grupo de casos, en 59 (57,8 %) pacientes se encontró el promotor metilado. Debe resaltarse que no se observó metilación del promotor en los individuos del grupo de control. Después del análisis mediante MSP, las muestras se sometieron a MS-HRM para semicuantificar su nivel de metilación, y se comprobó de nuevo que en el grupo de control no hubo metilación (0 %) en la región promotora del CDH1. Con respecto al grupo de casos, el análisis de MS-HRM mostró niveles de metilación inferiores al 1 % en los individuos que habían presentado el promotor metilado en la MSP.
En cuanto a los otros factores de riesgo considerados en el estudio, como la edad de la menarquia, la edad de la menopausia, la edad y el número de gestaciones, el uso de anticonceptivos y el tratamiento de reemplazo hormonal, no hubo asociación de ninguno de ellos con el perfil aberrante de metilación (p>0,05).
Con respecto a las características clínico-patológicas del tumor, el estado de metilación del promotor del CDH1 presentó diferencias estadísticamente significativas en cuanto a la edad en el momento del diagnóstico (c2=12,57; gl=1; p<0,001). Se encontraron resultados similares en relación con el tipo histológico, el tamaño del tumor y la presencia de metástasis (c2=14,185; gl=1; p<0,001; c2=13,123; gl=1; p<0,001, y c2=7,954; gl=1; p=0,005, respectivamente). En el cuadro 4 se resumen los resultados obtenidos y su distribución según las características clínico-patológicas del tumor. Cabe resaltar la tendencia del estado de la metilación con respecto a la progresión del cáncer. El porcentaje de pacientes que no presentaban metilación del promotor del CDH1 fue mayor en aquellos individuos con etapas más avanzadas de la enfermedad (tumor de mayor tamaño y grado, y metástasis).

Los resultados del estado de la metilación se analizaron en relación con la variante rs5030625 y no se encontraron diferencias en la distribución de los genotipos en cuanto al estado de la metilación del promotor del CDH1 (c2=0,930; gl=2; p=0,628). De igual manera, se encontró metilación del promotor en 42 de las 71 (59,2 %) pacientes homocigotas para el alelo silvestre, en 14 de las 27 (51,9 %) pacientes heterocigotas y en dos de las cuatro (50 %) pacientes homocigotas para la variante.
Perfil de expresión génica del CDH1 y su relación con el riesgo de cáncer de mama
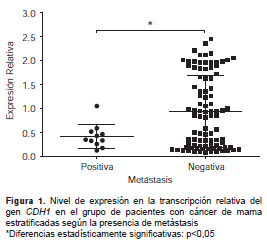
La expresión génica del CDH1 se evaluó en todos los individuos que participaron en el estudio. Los resultados evidenciaron que en el grupo de pacientes con cáncer de mama había una leve disminución de la expresión génica del CDH1, en comparación con el grupo de control; sin embargo, esta no fue estadísticamente significativa (log2 fold change=– 0,168 ± 0,13; p=0,506). En cuanto a las características histopatológicas del tumor, la presencia de metástasis distales se correlacionó con alteraciones en la expresión génica del CDH1 (figura 1). Los resultados mostraron una disminución estadísticamente significativa en las pacientes que presentaban metástasis distales, en comparación con aquellas que no la presentaban (p=0,002). Otras características tumorales y el estado de los receptores hormonales de estrógeno, de progesterona y del receptor 2 del factor de crecimiento epidérmico humano (Human Epidermal Growth Factor Receptor, HER2), no mostraron influencias significativas en la distribución de los resultados.
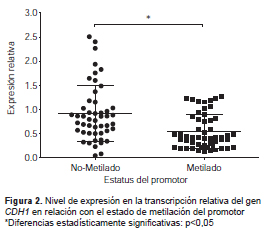
Con respecto a la variante rs5030625, los análisis no mostraron asociación entre los genotipos y los cambios en la expresión génica del CDH1, ni en el grupo de casos ni en el de control. Por último, los análisis del estado de la metilación del promotor del CDH1 revelaron una disminución significativa en la expresión génica del CDH1 en cuanto al estado de la metilación del promotor (p=0,004), como se muestra en la figura 2.
Discusión
La cadherina E tiene un papel importante en la regulación de las concentraciones citoplasmáticas de la beta catenina. En este estudio se analizó la variante rs5030625 localizada en la región promotora del gen CDH1. Este polimorfismo es una inserción del nucleótido adenina (A) en la posición 347 de la secuencia promotora. Los SNP en la región promotora pueden modular la eficiencia de la transcripción del gen y su función; Shin, et al., en líneas celulares específicas, demostraron que el polimorfismo rs5030625 presenta un efecto significativo en la actividad en la transcripción, ya que en la proximidad del promotor se encuentran dos cajas E, una caja CAAT y una caja rica en G-C con sitio de unión en el factor de transcripción SP1, el cual interactúa con varios elementos reguladores que controlan la expresión específica del epitelio del CDH1 (20).Hasta donde se pudo constatar, esta es la primera vez que la variante rs5030625 se estudia en población brasilera. Estos resultados no mostraron asociación significativa entre la presencia del polimorfismo y el riesgo de desarrollar cáncer de mama; tampoco se encontró asociación con los cambios en el nivel de expresión de la transcripción del gen. Tipirisetti, et al., evaluaron este mismo polimorfismo en las células mononucleares de sangre periférica de pacientes con cáncer de mama en población de la India y no encontraron asociación de esta variante con el riesgo de desarrollar cáncer de mama, lo cual coincide con los resultados de este estudio (10). En algunos estudios se ha analizado la relación de otro polimorfismo en esta región del CDH1, la variante –160A, con el riesgo de desarrollar cáncer de mama y no se ha encontrado asociación (21,22), mientras que en algunos otros estudios, se encontró un aumento significativo del riesgo de desarrollar este cáncer (10,23). Los resultados concernientes al efecto de las variables en la región promotora del CDH1 y su asociación con el cáncer de mama no fueron suficientemente claros, por lo que es necesario determinar el efecto real de estas mutaciones en el funcionamiento de la proteína. Contrario a lo observado en este estudio, en varias investigaciones se ha asociado el polimorfismo rs5030625 con la endometriosis (24,25) y el cáncer colorrectal en población china (2), con una mayor agresividad del adenocarcinoma esofágico en población holandesa (26) y, del cáncer gástrico familiar, en Corea del Sur (20,27), resultados que indican la importancia de estudiar estos polimorfismos en diferentes poblaciones, pues la variabilidad genética puede influir en su efecto en la propensión individual a desarrollar cáncer.
El gen CDH1 frecuentemente se encuentra en estado de metilación en el tejido tumoral en el cáncer de la mama, mas no en el tejido mamario normal (13). Los resultados que se presentan revelaron un estado aberrante de metilación en el promotor del gen CDH1, y en el análisis mediante MS-HRM se determinó un porcentaje de metilación bajo (inferior a 1 %), pero diferente en casos y controles, lo cual evidenció que los niveles de metilación de los promotores específicos en las PBMC de mujeres con cáncer de mama eran moderadamente mayores que los de las mujeres sanas, y que podrían servir de base para el desarrollo de marcadores específicos de diagnóstico de esta enfermedad. En este mismo sentido, Sebova, et al., encontraron el promotor del CDH1 en estado de metilación en la sangre periférica de 20 de 92 pacientes con cáncer de mama.
Al igual que en este estudio, el promotor metilado del CDH1 no se encontró en el grupo de control, y no se asoció con ninguna característica histopatológica del tumor (28). Contrario a lo aquí reportado, en los estudios de Sturgeon, et al., y de Zmetakova, et al., no se asoció la metilación del promotor del CDH1 con el cáncer de mama (29,30). En cuanto a las características histopatológicas del tumor, se observó una asociación entre el estado de la metilación del promotor y los estados iniciales del tumor, caracterizados por ausencia de metástasis y un menor tamaño del tumor, lo cual evidenciaría la necesidad de la célula maligna de inactivar la cadherina E en las primeras fases del proceso de carcinogénesis. El gen CDH1 es un importante supresor tumoral asociado previamente con muchos tipos de cáncer. En el caso del cáncer de mama, los resultados que se presentan ofrecen evidencia adicional de que en el proceso de carcinogénesis ocurren epimutaciones en genes claves del control celular, como el CDH1, que pueden alterar la expresión en la transcripción de sus productos génicos y estimular la transformación y aparición del fenotipo maligno.
Existen varios estudios que señalan el posible papel de la metilación constitutiva en el ADN extraído de sangre periférica como factor de predisposición al cáncer (31-33). Wong, et al., encontraron que la metilación del promotor del gen BRCA1 en sangre periférica era más frecuente en pacientes que habían desarrollado el cáncer de mama precozmente y, también, correlacionaron estos resultados con altos niveles de metilación del BRCA1 en muestras de biopsia de tumores (32).
En la presente investigación, los resultados evidenciaron un perfil diferencial de metilación del CDH1 en los grupos de estudio y se pudieron separar con mucha especificidad los casos de los controles; los datos sugieren la existencia de diferencias reales, aunque pequeñas, por lo cual se precisa prestar mucha atención y validarlos para evitar posibles falsos positivos. Tomados en conjunto, estos resultados sugieren que la metilación en sangre puede reflejar una predisposición al cáncer. Sin embargo, dichos cambios fueron relativamente pequeños y, además, se registró una gran superposición entre casos y controles, por lo cual la potencial utilidad de los perfiles de metilación del ADN en sangre periférica para la detección o como marcador de diagnóstico para cáncer puede ser limitada.
La expresión en la transcripción del CDH1 mostró una disminución no significativa en los pacientes con cáncer de mama. En este mismo sentido, en muchos estudios se ha encontrado una disminución significativa de la expresión génica del CDH1 en este tipo de cáncer, independientemente del tipo histológico o molecular, lo cual sugiere la importancia de este gen en la progresión del cáncer de mama (11,12). Con relación a las características histopatológicas del tumor, se encontró una disminución de la expresión en la transcripción del CDH1 en presencia de metástasis. En este estudio, se registró una clara diminución de los niveles de expresión en la transcripción del CDH1 en las pacientes con cáncer metastásico, lo cual concuerda con lo encontrado en estudios anteriores, e indica la importancia de la cadherina E en la adhesión celular y en el mantenimiento normal del epitelio que relacionan estrechamente esta molécula con el proceso de metástasis. Asimismo, los altos niveles de expresión del CDH1 se relacionaron con tumores en las etapas iniciales que mantenían su adherencia celular y tenían bajo poder metastásico, mientras que una menor expresión se relacionó con mayores grados del tumor, con la pérdida de la adhesión celular y con un acentuado comportamiento invasivo (2,11,34). Los niveles de expresión en la transcripción del CDH1 no se relacionaron con el estado de la metilación del promotor, posiblemente debido a que otros mecanismos, como las mutaciones génicas, la pérdida de heterocigosidad, los cambios en la estructura de la cromatina y las alteraciones específicas de la transcripción, regulan la expresión del CDH1 (11).
Puede concluirse que la cadherina E juega un papel muy importante en el proceso de carcinogénesis, especialmente relevante en la metástasis tumoral. El polimorfismo rs5030625 en la región promotora del CDH1 no mostró asociación con el desarrollo del cáncer de mama; sin embargo, los cambios en los perfiles de metilación y de expresión en la transcripción de este gen se relacionaron claramente con características histopatológicas del tumor, como la presencia de metástasis, y podrían servir de base para el desarrollo de marcadores específicos de diagnóstico de esta enfermedad. Los resultados que aquí se presentan evidencian el papel relevante de la cadherina-E en el desarrollo y la progresión del cáncer de mama.
Esta investigación fue financiada en parte por la Fundação de Amparo à pesquisa do Estado de São Paulo – FAPESP (Proceso no. 2013/00824), la Coordenação de Aperfeiçoamento de Pessoal de Nível Superior – CAPES y el Programa de Estudante Convénio – Pós-Graduação – PEC-PG (Proceso no. 5516-11-0).
Correspondencia:
Andrés Felipe Aristizábal-Pachón, Departamento de Genética, Faculdade de Medicina de Ribeirão Preto, Universidad de São Paulo, Avenida Bandeirantes, 3900, 14049-900 Ribeirão Preto, São Paulo, Brasil Teléfono: (5516) 2101-9368; fax: (5516) 3315-3761 sagipe07@gmail.com y sagipe07@gmail.com
1. Takeichi M. Cadherin cell adhesion receptors as a morpho-genetic regulator. Science. 1991;251:1451-5. http://dx.doi.org/10.1126/science.2006419
2. Zou XP, Dai WJ, Cao J. CDH1 promoter polymorphism (-347G->GA) is a possible prognostic factor in sporadic colorectal cancer. World J Gastroenterol. 2009;15:5340-5. http://dx.doi.org/10.3748/wjg.15.5340
3. van Roy F, Berx G. The cell-cell adhesion molecule E-cadherin. Cell Mol Life Sci. 2008;65:3756-88. http://dx.doi.org/10.1007/s00018-008-8281-1
4. Aristizábal-Pachón AF. Complexo de Degradação de b-Catenina e o Câncer de mama. Saarbrücken: Novas Edições Acadêmicas; 2015. p. 25-9.
5. Rimm DL, Morrow JS. Molecular cloning of human E-cadherin suggests a novel subdivision of the cadherin superfamily. Biochem Biophys Res Commun. 1994;200: 1754-61. http://dx.doi.org/S0006-291X(84)71656-1
6. Gumbiner BM, McCrea PD. Catenins as mediators of the cytoplasmic functions of cadherins. J Cell Sci Suppl. 1993;17:155-8.
7. Cavallaro U, Christofori G. Cell adhesion and signalling by cadherins and Ig-CAMs in cancer. Nat Rev Cancer. 2004;4:118-32. http://dx.doi.org/10.1038/nrc1276
8. Chien MH, Chou LSS, Chung TT, Lin CH, Chou MY, Weng MS, et al. Effects of E-cadherin (CDH1) gene promoter polymorphisms on the risk and clinicopathologic development of oral cancer. Head Neck. 2012;34:405-11. http://dx.doi.org/10.1002/hed.21746
9. Handschuh G, Candidus S, Luber B, Reich U, Schott C, Oswald S, et al. Tumour-associated E-cadherin mutations alter cellular morphology, decrease cellular adhesion and increase cellular motility. Oncogene. 1999;18:4301-12. http://dx.doi.org/10.1038/sj.onc.1202790
10. Tipirisetti NR, Govatati S, Govatati S, Kandukuri LR, Cingeetham A, Singh L, et al. Association of E-cadherin single-nucleotide polymorphisms with the increased risk of breast cancer: A study in South Indian women. Genet Test Mol Biomarkers. 2013;17:494-500. http://dx.doi.org/10.1089/gtmb.2012.0485
11. Asiaf A, Ahmad ST, Aziz SA, Malik AA, Rasool Z, Masood A, et al. Loss of expression and aberrant methylation of the CDH1 (E-cadherin) gene in breast cancer patients from Kashmir. Asian Pac J Cancer Prev. 2014;15:6397-403.
12. Tudoran O, Virtic O, Balacescu L, Pop L, Dragla F, Eniu A, et al. Differential peripheral blood gene expression profile based on HER2 expression on primary tumors of breast cancer patients. PLoS One. 2014;9:e102764. http://dx.doi.org/10.1371/journal.pone.0102764
13. Campan M, Weisenberger DJ, Laird PW. DNA methylation profiles of female steroid hormone-driven human malig-nancies. Curr Top Microbiol Immunol. 2006;310:141-78.
14. Aristizábal-Pachón AF, Carvalho TI, Carrara HH, Andrade J, Takahashi CS. AXIN2 polymorphisms, the beta-catenin destruction complex expression profile and breast cancer susceptibility. Asian Pac J Cancer Prev. 2015;16:7277-84. http://dx.doi.org/10.7314/APJCP.2015.16.16.7277
15. Aristizábal-Pachón AF, de Carvalho TI, Carrara HH, de Andrade JM, Takahashi CS. Detection of human mammaglobin A mRNA in peripheral blood of breast cancer patients before treatment and association with metastasis. J Egypt Natl Canc Inst. 2015;27:217-22. http://dx.doi.org/10. 1016/j.jnci.2015.09.003
16. Brito C, Portela MC, Vasconcellos MT. Survival of breast cancer women in the state of Rio de Janeiro, Southeastern Brazil. Rev Saúde Pública. 2009;43:481-9. http://dx.doi.org/S0034-89102009000300012
17. Instituto Nacional de Câncer. Estimativa 2014 – Incidência de Câncer no Brasil. Fecha de consulta: 1 de mayo de 2015. Disponible en: http://www.inca.gov.br/rbc/n_60/v01/pdf/11-resenha-estimativa-2014-incidencia-de-cancer-no-brasil.pdf
18. Tahara T, Shibata T, Nakamura M, Okubo M, Yamashita H, Yoshioka D, et al. Association between polymorphisms in the XRCC1 and GST genes, and CpG island methylation status in colonic mucosa in ulcerative colitis. Virchows Arch. 2011;458:205-11. http://dx.doi.org/10.1007/s00428-010-1038-x
19. Schmittgen TD, Livak KJ. Analyzing real-time PCR data by the comparative C(T) method. Nat Protoc. 2008;3:1101-8.
20. Shin Y, Kim IJ, Kang HC, Park JH, Park HR, Park HW, et al. The E-cadherin -347G-->GA promoter polymorphism and its effect on transcriptional regulation. Carcinogenesis. 2004;25:895-9. http://dx.doi.org/10.1093/carcin/bgh073
21. Beeghly-Fadiel A, Lu W, Gao YT, Long J, Deming SL, Cai Q, et al. E-cadherin polymorphisms and breast cancer susceptibility: A report from the Shanghai Breast Cancer Study. Breast Cancer Res Treat. 2010;121:445-52. http://dx.doi.org/10.1007/s10549-009-0579-7
22. Lei H, Sjoberg-Margolin S, Salahshor S, Werelius B, Jandakova E, Hemminki K, et al. CDH1 mutations are present in both ductal and lobular breast cancer, but promoter allelic variants show no detectable breast cancer risk. Int J Cancer. 2002;98:199-204. http://dx.doi.org/10.1002/ijc.10176
23. Yu JC, Hsu HM, Chen ST, Hsu GC, Huang CS, Hou MF, et al. Breast cancer risk associated with genotypic polymorphism of the genes involved in the estrogen-receptor-signaling pathway: A multigenic study on cancer susceptibility. J Biomed Sci. 2006;13:419-32. http://dx.doi.org/10.1007/s11373-006-9069-7
24. Shan K, Xiao-Wei M, Na W, Xiu-Feng Z, Deng-Gui W, Wei G, et al. Association of three single nucleotide polymorphisms of the E-cadherin gene with endometriosis in a Chinese population. Reproduction. 2007;134:373-8. http://dx.doi.org/134/2/373
25. Tempfer CB, Simoni M, Destenaves B, Fauser BC. Functional genetic polymorphisms and female reproductive disorders: Part II--endometriosis. Hum Reprod Update. 2009;15:97-118. http://dx.doi.org/10.1093/humupd/dmn040
26. Boonstra JJ, Marion R, Tilanus HW, Dinjens WN. Functional polymorphisms associated with disease-free survival in resected carcinoma of the esophagus. J Gastro-intest Surg. 2010;15:48-56. http://dx.doi.org/10.1007/s11605-010-1358-9
27. Yamada H, Shinmura K, Goto M, Iwaizumi M, Konno H, Kataoka H, et al. Absence of germline mono-allelic promoter hypermethylation of the CDH1 gene in gastric cancer patients. Mol Cancer. 2009;8:63. http://dx.doi.org/10. 1186/1476-4598-8-63
28. Sebova K, Zmetakova I, Bella V, Kajo K, Stankovicova I, Kajabova V, et al. RASSF1A and CDH1 hypermethylation as potential epimarkers in breast cancer. Cancer Biomark. 2011;10:13-26. http://dx.doi.org/10.3233/CBM-2012-0230
29. Sturgeon SR, Balasubramanian R, Schairer C, Muss HB, Ziegler RG, Arcaro KF. Detection of promoter methylation of tumor suppressor genes in serum DNA of breast cancer cases and benign breast disease controls. Epigenetics. 2012;7:1258-67. http://dx.doi.org/10.4161/epi.22220
30. Zmetakova I, Danihel L, Smolkova B, Mego M, Kajabova V, Krivulcik T, et al. Evaluation of protein expression and DNA methylation profiles detected by pyrosequencing in invasive breast cancer. Neoplasma. 2013;60:635-46. http://dx.doi.org/10.4149/Neo_2013_082
31. Marsit CJ, Koestler DC, Christensen BC, Karagas MR, Houseman EA, Kelsey KT. DNA methylation array analysis identifies profiles of blood-derived DNA methylation associated with bladder cancer. J Clin Oncol. 2011;29:1133-9. http://dx.doi.org/10.1200/JCO.2010.31.3577
32. Wong EM, Southey MC, Fox SB, Brown MA, Dowty JG, Jenkins MA, et al. Constitutional methylation of the BRCA1 promoter is specifically associated with BRCA1 mutation-associated pathology in early-onset breast cancer. Cancer Prev Res (Phila). 2011;4:23-33. http://dx.doi.org/10. 1158/1940-6207.CAPR-10-0212
33. Brennan K, García-Closas M, Orr N, Fletcher O, Jones M, Ashworth A, et al. Intragenic ATM methylation in peripheral blood DNA as a biomarker of breast cancer risk. Cancer Res. 2012;72:2304-13. http://dx.doi.org/10.1158/0008-5472.CAN-11-3157
34. Yuan YL, Wang YM, Liu H, Qin GF, Tang AG, Duan Y. Aberrant expression of E-cadherin in lung tissues of patients with probable lung cancer. Asian Pac J Cancer Prev. 2012;13:5149-53.