
Biomédica 2017;37:425-30
doi: https://doi.org/10.7705/biomedica.v34i2.3322
COMUNICACIÓN BREVE
1 Facultad de Ciencias Médicas “Dr. Enrique Ortega Moreira”, Universidad Espíritu Santo, Guayaquil, Ecuador
2 Unidad de Infecciones de Transmisión Sexual, Instituto Nacional de Investigación en Salud Pública, Guayaquil, Ecuador
3 Facultad de Ingeniería Marítima, Ciencias Biológicas, Oceánicas y Recursos Naturales, Escuela Superior Politécnica del Litoral, Guayaquil, Ecuador
4 Laboratorio de Micología, Centro de Investigación, Diagnóstico y Referencia, Instituto de Medicina Tropical “Pedro Kourí”, La Habana, Cuba
Contribución de los autores:
Sunny Sánchez y Dolores Zambrano: recolección de datos y muestras, caracterización convencional, escritura del manuscrito
Maylen García y César Bedoya: caracterización molecular
Carlos Fernández y María Teresa Illnait-Zaragozí: diseño del estudio y revisión crítica del manuscrito
Recibido: 18/05/16; aceptado: 14/10/16
Introducción: La neurocriptococosis es una infección fúngica oportunista que representa un alto costo en vidas humanas y para la economía de los países. Sus agentes causales, las especies del complejo Cryptococcus neoformans/Cryptococcus gattii, tienen una fase sexuada y otra asexuada, cuatro serotipos principales y siete variedades moleculares con diferencias clínico-epidemiológicas, fenotípicas y de sensibilidad a los antifúngicos.
Objetivo: Caracterizar molecularmente los aislamientos clínicos de C. neoformans de Guayaquil, Ecuador.
Materiales y métodos: Se determinó el tipo de apareamiento, el serotipo y la variedad molecular mediante reacción en cadena de la polimerasa y análisis del polimorfismo de los fragmentos de restricción de 27 aislamientos levaduriformes previamente identificados como C. neoformans mediante métodos convencionales. Los aislamientos fueron recuperados del líquido cefalorraquídeo de pacientes con síndrome neurológico seropositivos para HIV, internados en el Hospital de Infectología “Dr. José Daniel Rodríguez Maridueña”, entre diciembre de 2013 y enero de 2015.
Resultados: Se demostró el amplio predominio de C. neoformans del serotipo A, MATα y el genotipo VNI entre los aislamientos estudiados.
Conclusiones:. Estos datos son similares a los obtenidos en otros países y son los primeros de su tipo en Guayaquil, Ecuador, por lo cual constituyen un aporte importante al conocimiento de la criptococosis en esta ciudad.
Palabras clave:
Cryptococcus neoformans, criptococosis, HIV, síndrome de inmunodeficiencia adquirida, genotipo, Ecuador.doi: https://doi.org/10.7705/biomedica.v34i2.3322
Molecular characterization of Cryptococcus neoformans isolates from HIV patients, Guayaquil, Ecuador
Introduction: Neurocryptococcosis is an opportunistic fungal infection that represents a high cost in human lives and for the economy of countries. Its causative agent, the Cryptococcus neoformans/ Cryptococcus gattii species complex, has a sexual and an asexual phase, four major serotypes and seven molecular varieties with phenotypic, clinical-epidemiological and antifungal susceptibility differences.
Objective: To characterize by molecular methods clinical isolates of C. neoformans from Guayaquil, Ecuador.
Materials and methods: We determined mating types, serotypes and molecular varieties by PCR and RFLP in 27 yeast isolates previously identified as C. neoformans by conventional methods.
The isolates were recovered from cerebrospinal fluid of HIV seropositive patients with neurological syndrome admitted at “Dr. José Daniel Rodríguez Maridueña” Hospital from December, 2013, to January, 2015.
Results: We established a wide prevalence of C. neoformans serotype A, MATα and genotype VNI among the studied isolates.
Conclusions: These data are similar to those obtained in other countries and the first identified by molecular characterization in Guayaquil, Ecuador. Therefore, they constitute an important contribution to the knowledge on cryptococcosis in this country.
Key words: Cryptococcus neoformans, cryptococcosis, HIV, acquired immunodeficiency syndrome, genotype, Ecuador.
doi: https://doi.org/10.7705/biomedica.v34i2.3322
Según la Organización Mundial de la Salud (OMS) en un informe del 2014, Venezuela, Ecuador y Colombia se encontraban entre los países de la región de las Américas con las tasas de prevalencia más altas de casos de infección por el virus de la inmunodeficiencia humana (HIV) en el 2012 (360, 338 y 307 por 100.000 habitantes, respectivamente); por el contrario, Cuba presentaba las tasas más bajas (42 por 100.000 habitantes). En cuanto a la mortalidad, en el mismo reporte se informaba de una tasa de 17 por 100.000 habitantes en Ecuador, en tanto que en Cuba y Chile se presentaban las más bajas (2,6 y 2,4 por 100.000 habitantes en cada caso) (1).
En la medida en que la pandemia del síndrome de inmunodeficiencia adquirida se incrementó, se convirtió en el principal factor predisponente de varias micosis superficiales y profundas (2). A pesar de la introducción del tratamiento antirretroviral altamente efectivo, cada año se diagnostican alrededor de un millón de casos de meningitis criptococósica en pacientes con HIV. Se calcula que entre 3 y 5 % de los individuos con sida en Europa la padecen, cifra que asciende a 10 % en Estados Unidos y Brasil, en tanto que en áfrica Central supera el doble de este porcentaje y constituye la infección más común, conjuntamente con la tuberculosis y la malaria (3).
Hay poca información publicada sobre la situación de las micosis oportunistas en los pacientes con HIV-sida en Ecuador. En el 2009, Chiang, et al., publicaron un trabajo en el Hospital de Infectología de Guayaquil y demostraron que la criptococosis era la cuarta causa de ingreso por infección oportunista (4).
Las especies del complejo Cryptococcus neoformans/Cryptococcus gattii, agentes causales de la criptococosis, tienen una fase sexuada y otra asexuada, cuatro serotipos principales y ocho variedades moleculares con diferencias clínico-epidemiológicas, fenotípicas y de sensibilidad a los antifúngicos (5).
Existen numerosos métodos para estudiar la epidemiología molecular de las especies C. neoformans y C. gattii. Uno de los más ampliamente utilizados es el de los polimorfismos de longitud de los fragmentos de restricción (Restriction Fragment Length Polymorphism, RFLP). Con base en las diferencias genéticas reconocidas mediante estas pruebas, se estableció que el serotipo A comprende el genotipo VNI (el más frecuente a nivel mundial, especialmente en los países asiáticos), el VNII y el VNB (inicialmente considerado como restringido a Botsuana); el híbrido AD comprende el VNIII, así como el D y el VNIV. Los serotipos B y C incluyen los genotipos VGI (el más frecuente en las infecciones causadas por C. gattii), VGII, VGIII y VGIV (6,7).
Hasta el momento no hay datos sobre las variantes moleculares circulantes en los pacientes seropositivos para HIV en Guayaquil.
Dada la relevancia clínico-epidemiológica de dicha información, el presente trabajo tuvo como objetivo caracterizar molecularmente los aislamientos previamente recuperados de pacientes con HIV-sida e identificados como C. neoformans. Los resultados en esta investigación permitirán comprender mejor la situación de la criptococosis en Guayaquil, predecir la evolución de la enfermedad y mejorar los protocolos de tratamiento de los pacientes afectados.
Materiales y métodos
Se llevó a cabo un estudio transversal, descriptivo y prospectivo entre el 1° de diciembre de 2013 y el 31 de enero de 2015. Se incluyeron 27 aislamien- tos levaduriformes previamente identificados como C. neoformans mediante métodos convencionales (tinta china, aislamiento en agar de Sabouraud, prueba de la ureasa en agar urea de Christensen, producción de tubos germinales (filamentation) en agar harina de maíz con Tween 80®, asimilación de azúcares y crecimiento en canavanina-glicina-azul de bromotimol). Los aislamientos se recuperaron del líquido cefalorraquídeo de pacientes seropositivos para HIV internados en el Hospital de Infectología “Dr. José Daniel Rodríguez Maridueña” de Guayaquil, Ecuador (8).
Extracción de ADN
A partir del cultivo fresco de cada uno de los aislamientos, se tomaron 10 unidades formadoras de colonia y se suspendieron en 200 μl de solución tampón fosfato (PBS), pH 8,3. Para extraer el ADN, se empleó el estuche comercial High Pure PCR Template Preparation Kit® (Roche, Alemania), siguiendo las instrucciones del fabricante. Se adicionó un paso previo de lisis celular con liticasa (10 μl por muestra a 0,5 mg/ml) y se incubó a 37 °C durante 30 minutos.
Serotipo y tipo de apareamiento
Para la determinación del serotipo y del tipo de apareamiento, se realizaron dos reacciones en cadena de la polimerasa (PCR) según el proce- dimiento de Carvalho, et al. (9), en las cuales se emplearon los juegos de cebadores que se presentan en el cuadro 1.

En cada PCR se emplearon 0,2 μM de cebadores, 0,02 U/μl de Taq polimerasa, 1,5 mM de MgCl y 0,2 mM de solución trifosfato de desoxinucleótidos (dNTP). La primera PCR se sometió a 94 °C durante un minuto, seguida de 59 °C durante 30 segundos, luego 30 ciclos a 72 °C durante un minuto, finalizando con 72 °C durante 10 minutos. La segunda se sometió a 94 °C durante un minuto, a 61 °C durante 30 segundos, 30 ciclos a 72 °C durante un minuto y, por último, a 72 °C durante 10 minutos. Todos los productos amplificados se separaron en geles de agarosa al 2 % a 100 V durante una hora (9).
Variedades moleculares
Se aplicó la técnica de RFLP siguiendo el protocolo desarrollado por Meyer, et al. (10). Se utilizaron los cebadores URA5 (5′ATGTCCTCCCAAGCCCTCG ACTCCG3′) y SJ01 (5’TTAAGACCTCTGAACACCGTACTC3’). El producto de la reacción se digirió con las enzimas de restricción HhaI y Sau96I; los fragmentos obtenidos se visualizaron en gel de agarosa al 3 % después de aplicarlo a 80 V durante 3,5 horas.
Como controles positivos, se emplearon las cepas de Cryptococcus CBS9172 (Aa), CBS8710 (Aα) (ambas AFLP1/VNI), CBS10511 (Da) y CBS10513 (Dα) (ambas AFLP2/VNIV), y como negativos, la CBS6956 (Bα) AFLP6/VGII y la CBS10101 (Cα) AFLP7/VGIV, y agua.
Consideraciones éticas
El estudio se llevó a cabo previa autorización de la Decanatura de la Facultad de Ciencias Médicas de la Universidad Espíritu Santo y de la Jefatura de Docencia del Hospital de Infectología “Dr. José Daniel Rodríguez Maridueña”, Guayaquil, Ecuador.
Además, el estudio fue evaluado y aprobado por el Comité de Bioética COBI-ASFORUM (acreditado por las autoridades locales de salud del Ecuador) y el Comité de ética de la Investigación del Instituto de Medicina Tropical “Pedro Kourí”, La Habana, Cuba (dictamen No. CEI-IPK 44-14).
Resultados
Los 27 aislamientos previamente identificados como C. neoformans según sus características fenotípicas (8) correspondieron al serotipo A, tipo de apareamiento MATα. En la figura 1 se muestra la corrida electroforética de seis de los aislamientos, en la cual se evidenció la presencia de la amplificación de fragmentos de 1.200 pb que lo caracteriza.
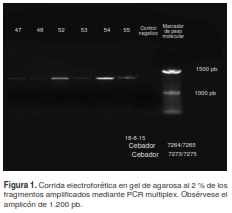
Según los resultados de la PCR-RFLP del gen URA5, 24 aislamientos correspondían al genotipo VNI, mientras que en tres de ellos no se logró la amplificación del fragmento.
Discusión
La criptococosis es una micosis que compromete seriamente la vida de los pacientes afectados y se desarrolla por la inhalación y la diseminación de conidias o levaduras disecadas de C. neoformans/ C. gattii (11). Las especies de este complejo son de vida libre y pueden sobrevivir en diversos nichos ecológicos. Cryptococcus neoformans presenta una distribución universal, afecta por lo general a individuos inmunodeficientes e incluye dos variedades fundamentales: C. neoformans var. grubii (serotipo A) y C. neoformans var. neoformans (serotipo D); además, existe el estado intervariedad (intervarietal) de C. neoformans, serotipo AD. Por su parte C. gattii (serotipos B y C) tiene una distribución geográfica más restringida a zonas tropicales y subtropicales, aunque los hallazgos recientes indican su adaptación a otros ambientes, y suele presentarse en individuos sin aparente alteración del sistema inmunitario (6,12), lo cual podría explicar el amplio predominio de C. neoformans en el presente estudio.
El Hospital de Infectología de Guayaquil atiende a 4.000 pacientes con HIV-sida de Ecuador (13).
Esta condición es ampliamente reconocida como la principal predisponente para el desarrollo de la criptococosis, especialmente la causada por C. neoformans (14). Diversos investigadores de todas las latitudes han respaldado esto, entre ellos, Meyer y Cogliati en sus respectivos metaanálisis sobre epidemiología molecular global de C. neoformans y C. gattii (10,12). Más recientemente, en Cuba, Illnait-Zaragozí, et al., también demostraron que el 100 % de los más de 100 aislamientos de Cryptococcus spp. autóctonos de esta isla incluidos en su estudio eran de C. neoformans, serotipo A, MATα, AFLP1 (correspondiente a VNI) (11), información que puede ser de gran utilidad para lograr un mejor manejo de los pacientes afectados por la criptococosis.
En la actualidad, se conoce la repercusión clínica de la infección en aislamientos del tipo MATα, los cuales presentan un comportamiento más virulento que el tipo MATa (15,16). Asimismo, se confirmó que los genotipos se distinguían entre sí por los valores de corte para la concentración inhibitoria mínima de los antifúngicos más importantes, entre otros aspectos prácticos (17,18).
Aunque no se usan en el diagnóstico de rutina, los métodos moleculares se emplean para la detección de secuencias genéticas específicas del complejo C. neoformans, tanto a partir de muestras clínicas como de cultivos (19,20). Estas herramientas permiten la identificación de las especies del complejo C. neoformans/C. gattii y otras descritas con menor frecuencia, como C. laurentii, por lo que suelen brindar resultados más completos que el diagnóstico convencional y contribuyen a una mejor comprensión de la epidemiología y la historia natural de la criptococosis.
Uno de los métodos más empleados es el RFLP, basado en el análisis del polimorfismo de un fragmento amplificado y digerido posteriormente con endonucleasas, en este caso, Sau96I y HhaI. Los estudios de epidemiología molecular demuestran que esta técnica se puede aplicar a la detección rápida de los ocho tipos moleculares principales del complejo C. neoformans (10,21). No obstante, la técnica tiene limitaciones, una de las principales, además de la compleja interpretación de los resultados, es el gran número de pasos del proceso, la cantidad requerida de reactivos y equipos, así como la demanda de ADN de alta calidad, factor crucial para su éxito (22).
La secuencia del gen URA5 es una de las más utilizadas debido a su gran sensibilidad (21). No obstante, en el presente estudio no se obtuvo ningún producto después de la amplificación de este fragmento en tres de los 27 aislamientos. Este fenómeno pudo deberse a la inhibición de la reacción de amplificación o a la cantidad insuficiente de material genético de la levadura. Hubo una concordancia del 88,8 % entre el método convencional previamente empleado para la identificación y la técnica molecular aplicada en este estudio. Este resultado avala la utilidad de estas pruebas en la identificación micológica, especialmente, cuando se emplean de forma combinada con otros métodos.
Los resultados de esta investigación son muy similares a lo ya reportado, especialmente en Asia y otros países de América Latina. En Seúl, se determinó mediante RFLP y tipificación multilocus de secuencia (Multi-Locus Sequence Typing, MLST) que el 100 % de los 46 aislamientos estudiados pertenecían al serotipo Aα (VNI) (23). Por su parte, en la India, Jain demostró que 51 de 57 aislamientos clínicos correspondían a este mismo genotipo, uno a VNIV y cinco a VGII (24).
Colombia es quizá el país de Latinoamérica que más ha estudiado la situación molecular de sus aislamientos de Cryptococcus spp. La encuesta epidemiológica conducida por Meyer, et al., brindó información valiosa sobre las variedades moleculares circulantes en nueve países de Iberoamérica (10). Sus resultados fueron corroborados posteriormente por Sánchez, et al., quienes confirmaron que 35 de los 54 aislamientos procedentes de Bogotá eran C. neoformans (93 % era Aα y 82,9 %, VNI) y 19 eran C. gattii (47,5 % correspondientes a MATay 52,6 % a VGII) (25). De igual forma, los estudios de Igreja y Delgado a partir de muestras clínicas, demostraron la prevalencia de VNI y VNII en Brasil (26-28).
Actualmente, la tipificación molecular de los microorganismos tiene un papel mucho más amplio que la simple determinación de los patrones genéticos que caracterizan un aislamiento (19). El análisis mediante estas técnicas de 27 aislamientos de C. neoformans recuperados de pacientes seropositivos para HIV en Guayaquil, demostró la prevalencia del tipo MATαA VNI. Estos resultados constituyen los primeros de su tipo en esta ciudad y representan un aporte importante al conocimiento de la criptococosis y a un mejor manejo de los pacientes afectados.
A la Universidad Espíritu Santo, al Instituto Nacional de Salud Pública e Investigación, al Hospital de Infectología “Dr. José Daniel Rodríguez” y al Laboratorio Nacional de Referencia de Micología del Instituto de Medicina Tropical “Pedro Kourí”, por su colaboración en este trabajo.
Todos los autores manifestaron no tener ningún.
No se recibió financiación para el desarrollo de este trabajo.
Correspondencia:
María Teresa Illnait-Zaragozí, Laboratorio de Micología, Instituto de Medicina Tropical “Pedro Kourí”, Avenida Novia del Mediodía, km 6 1/2, municipio La Lisa, La Habana, Cuba Fax: (537) 202 0633 y 204 6051 mtilnait@ipk.sld.cu
1. Organización Mundial de la Salud. Estadísticas sanitarias mundiales 2014. Fecha de consulta: 25 de julio de 2015. Disponible en: http://www.who.int/gho/publications/world_health_statistics/2014/es/
2. Idnurm A, Lin X. Rising to the challenge of multiple Cryptococcus species and the diseases they cause. Fungal Genet Biol. 2015;78:1-6. https://doi.org/10.1016/j.fgb.2015.05.002
3. Perfect JR, Bicanic T. Cryptococcosis diagnosis and treatment: What do we know now. Fungal Genet Biol.2015;78:49-54. https://doi.org/10.1016/j.fgb.2014.10.003
4. Chiang H, Tettamanti D, Castro G. Tinta china en orina como método de diagnóstico en criptococosis diseminada asociado a VIH/sida. Estudio transversal realizado en el hospital de infectología “José Rodríguez M.” durante el año 2009. Rev Med FCM-UCSG. 2010;16:116-23.
5. Hagen F, Khayhan K, Theelen B, Kolecka A, Polacheck I, Sionov E, et al. Recognition of seven species in the Cryptococcus gattii/Cryptococcus neoformans species complex. Fungal Genet Biol. 2015;78:16-48. https://doi.org/10.1016/j.fgb.2015.02.009
6. Trilles L, Wang B, Firacative C, Lazéra M dos S, Wanke B, Meyer W. Identification of the major molecular types of Cryptococcus neoformans and C. gattii by hyperbranched rolling circle amplification. PLoS One.2014;9:e94648. https://doi.org/10.1371/journal.pone.0094648
7. Inglis DO, Skrzypek MS, Liaw E, Moktali V, Sherlock G, Stajich JE. Literature-based gene curation and proposed genetic nomenclature for Cryptococcus. Eukaryot Cell. 2014; 13:878-83. https://doi.org/10.1128/EC.00083-14
8. Sánchez-Giler S, Zambrano-Castro D, Martínez-Machín GF, Fernández-Andreu CM, Illnait-Zaragozí MT. Neurocriptococosis en el contexto de la infección con el VIH en Guayaquil, Ecuador. Rev Cubana Med Trop. 2016;68.
9. Carvalho VG, Terceti MS, Dias ALT, Paula CR, Lyon JP, Siqueira AM de, et a. Serotype and mating type characterization of Cryptococcus neoformans by multiplex PCR. Rev Inst Med Trop Sao Paulo. 2007;49:207-10. https://doi.org/10.1590/S0036-46652007000400002
10. Meyer W, Castañeda A, Jackson S, Huynh M, Castañeda E, Arechavala A, et al. Molecular typing of IberoAmerican Cryptococcus neoformans isolates. Emerg Infect Dis. 2003;9: 189-95. https://doi.org/10.3201/eid0902.020246
11. Illnait-Zaragozí MT, Martínez-Machín GF, Fernández-Andreu CM, Perurena-Lancha MR, Hagen F, Meis JF. Cryptococcus and cryptococcosis in Cuba. A minireview. Mycoses. 2014;57:707-17. https://doi.org/10.1111/myc.12275
12. Cogliati M. Global molecular epidemiology of Cryptococcus neoformans and Cryptococcus gattii: An atlas of the molecular types. Scientifica (Cairo). 2013;2013:675213. https://doi.org/10.1155/2013/675213
13. Ministerio de Salud Pública. Ministra de Salud inaugurará área de consulta externa para pacientes con VIH en Guayaquil. Fecha de consulta: 25 de abril de 2015. Disponible en: http://instituciones.msp.gob.ec/cz8/index.php/component/content/?view=featured
14. Alspaugh JA. Virulence mechanisms and Cryptococcus neoformans pathogenesis. Fungal Genet Biol. 2015;78:55-8. https://doi.org/10.1016/j.fgb.2014.09.004
15. La Hoz RM, Pappas PG. Cryptococcal infections: Changing epidemiology and implications for therapy. Drugs. 2013;73:495-504. https://doi.org/10.1007/s40265-013-0037-z
16. Hagen F, Ceresini PC, Polacheck I, Ma H, van Nieuwerburgh F, Gabaldón T, et al. Ancient dispersal of the human fungal pathogen Cryptococcus gattii from the Amazon rainforest. PloS One. 2013;8:e71148. https://doi.org/10.1371/journal.pone.0071148
17. Espinel-Ingroff A, Chowdhary A, Cuenca-Estrella M, Fothergill A, Fuller J, Hagen F, et al. Cryptococcus neoformans-Cryptococcus gattii species complex: An international study of wild-type susceptibility endpoint distributions and epidemiological cutoff values for amphotericin b and flucytosine. Antimicrob Agents Chemother. 2012;56:3107-13. https://doi.org/10.1128/AAC.06252-11
18. Espinel-Ingroff A, Aller AI, Cantón E, Castañón-Olivares LR, Chowdhary A, Córdoba S, et al. Cryptococcus neoformans-Cryptococcus gattii species complex: An international study of wild-type susceptibility endpoint distributions and epidemiological cutoff values for fluconazole, itraconazole, posaconazole, and voriconazole. Antimicrob Agents Chemother. 2012;56:5898-906. https://doi.org/10.1128/AAC.01115-12
19. Bovers M, Hagen F, Boekhout T. Diversity of the Cryptococcus neoformans-Cryptococcus gattii species complex. Rev Iberoam Micol. 2008;25:S4-12.
20. Wengenack NL, Binnicker MJ. Fungal molecular diagnostics. Clin Chest Med. 2009;30:391-408. https://doi.org/10.1016/j.ccm.2009.02.014
21. Kidd SE, Chow Y, Mak S, Bach PJ, Chen H, Hingston AO, et al. Characterization of environmental sources of the human and animal pathogen Cryptococcus gattii in British Columbia, Canada, and the Pacific Northwest of the United States. Appl Environ Microbiol. 2007;73:1433-43. https://doi.org/10.1128/AEM.01330-06
22. Sidrim JJC, Costa AKF, Cordeiro RA, Brilhante RSN, Moura FEA, Castelo-Branco DS, et al. Molecular methods for the diagnosis and characterization of Cryptococcus: A review. Can J Microbiol. 2010;56:445-58. https://doi.org/10.1139/w10-030
23. Park SH, Kim M, Joo SI, Hwang SM. Molecular epidemiology of clinical Cryptococcus neoformans isolates in Seoul, Korea. Mycobiology. 2014;42:73-8. https://doi.org/10.5941/MYCO.2014.42.1.73
24. Jain N, Wickes BL, Keller SM, Fu J, Casadevall A, Jain P, et al. Molecular epidemiology of clinical Cryptococcus neoformans strains from India. J Clin Microbiol. 2005;43:5733-42. https://doi.org/10.1128/JCM.43.11.5733-5742.2005
25. Sánchez A, Escandón P, Castañeda E. Determinación in vitro de la actividad de los factores asociados con la virulencia de aislamientos clínicos del complejo Cryptococcus neoformans. Rev Iberoam Micol. 2008;25:145-9. https://doi.org/10.1016/S1130-1406(08)70034-2
26. Igreja RP, Santos-Lazéra MD, Wanke B, Gutiérrez-Galhardo MC, Kidd SE, Meyer W. Molecular epidemiology of Cryptococcus neoformans isolates from AIDS patients of the Brazilian city, Rio de Janeiro. Med Mycol. 2004. https://doi.org/10.1080/13693780310001644743
27. Delgado AC, Taguchi H, Mikami Y, Myiajy M, Villares MC, Moretti ML. Human cryptococcosis: Relationship of environmental and clinical strains of Cryptococcus neoformans var. neoformans from urban and rural areas. Mycopathologia. 2005;159:7-11. https://doi.org/10.1007/s11046-004-9618-4
28. Da Silva BK, Freire AK, Dos Santos Bentes A, De Lima Sampaio I, Oliveira Santos L, Silva Dos Santos M, et al. Characterization of clinical isolates of the Cryptococcus neoformans-Cryptococcus gattii species complex from the Amazonas State in Brazil. Rev Iberoam Micol. 2012;29:40-3. https://doi.org/10.1016/j.riam.2011.05.003