
Biomédica 2017;37(Supl.2):187-92
doi: https://doi.org/10.7705/biomedica.v34i2.3396
COMUNICACIÓN BREVE
Contribución de los autores:
José Gabriel Vergara: recolección de muestras, ensayos moleculares, análisis de los datos y redacción del manuscrito
Ana Milena Montesino: recolección de muestras y ensayos moleculares
Daniel Verbel-Vergara: diseño de experimentos de laboratorio y ensayos moleculares
Alveiro Pérez-Doria y Eduar Elías Bejarano: diseño de experimentos de laboratorio, análisis de los datos y corrección del manuscrito
Recibido: 22/06/16; aceptado: 10/05/17
Introducción. Las técnicas de biología molecular han permitido ampliar el conocimiento sobre las fuentes de ingestión de sangre de los insectos vectores. Sin embargo, la utilidad de estas técnicas depende de la cantidad de sangre ingerida y del proceso de digestión en el insecto.
Objetivo. Determinar el tiempo límite de detección del gen citocromo b (Cyt b) de humanos en hembras de Lutzomyia evansi alimentadas experimentalmente.
Materiales y métodos. Se evaluaron ocho grupos de hembras de L. evansi alimentadas con sangre humana, las cuales fueron sacrificadas en intervalos de 24 horas desde el momento de la ingestión sanguínea. Se extrajo el ADN total de cada hembra y se amplificó un segmento de 358 pb del gen Cyt b. Los productos amplificados fueron sometidos a un análisis de polimorfismos en la longitud de los fragmentos de restricción (Restriction Fragment Length Polymorphism, RFLP), con el fin de descartar falsos positivos.
Resultados. El segmento del gen Cyt b de humanos fue detectado en 86 % (49/57) de las hembras de L. evansi a partir de las 0 horas y hasta 168 horas después de la ingestión de sangre. En 7 % (4/57) de los individuos se amplificó el ADN del insecto y en el 7 % restante no se amplificó la banda de interés. No se encontraron diferencias estadísticas en cuanto a la amplificación del segmento del gen Cyt b de humanos ni al número de muestras amplificadas entre los grupos de hembras sacrificadas a distintas horas después de la ingestión.
Conclusión. El segmento del gen Cyt b de humanos fue detectable en hembras de L. evansi hasta 168 horas después de la ingestión de sangre.
Palabras clave: Psychodidae; citocromo b; insectos vectores; leishmaniasis; sangre.
doi: https://doi.org/10.7705/biomedica.v34i2.3396
Estimation of time detection limit for human cytochrome b in females of Lutzomyia evansi
Introduction: Molecular biology techniques have allowed a better knowledge of sources of blood meals in vector insects. However, the usefulness of these techniques depends on both the quantity of
ingested blood and the digestion process in the insect.
Objective: To identify the time limit for detection of the human cytochrome b (Cyt b) gene in experimentally fed females of Lutzomyia evansi.
Materials and methods: Eight groups of L. evansi females were fed on human blood and sacrificed at intervals of 24 hours post-ingestion. Total DNA was extracted from each female and a segment of 358 bp of Cyt b was amplified. In order to eliminate false positives, amplification products were subjected to a restriction fragment length polymorphism (RFLP) analysis.
Results: The human Cyt b gene segment was detected in 86% (49/57) of the females of L. evansi, from 0 to 168 hours after blood ingestion. In 7% (4/57) of the individuals we amplified insect DNA, while in the remaining 7%, the band of interest was not amplified. We did not find any statistical differences between groups of females sacrificed at different times post-blood meal regarding the amplification of the human Cyt b gene segment or the number of samples amplified.
Conclusion: The human Cyt b gene segment was detectable in L. evansi females up to 168 hours after blood ingestion.
Key words: Psychodidae; cytochrome b; insect vectors; blood; leishmaniasis.
doi: https://doi.org/10.7705/biomedica.v34i2.3396
La determinación de los vertebrados que sirven como reservorios silvestres de parásitos del género Leishmania spp. es importante para comprender el ciclo epidemiológico de la leishmaniasis, y suministra información para el control de la enfermedad (1).
Anteriormente, dicha determinación se hacía mediante la prueba de precipitina, un ensayo inmunológico que permitía detectar antígenos sanguíneos específicos de grupos de vertebrados a partir del contenido del tubo digestivo de flebotomíneos alimentados (2). Aunque esta técnica permitió conocer las relaciones biológicas entre algunas especies de flebotomíneos y vertebrados (3,4), la enorme diversidad biológica de los grupos taxonómicos involucrados (aves, mamíferos, reptiles y anfibios) restringía su uso por la carencia de los respectivos antisueros para cada uno de estos animales (5). Además, los procesos digestivos de los flebotomíneos provocan modificaciones en los epítopos reconocidos por los anticuerpos utilizados, lo que reduce la especificidad del ensayo serológico y el tiempo efectivo de detección del vertebrado (6).
Frente a estas limitaciones, las técnicas de biología molecular se han convertido en una herramienta de primera elección en la determinación de las fuentes de ingestión de sangre de los flebotomíneos porque amplían la gama de especies de vertebrados que se puede caracterizar.
El gen citocromo b (Cyt b) es el marcador molecular más empleado en la determinación de las fuentes de ingestión de sangre de los mosquitos (7,8) y los flebotomíneos (9,10). A pesar de ello, algunos investigadores se abstienen de usarlo dada la posibilidad de amplificar también parte del genoma del insecto en el proceso; en su lugar, prefieren emplear genes nucleares que, aunque presentan menor número de copias por célula, son exclusivos de grupos particulares de vertebrados, como sucede con el gen de la prepronociceptina (PNOC), presente únicamente en mamíferos, y cuya utilidad se ha comprobado en flebotomíneos del Viejo y el Nuevo Mundo (5,11). Sin embargo, aunque con este marcador se reduce la inespecificidad, el ADN blanco solo se puede detectar en un periodo máximo de 24 horas después de la ingestión de sangre (5,11), lo cual reduce el número de muestras aptas para determinar las fuentes de ingestión e implica la pérdida de información.
Por ello, es necesario establecer para cada especie de insecto hematófago el tiempo efectivo de detección de cada marcador molecular en la sangre ingerida, en lo cual influyen, no solo el número de copias del marcador, sino también la cantidad de sangre ingerida y las particularidades del proceso digestivo en el vector como, por ejemplo, la actividad catalítica de las enzimas que degradan el ADN (12). Se ha demostrado que la actividad de estas enzimas en los flebotomíneos es mayor entre las 24 y las 48 horas del momento de la alimentación, según la especie involucrada (13).
En este contexto, en el presente estudio se estableció el tiempo límite de detección del gen Cytb de humanos en hembras de Lutzomyia evansi, principal vector de Leishmania spp. en la región Caribe colombiana.
Materiales y métodos
Se recolectaron, aproximadamente, 100 flebotomíneos con ayuda de aspiradores bucales y una trampa de luz de tipo Shannon en el municipio de Colosó (09°31’46,975”N, 75°21’06,056”O), departamento de Sucre, Colombia.
Los insectos se mantuvieron vivos en jaulas de cría con una dieta basada en una solución estéril de sacarosa (30 %), la cual se retiró 24 horas antes de la alimentación con sangre; esta se suministró a los flebotomíneos en un alimentador artificial de polipropileno cilíndrico de 11 por 3,7 cm, cubierto en la parte inferior con piel de pollo de dos días de edad. Sobre esta membrana se depositaron 2 ml de sangre humana desfibrinada y descomplementada.
Los insectos se extrajeron del dispositivo de alimentación con un aspirador bucal y allí se examinaron con la ayuda de un estereomicroscopio Led Zeiss Stemi 2000-C, con el fin de confirmar que se hubieran alimentado. Las hembras alimentadas se mantuvieron en jaulas de cría a una temperatura media de 28,5 °C y una humedad relativa de 85 % en el laboratorio del Grupo de Investigaciones Biomédicas de la Universidad de Sucre.
Para los ensayos del tiempo efectivo de detección del gen Cyt b, se sacrificaron entre cuatro y diez hembras diariamente durante ocho días, en un periodo comprendido entre las 0 y las 168 horas de la ingestión de sangre (cuadro 1).

Cada espécimen sacrificado fue revisado bajo el estereomicroscopio, con el fin de registrar el grado de dilatación del abdomen provocado por la sangre según los estados de digestión descritos por Dolmatova, et al. (14).
Cada hembra se fragmentó para separar los últimos segmentos del abdomen, la cabeza y el ala, y emplearlos para la determinación taxonómica de la especie (15,16).
La extracción del ADN genómico total se hizo del tórax y los segmentos proximales del abdomen de cada hembra alimentada mediante el procedimiento de alta concentración de sales descrito por PérezDoria, et al. (17), al cual se le agregó un paso de digestión enzimática con proteinasa K (6,3 µg/µl) a 65 °C durante dos horas.
La reacción en cadena de la polimerasa (PCR) se realizó con los iniciadores L14841 (5’-CCATC CAACATYTCADCATGATGAAA-3’) y H15149 (5’GCHCCTCAGAATGATATTTGKCCTCA-3’), que flanquean un segmento de 358 pb del gen Cyt b en vertebrados (18-21). La amplificación se hizo con la enzima ExAct Taq DNA Polymerase™ (M. Biotech), en un volumen final de 50 µl, que incluyó 10 pmoles/µl de los iniciadores, 5 µl de solución tampón de PCR con los dNTP incorporados, 5 µl de solución tampón Q Plus y 3 µl de la solución con ADN (aproximadamente10 ng de ADN).
El perfil térmico empleado fue el ya descrito por Paternina, et al. (10). Los productos amplificados se separaron por electroforesis en gel de agarosa (2 %) y se visualizaron en un transiluminador de luz ultravioleta. Cada una de las muestras se cuantificó en un espectrofotómetro Nano Drop 2000™, Thermo Scientific, para determinar la reproducibilidad del ensayo en los individuos de cada grupo de hembras después de la ingestión. La pureza de los ácidos nucleicos se estimó en el equipo mediante la relación de absorbancia a 260 y 280 nm.
Teniendo en cuenta que en estudios previos sobre las fuentes de ingestión se ha reportado la amplificación concomitante del gen Cyt b de insectos y el de vertebrados (10), se diseñó un ensayo de polimorfismo en la longitud de los fragmentos de restricción (Restriction Fragment Length Polymorphism, RFLP) de este gen,para determinar la especificidad de los iniciadores empleados y descartar falsos positivos que pudieran afectar los límites de detección y la reproducibilidad del experimento.
El ensayo en la simulación computacional se hizo con secuencias de nucleótidos del gen Cyt b de humanos y de L. evansi de la región Caribe obtenidos en trabajos previos (datos de Pérez-Doria, et al., aún no publicados). Con el programa Geneious 8 (22) se buscaron los sitios de corte de enzimas de restricción en la secuencia del segmento amplificado del gen Cyt b y se encontró que la enzima D corta el ADN de humanos pero no el de L. evansi. El sitio de corte de la endonucleasa DdeI (Promega) corresponde a la secuencia 5’…C ¯TNA G…3’, lo que en el caso del gen humano genera dos bandas de 100 y 250 pb, aproximadamente, mientras que por no poseer sitio blanco para la enzima, en L. evansi se mantiene una sola banda de 350 pb, aproximadamente.
La digestión de todos los productos amplificados se llevó a cabo en un volumen de reacción de 20 µl, en el que se depositaron 3 µl del producto de la PCR, cinco unidades de la enzima de restricción, 2 µl de solución tampón RE 10X y 0,3 µl de albúmina de suero bovino (10 µg/µl). La incubación se hizo siguiendo las indicaciones del fabricante (Promega).
Con los ensayos de RFLP se depuraron los resultados iniciales obtenidos por PCR, y las muestras con los productos de la PCR de insecto se descartaron del análisis para determinar el tiempo efectivo de detección del gen Cyt b. Además, se evaluó la relación entre el tiempo posterior a la ingestión, agrupado en categorías de dos días, y la reproducibilidad de los resultados mediante un análisis de proporciones con la prueba de ji al cuadrado en el programa estadístico R (23).
Todos los experimentos fueron avalados por el Comité de Bioética de la Universidad de Sucre, según consta en el Acta N° 02 del 18 de octubre de 2011. La muestra de sangre humana se obtuvo previo consentimiento informado.
Resultados
Del grupo inicial de 100 flebotomíneos recolectados, solo 80 se alimentaron bajo las condiciones experimentales y, de estos, 57 hembras se identificaron como L. evansi (cuadro 1). Los 23 individuos restantes murieron en las jaulas de cría y, como no se pudo determinar el tiempo exacto de digestión, se excluyeron del análisis.
Durante la observación diaria de cada grupo de hembras sacrificadas, se registraron los estados de digestión descritos a continuación. Entre las 0 y las 24 horas, L. evansi presentó un estado de digestión II, con el abdomen de color rojo y completamente lleno de sangre. El estado de digestión III se observó en hembras sacrificadas entre las 48 y 72 horas, durante las cuales, a pesar de que la presencia de sangre fue notoria, entre dos y tres segmentos abdominales se observaron vacíos. El estado de digestión V se registró a las 96 horas de la ingestión, momento en el que la sangre solo ocupaba tres o cuatro segmentos del abdomen. A las 120 horas, los individuos mostraron un estado de digestión VI, en el cual la sangre ocupaba dos segmentos del abdomen, mientras que las hembras sacrificadas a las 144 y a las 168 horas presentaron el estado de digestión VII, en el que no se observó sangre en el abdomen a pesar de estar distendido (figura 1).

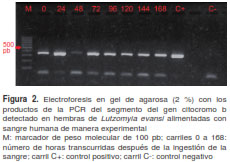
Con la técnica de PCR se amplificó un segmento de 358 pb del gen Cyt b en el 93 % de las hembras analizadas (n=57) y en todos los grupos evaluados (figura 2), aunque en algunos de los individuos sacrificados a las 72, 96 y 120 horas no hubo amplificación. Mediante el análisis de especificidad con la técnica de RFLP, se determinó que en 86 % (n=49/57) de las hembras de L. evansi estudiadas, el segmento amplificado correspondía al gen Cyt b de humanos, mientras que en 7 % (n=4/57) de estas correspondía al gen Cyt b del insecto (cuadro 1).
Con estos valores se hizo el análisis estadístico en el cual no se encontraron diferencias entre los grupos evaluados en cuanto a la capacidad de amplificar el ADN de humanos, tal como lo indicó el análisis de proporciones (c 2 =4,41; gl=3; p=0,220), es decir, todas las comparaciones entre los grupos arrojaron valores de p mayores de 0,050, lo que permite concluir que las proporciones no fueron diferentes.
Discusión
El tiempo efectivo de detección del segmento de 358 pb del gen Cyt b de humanos en la sangre contenida en el tubo digestivo de hembras de L. evansi fue de 168 horas a partir del momento de la ingestión. Este margen de tiempo es el más alto registrado hasta la fecha para la detección del gen Cyt b en flebotomíneos y, también, es superior al observado con los demás marcadores moleculares usados en la determinación de las fuentes de ingestión de sangre. En trabajos previos, la detección de este gen no superó las 96 horas después de la alimentación (5,24-26). Además, en otros estudios solo se logró amplificar el gen de vertebrados en 65 % (24) y 72 % (10) de los flebotomíneos evaluados, mientras que en el presente trabajo se amplificó en 86 % de estos (n=49/57).
Al comparar los resultados obtenidos con el tiempo de detección del gen nuclear PNOC, que aparece como alternativa para la caracterización de la ingestión de sangre en flebotomíneos, el gen Cyt b ofrece un umbral de tiempo siete veces mayor, puesto que el gen PNOC solo es detectado hasta las 24 horas después de la ingestión (5,11). De esta manera, la ventaja que se obtiene del amplio límite de detección del gen Cyt b y del mayor porcentaje de muestras detectadas compensaría la amplificación inespecífica del genoma del insecto, que en trabajos previos alcanzó a registrarse en 26 % de las muestras analizadas (10), en tanto que en el presente estudio no superó el 7 % (n=4/53). Más aún, el límite de detección de 168 horas reportado en esta investigación es superior al encontrado con técnicas tradicionalmente más sensibles y costosas, como la PCR en tiempo real, con las que el mismo gen pudo detectarse como máximo hasta los cinco días después de la ingestión (27).
Probablemente, el amplio rango de detección del gen Cyt b observado en este trabajo se debe, en parte, al gran número de copias presentes en la célula, que puede alcanzar los 200 (28), así como al tamaño de 358 pb de la región amplificada, la cual es lo suficientemente corta para escapar a los daños producidos en el ADN durante el proceso de digestión de las enzimas presentes en el intestino del insecto, y a que el gen posee un número suficiente de sitios polimorfos que permiten la identificación genética de la especie. Tampoco se puede descartar el potencial efecto de la solución tampón Q Plus usada en la amplificación, la cual mantiene la linealidad de la cadena de ADN.
Las características descritas hacen del gen Cyt b un marcador idóneo para la búsqueda de fuentes de ingestión de sangre, especialmente porque permitiría a los investigadores recuperar un mayor número de muestras para el análisis y, así, obtener un mejor conocimiento de los animales que intervienen en el ciclo eco-epidemiológico de la leishmaniasis. Teniendo en cuenta que el ciclo de Leishmania es complejo y que frecuentemente involucra diferentes reservorios y vectores en un mismo foco, el análisis cuantitativo de las especies de vectores y de sus preferencias alimenticias contribuye a entender mejor la dinámica de transmisión en dichos focos. Este conocimiento es relevante para el establecimiento de medidas de control de la leishmaniasis, al igual que de otras enfermedades asociadas a insectos hematófagos.
En conclusión, la amplificación del gen Cyt b es una herramienta útil para determinar las fuentes de ingestión de sangre en especímenes del género Lutzomyia recolectados en campo, donde se desconoce el tiempo exacto transcurrido desde la alimentación. En este trabajo se detectó ADN humano en la sangre ingerida por los insectos hasta 168 horas después de haberse alimentado, lo cual permitió caracterizar un mayor porcentaje de muestras en comparación con estudios previos. Esto indica que el gen se puede amplificar en cualquier punto del proceso de digestión de la sangre, incluso en insectos en los que no se observan restos de ella. Por último, desde una perspectiva de costobeneficio, la metodología aquí descrita es una técnica de fácil acceso.
Los autores manifiestan que no tienen ningún conflicto de intereses.
Este trabajo se desarrolló en el marco del proyecto de investigación “Estudio ecoepidemiológico de vectores de Leishmania spp. asociados a la aparición de casos autóctonos de leishmaniasis cutánea en núcleos urbanos de la costa Caribe colombiana”, financiado por el Departamento Administrativo de Ciencia, Tecnología e Innovación – Colciencias (código 112954531473). Daniel Verbel-Vergara recibió una beca-pasantía del programa de formación de jóvenes investigadores e innovadores “Virginia Gutiérrez de Pineda”, de Colciencias, 2012.
Correspondencia:
José Gabriel Vergara, Grupo de Investigaciones Biomédicas, Universidad de Sucre, Carrera 14 N° 16B-32, Sincelejo, Colombia Teléfono: (575) 282 0830; fax: (575) 282 1240 josegabriel_vergara@yahoo.com
1. Ashford RW. Leishmaniasis reservoirs and their significance in control. Clin Dermatol. 1996;14:523-32. https://doi.org/10.1016/0738-081X(96)00041-7
2. Tempelis CH. Host-feeding patterns of mosquitoes, with review of advances in analysis of blood meals by serology. J Med Entomol. 1975;11:635-53 https://doi.org/10.1093/jmedent/11.6.635
3. Morrison AC, Ferro C, Tesh RB. Host preferences of the sand fly Lutzomyia longipalpis at an endemic focus of American visceral leishmaniasis in Colombia. Am J Trop Med Hyg. 1993;49:68-75. https://doi.org/10.4269/ajtmh.1993.49.68
4. Nery LC, Lorosa NE, Franco AM. Feeding preference of the sand flies Lutzomyia umbratilis and L. spathotrichia (diptera: Psychodidae, Phlebotominae) in an urban forest patch in the city of Manaus, Amazonas, Brazil. Mem Inst Oswaldo Cruz. 2004;99:571-4. https://doi.org/10.1590/S0074-02762004000600006
5. Haouas N, Pesson B, Boudabous R, Dedet JP, Babba H, Ravel C. Development of a molecular tool for the identification of Leishmania reservoir hosts by blood meal analysis in the insect vectors. Am J Trop Med Hyg. 2007;77:1054-9.
6. Marassa AM, Consales CA, Galati EA. Enzyme-linked immunosorbent assay biotin/avidin method standardization, for identification of Lutzomyia (Lutzomyia) longipalpis bloodmeals (Lutz & Neiva, 1912). Rev Soc Bras Med Trop. 2004;37:441-6. https://doi.org/10.1590/S0037-86822004000600003
7. Gebresilassie A, Abbasi I, Aklilu E, Yared S, Kirstein OD, Moncaz A, et al. Host-feeding preference of Phlebotomus orientalis (Diptera: Psychodidae) in an endemic focus of visceral leishmaniasis in northern Ethiopia. Parasit Vectors. 2015;8:270. https://doi.org/10.1186/s13071-015-0883-5
8. Omondi D, Masiga DK, Ajamma YU, Fielding BC, Njoroge L, Villinger J. Unraveling host-vector-arbovirus interactions by two-gene high resolution melting mosquito bloodmeal analysis in a Kenyan wildlife-livestock interface. PLoS One. 2015;10:e0134375. https://doi.org/10.1371/journal.pone.0134375
9. Osorio HC, Ze-Ze L, Amaro F, Nunes A, Alves MJ. Sympatric occurrence of Culex pipiens (Diptera, Culicidae) biotypes pipiens, molestus and their hybrids in Portugal, Western Europe: Feeding patterns and habitat determinants. Med Vet Entomol. 2014;28:103-9. https://doi.org/10.1111/mve.12020
10. Paternina LE, Verbel-Vergara D, Romero-Ricardo L, Pérez-Doria A, Paternina-Gómez M, Martínez L, et al. Evidence for anthropophily in five species of phlebotomine sand flies (Diptera: Psychodidae) from northern Colombia, revealed by molecular identification of bloodmeals. Acta Trop. 2016;153:86-92. https://doi.org/10.1016/j.actatropica.2015.10.005
11. Baum M, de Castro EA, Pinto MC, Goulart TM, Baura W, Klisiowicz Ddo R, et al. Molecular detection of the blood meal source of sand flies (Diptera: Psychodidae) in a transmission area of American cutaneous leishmaniasis, Paraná State, Brazil. Acta Trop. 2015;143:8-12. https://doi.org/10.1016/j.actatropica.2014.11.006
12. Kirstein F, Gray JS. A molecular marker for the identification of the zoonotic reservoirs of Lyme borreliosis by analysis of the blood meal in its European vector Ixodes ricinus. Appl Environ Microbiol. 1996;62:4060-5.
13. Dillon RJ, Lane RP. Bloodmeal digestion in the midgut of Phlebotomus papatasi and Phlebotomus langeroni. Med Vet Entomol. 1993;7:225-32. https://doi.org/10.1111/j.13652915.1993.tb00681.x
14. Dolmatova AV, Demina NA. Les phlébotomes (Phlebotominae) et les maladies qu’ils transmettent. Paris: Office de la Recherche Scientifique et Technique Outre-mer; 1971. p. 168.
15. Young DG, Duran MA. Guide to the identification and geographic distribution of Lutzomyia sand flies in México, the West Indies, Central and South America (Diptera:Psychodidae). Mem Am Entomol Inst. 1994;54:1-881.
16. Galati EA. Phlebotominae (Diptera, Psychodidae). Classificação morfologia e terminologia e identificação de adultos. En: Rangel EF, Lainson R, editores. Flebotomíneos do Brasil. Rio de Janeiro: FIOCRUZ; 2003. p. 367.
17. Pérez-Doria A, Bejarano EE, Sierra D, Vélez ID. Molecular evidence confirms the taxonomic separation of Lutzomyia tihuiliensis from Lutzomyia pia (Diptera: Psychodidae) and the usefulness of pleural pigmentation patterns in species identification. J Med Entomol. 2008;45:653-9 https://doi.org/10.1093/jmedent/45.4.653
18. Kocher TD, Thomas WK, Meyer A, Edwards SV, Paabo S, Villablanca FX, et al. Dynamics of mitochondrial DNA evolution in animals: Amplification and sequencing with conserved primers. Proc Natl Acad Sci USA. 1989;86:6196-200
19. Oshaghi MA, Chavshin AR, Vatandoost H, Yaaghoobi F, Mohtarami F, Noorjah N. Effects of post-ingestion and physical conditions on PCR amplification of host blood meal DNA in mosquitoes. Exp Parasitol. 2006;112:232-6 https://doi.org/10.1016/j.exppara.2005.11.008
20. Steuber S, Abdel-Rady A, Clausen PH. PCR-RFLP analysis: A promising technique for host species identification of blood meals from tsetse flies (Diptera: Glossinidae). Parasitol Res. 2005;97:247-54. https://doi.org/10.1007/s00436-005-1410-y
21. Svobodova M, Alten B, Zidkova L, Dvorak V, Hlavackova J, Myskova J, et al. Cutaneous leishmaniasis caused by Leishmania infantum transmitted by Phlebotomus tobbi. Int J Parasitol. 2009;39:251-6 https://doi.org/10.1016/j.ijpara.2008.06.016
22. Kearse M, Moir R, Wilson A, Stones-Havas S, Cheung M, Sturrock S, et al. Geneious Basic: An integrated and extendable desktop software platform for the organization and analysis of sequence data. Bioinformatics. 2012;28:1647-9. https://doi.org/10.1093/bioinformatics/bts199
23. RCoreTeam. R: A language and environment for statistical computing. Vienna: R Foundation for Statistical Computing; 2015.
24. Abbasi I, Cunio R, Warburg A. Identification of blood meals imbibed by phlebotomine sand flies using cytochrome B PCR and reverse line blotting. Vector Borne Zoonotic Dis. 2009;9:79-86. https://doi.org/10.1089/vbz.2008.0064
25. Garlapati RB, Abbasi I, Warburg A, Poche D, Poche R. Identification of bloodmeals in wild caught blood fed Phlebotomus argentipes (Diptera: Psychodidae) using cytochrome b PCR and reverse line blotting in Bihar, India. J Med Entomol. 2012;49:515-21. https://doi.org/10.1603/ME11115
26. Sant’Anna MR, Jones NG, Hindley JA, Mendes-Sousa AF, Dillon RJ, Cavalcante RR, et al. Blood meal identification and parasite detection in laboratory-fed and field-captured Lutzomyia longipalpis by PCR using FTA databasing paper. Acta Trop. 2008;107:230-7. https://doi.org/10.1016/j.actatropica.2008.06.003
27. Sales KG, Costa PL, de Morais RC, Otranto D, Brandao-Filho SP, Cavalcanti MP, et al. Identification of phlebotomine sand fly blood meals by real-time PCR. Parasit Vectors. 2015;8:230. https://doi.org/10.1186/s13071-015-0840-3
28. Wallace DC. Mitochondrial DNA sequence variation in human evolution and disease. Proc Natl Acad Sci USA. 1994;91:8739-46.