Biomédica 2017;37(Supl.2):208-14
doi: https://doi.org/10.7705/biomedica.v34i2.3435
COMUNICACIÓN BREVE
1 Laboratorios de Biología Molecular y Celular, Escuela de Medicina Humana, Facultad de Ciencias de la Salud, Universidad Científica del Sur, Lima, Perú
2 Departamento Académico de Ciencias Básicas, Universidad Nacional Amazónica de Madre de Dios, Madre de Dios, Perú
3 Laboratorio de Referencia Nacional de Leishmaniasis, Instituto Nacional de Salud, Lima, Perú
4 Laboratorio de Referencia Nacional de Biotecnología y Biología Molecular, Instituto Nacional de Salud, Lima, Perú
Contribución de los autores:
Jesús E. Rojas-Jaimes, Germán H. Correa-Nuñez: concepción y diseño del estudio, recolección e interpretación de los datos y redacción del manuscrito
Nyshon Rojas-Palomino: diseño del estudio molecular para identificación de especies de Leishmania y análisis de resultados
Omar Cáceres-Rey: diseño del estudio molecular
Todos los autores participaron en la revisión crítica del manuscrito.
Recibido: 27/07/16; aceptado: 16/08/17
Introducción. En estudios previos se detectó la presencia de Leishmania infantum en Rhipicephalus sanguineus, lo cual planteaba la posibilidad de que R. sanguineus transmitiera la leishmaniasis a una variedad de huéspedes.
Objetivo. Identificar Leishmania (Viannia) spp. en garrapatas recolectadas en animales silvestres de una zona endémica para leishmaniasis.
Materiales y métodos. Se hicieron 81 extracciones individuales de ADN en las garrapatas recogidas de tres tapires o dantas (Tapirus terrestres) y tres pecaríes de collar (Pecari tajacu)cazados en Madre de Dios, Perú. Las garrapatas recolectadas se identificaron taxonómicamente y se prepararon para la identificación del cinetoblasto (kDNA) de Leishmania (Viannia) spp. mediante reacción en cadena de la polimerasa (PCR), así como de la especie de Leishmania mediante PCR de fusión de alta resolución (High Resolution Melt, HRM).
Resultados.Se detectó el kDNA de Leishmania (V) spp. en tres garrapatas silvestres de R. (Boophilus) microplus, Canestrini, 1888, recolectadas en un pecarí de collar cazado en la selva de Madre de Dios. El análisis mediante HRM-PCR evidenció que una de las muestras positivas de kDNA tenía una curva compatible con L. (V) guyanensis.
Conclusión. Los resultados evidenciaron la presencia de ADN de L. (V) guyanensis en R. (Boophilus) microplus, probablemente adquirida después de picar al pecarí. Es importante hacer nuevos estudios para aclarar la participación de R. (Boophilus) microplus en la transmisión de la leishmaniasis.
Palabras clave: Leishmania guyanensis; leishmaniasis; Rhipicephalus; artiodáctilos; Perú
doi: https://doi.org/10.7705/biomedica.v34i2.3435
Detection ofLeishmania (V) guyanensis inRhipicephalus (Boophilus)microplus (Acari:Ixodidae) collected from Pecari tajacu
Introduction: Previous studies identified the presence of Leishmania infantum in Rhipicephalus sanguineus and indicated the possibility that it could transmit leishmaniasis to a variety of hosts.
Objective: To identify parasites of Leishmania (Viannia) spp. in ticks collected from wild animals in an endemic area for leishmaniasis.
Materials and methods: We performed 81 individual DNA extractions from ticks collected from three Tapirus terrestris and three Pecari tajacu in Madre de Dios, Perú. Ticks were taxonomically identified and they were subsequently prepared to identify Leishmania (Viannia) spp. kDNA by PCR and the species of Leishmania by HRM-PCR.
Results: Leishmania (Viannia) kDNA was detected in three wild ticks of the species R. microplus, collected from a collard peccary (P. tajacu) hunted in the forests of Madre de Dios. The HRM-PCR showed that one of the positive samples had a kDNA curve compatible with L. (V) guyanensis.
Conclusion: The results showed the presence of L. (V) guyanensis DNA in R. microplus possibly acquired after biting a collarde peccary. Therefore, it is important to design future studies to clarify R. microplus involvement in the transmission of leishmaniasis.
Key words: Leishmania guyanensis; leishmaniasis; Rhipicephalus; artiodactyla; Peru.
doi: https://doi.org/10.7705/biomedica.v34i2.3435
En América, los flebotomíneos del género Lutzomyia son vectores de parásitos del género Leishmania. Sin embargo, se ha sugerido que las garrapatas también pueden ser vectores de especies de Leishmania, como lo reporta un estudio previo en algunos lugares de Suramérica donde la leishmaniasis está presente y el vector natural no se ha encontrado (1). En otro estudio se detectó Leishmania infantum en Rhipicephalus sanguineus recolectadas de perros, los cuales podrían infectar a los humanos (2).
Estos datos sugieren que existen otros artrópodos que pueden ser vectores de la leishmaniasis. Además, en un estudio se encontró que 50 % de las garrapatas analizadas estaban infectadas con Leishmania spp. viables, según la identificación del ARN de parásitos en perros que sufrían de leishmaniasis visceral (3). Este porcentaje es mayor que el que se encuentra en el vector natural, es decir, Lutzomyia longipalpis (3). En este contexto, es necesario aclarar si las garrapatas son vectores de la leishmaniasis en los perros. Otros resultados dicientes sobre la posible relación entre garrapatas y leishmaniasis fue la detección de la transmisión transovárica de L. infantum en R. sanguineus (3). Sin embargo, la hipótesis de que R. sanguineus es un vector sigue generando controversia, especialmente en áreas en donde aún no se ha encontrado el vector natural (2,4). Además, en otros estudios se encontró ADN del cinetoplasto (kDNA) de L. infantum en la glándula salival de R. sanguineus en Brasil e Italia (5-7).
Por otro lado, el pecarí de collar, Pecari tajacu (Linnaeus, 1758), presenta un amplio rango de distribución y es la base de la alimentación de diversas comunidades indígenas y, además, existe un apreciable intercambio de pieles de pecarí entre Perú y Brasil, por lo que el contacto con este animal y con sus ectoparásitos, como las garrapatas, es evidente, especialmente entre los nativos, los agricultores, los mineros y los investigadores que ingresan a su hábitat (8). Esta especie vive en manadas de ocho individuos en promedio, sus hábitos son diurnos y nocturnos, y en sus recorridos puede llegar a abarcar un área de 123 hectáreas, en promedio, por lo cual podría ocasionar una significativa dispersión de ectoparásitos (8). Actualmente, no hay información sobre si el pecarí de collar es reservorio o huésped de parásitos del género Leishmania. Dicha asociación debe investigarse dado que ya se ha detectado Leishmania spp. en garrapatas recolectadas en perros (7).
En este sentido, el objetivo del presente estudio fue la detección e identificación molecular de Leishmania spp. en garrapatas recolectadas en ejemplares de P. tajacu y Tapirus terrestris, los cuales son cazados por los pobladores del área de estudio para su subsistencia, y pueden constituir un factor de riesgo de transmisión de patógenos a la población que entra en contacto con ellos.
Materiales y métodos
Aspectos éticos y trabajo de campo
Esta investigación fue aprobada por la Oficina de Salud Pública y Medio Ambiente del Consejo Regional de Madre de Dios, Perú. En el estudio, las pruebas para la identificación de Leishmania spp. se completaron con procedimientos de laboratorio ajustados a las guías internacionales sobre la investigación con animales y las normas del Comité de Cuidado y Uso de Animales del área de Investigación en Salud de la Junta del Consejo Regional de Madre de Dios.
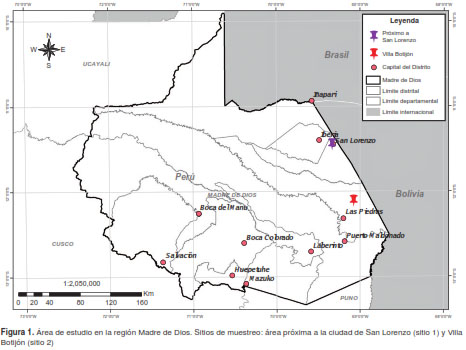
El estudio se llevó a cabo en dos lugares de la cuenca del río Tahuamanu, situado en la región de Madre de Dios, en las provincias de Tahuamanu y Tambopata, Perú, cerca de la frontera con Bolivia. El sitio 1 incluía el área cercana a la ciudad de San Lorenzo (11°27’13,73”S, 69°20’2,54”O; World Geodetic System (WGS) 84, 285 msnm), capital del distrito de Tahuamanu, en la provincia de Tahuamanu, y el sitio 2, a Pueblo Botijón (12°07’12,95”S, 69°04’31,47”O; WGS 84, 267 msnm), cercano a la ciudad de Planchón, capital del distrito de Las Piedras, en la provincia de Tambopata (figura 1).

Los sitios de recolección se ubicaron en una zona forestal donde normalmente hay caza de animales silvestres sin restricción, pues está fuera de las zonas de reserva. Además, alrededor de los sitios de estudio, en el distrito de Las Piedras, hay áreas endémicas de leishmaniasis en donde los pacientes han reportado que la enfermedad se produce por la picadura de las garrapatas, información que ha sido registrada por el personal de salud de la Dirección Regional de Salud de Madre de Dios en el curso de la vigilancia epidemiológica. En el periodo de 12 meses del estudio, se recogieron ocho reportes orales de este tipo. En cuanto al clima, el área de estudio tiene una precipitación media anual de 1. 600 mm y una temperatura media anual de 25 °C . El área se clasifica en la zona de vida de selva húmeda tropical. Durante la recolección el clima fue cálido y húmedo.
En junio de 2012, se entró en contacto con la población local con el fin de hacer los arreglos necesarios para desarrollar la recolección de garrapatas según el proyecto aprobado. Los animales silvestres provenían de la caza local para el consumo. A las tres horas del sacrificio del animal, se recolectaron con una pinza las garrapatas de la región abdominal y se colocaron de forma individual en crioviales de 2 ml con alcohol etílico al 96 %, que luego se rotularon con un código de identificación del lugar de recolección y el animal del cual provenía la muestra.
Se seleccionaron los animales que presentaban más infestación con garrapatas, abundantes en la zona, ya que sus ectoparásitos se depositan en las hojas de las plantas, lo cual constituye un factor de riesgo para la infestación de garrapatas en los humanos.
Clasificación taxonómica y estudios moleculares
Las garrapatas se trasladaron al Laboratorio de Entomología del Instituto Nacional de Salud del Perú en Lima, y se identificaron utilizando las claves de Barros-Battesti, et al. (9).
Para la identificación de los parásitos de Leishmania, inicialmente se extrajo el ADN de las garrapatas con el estuche Gentra Puregene (QIAGEN) siguiendo el protocolo de aislamiento de ADN en tejidos (10).
Las garrapatas se cortaron en cuatro a seis piezas y se colocaron en un microtubo de 1,5 ml con 300 µl de solución de lisis celular. Dependiendo del tamaño de la garrapata, se agregó un volumen de proteinasa K (1,5 µl, 5 µl o 20 µl), se mezcló suavemente con el mezclador de vórtice, se centrifugó solo si era necesario, y se incubó a 55 °C durante toda la noche. Luego se añadieron 100 µl de la solución hipertónica para precipitación de proteínas, se agitó vigorosamente durante 20 segundos, y se centrifugó durante 10 minutos a 13.000 rpm. El sobrenadante se recolectó y se mezcló con 300 µl de isopropanol frío y se agitó suavemente por inversión 50 veces para después centrifugarlo durante 10 minutos a 13.000 rpm, y mantenerlo en el congelador durante una hora a -10 °C. Posteriormente, se desechó el sobrenadante con cuidado, se colocó el tubo invertido en un pedazo de papel absorbente blanco y se dejó secar al aire durante 5 minutos. Se añadieron 60 µl de solución de hidratación (agua para biología molecular) y se agitó durante cinco segundos. Por último, se incubó a 65 °C durante una hora para hacer soluble el ADN.
Se utilizó una PCR en cada muestra para detectar el ADN del cinetoplasto (kDNA) de Leishmania (Viannia) spp. Los cebadores para la amplificación fueron los siguientes: MP1-L: 5’-TAC TCC CCG ACA TGC CTC TG-3’ y MP3-H: 5’GAA CGG GGT TGT TTC ATG C-3’ (11). Las reacciones se llevaron a cabo en un volumen final por reacción de 20 µl, que contenían 4 µl de ADN, 2 µl de solución tampón 10X PCR (Invitrogen), 1 µl de cada cebador (10 µM), 0,2 µl de Taq ADN polimerasa (5 U/µl) (Invitrogen, Grand Island, NY), 0,6 µl de 50 mM de MgCl2 y 2 µl de 1,25 mM de cada dNTP. Se hizo desnaturalización inicial a 94 °C durante cinco minutos, seguidos de 35 ciclos de desnaturalización a 94 °C durante 45 segundos, hibridación a 58 °C durante 45 segundos, una extensión a 72 °C durante 60 segundos, y una extensión final a 72 °C durante cinco minutos. Como control positivo se usó la cepa de referencia LTB-300 (L. (V) braziliensis) y, como control negativo, agua para biología molecular. El producto esperado fue de 70 pb y se visualizó usando geles de agarosa al 2 %.
Para la identificación de Leishmania spp., se utilizó una PCR de fusión de alta resolución (HRM-PCR) con una concentración de 5 ng/µl de ADN y 0,7 µM de los cebadores OL1 – Directo 5´ GGG GAG GGG CGT TCT GCG AA 3´ y OL2- Reverso 5´CCG CCC CTA TTT TAC ACC AAC CCC 3´ (12). La PCR-HRM consistió en 35 ciclos así: desnaturalización a 95 °C durante 10 segundos, hibridación a 55 °C durante 30 segundos y una extensión a 72 °C durante 10 segundos, seguidos por un aumento progresivo de la temperatura de 1 °C en el intervalo de 75 a 95 °C para lograr la desnaturalización del amplicón de doble cadena y la disociación del fluoróforo (Eva Green, Qiagen).
El análisis de las curvas de disociación para la diferenciación de especies de Leishmania y el kDNA positivo se hizo en las áreas de las muestras con problemas, las cuales se compararon con las áreas de las cepas de referencia mediante el algoritmo del programa Rotor Gene Q, versión V2.1.0. Solo las áreas con una confianza de similitud mayor de 90 % comparada con el área de la cepa de referencia, se definieron como muestras ya genotipificadas. Este método permitió identificar cuatro curvas diferentes de modelo, que correspondían a cada una de las siguientes cepas de referencia de Leishmania: Leishmania (V) braziliensis, L. (V) peruviana, L. (V) guyanensis y L. (Leishmania) amazonensis. Como control negativo, se utilizó el ADN de garrapata negativo para kDNA (código 80) y, como control blanco, el agua para biología molecular. Para las PCR ejecutadas no se realizaron pruebas de inhibición ni tampoco nuevas amplificaciones de las muestras con poca cantidad de ADN blanco.
Resultados
Se recolectaron 81 garrapatas, de las cuales 40 (49,4 %) correspondieron al género Amblyomma y proveníandetres ejemplares adultos de T. terrestres,y las otras 41 (50,6 %) pertenecían al género Rhipicephalus y provenían de tres ejemplares adultos de P. tajacu.
Mediante el análisis de la PCR del kDNA de las garrapatas recolectadas, se detectaron tres muestras positivas para Leishmania (Viannia) spp. (figura 2), una con una banda muy débil. Todas las garrapatas positivas eran machos de la especie R. (Boophilus) microplus recolectados en ejemplares de P. tajacu en la localidad de Botijón, pero no se determinó a cuál de los tres pecaríes pertenecían; la prevalencia fue de 7,3 % (IC95% 1,5-19,9).
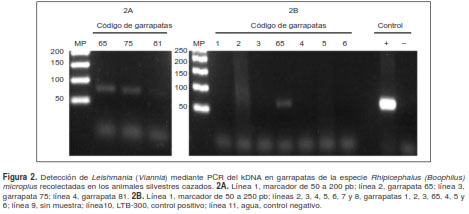
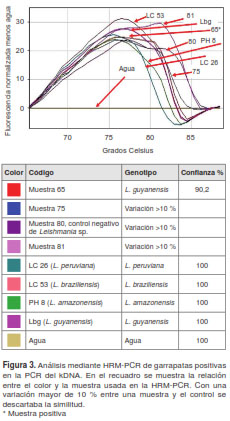
El análisis de HRM-PCR en las garrapatas positivas evidenció que solo una de ellas (código 65) tenía una curva similar a Leishmania (V) guyanensis (figura 3), en tanto que en las otras dos no se pudo detectar correspondencia con las curvas de referencia. En el caso de la correspondiente al código 80, aunque sí generó una curva, las condiciones de saturación de la PCR no permitieron relacionarla con ninguna de las cepas de referencia, además de ser negativa en la PCR del kDNA. Por otra parte, todas las restantes garrapatas de las cinco especies del género Amblyomma fueron negativas en esta PCR (cuadro 1).

Discusión
Las garrapatas son endémicas en India, Asia, Australia, Madagascar, áfrica, el Caribe, México, Centroamérica y Suramérica (9), y se consideran como el parásito del ganado más importante del mundo, aunque también infectan otros animales domésticos y salvajes (13,14). Rhipicephalus (Boophilus) microplus se ha reportado como vector de Babesia bigemina, B. bovis y Anaplasma marginale y está presente en regiones tropicales y subtropicales en todo el mundo (15,16), pertenece a la familia Ixodidae, y parasita ovejas y ganado vacuno (16).Tiene una gran capacidad invasiva debido a su gran movilidad para buscar alimento y se distribuye ampliamente en América, áfrica y Oceanía (17). Desde el punto de vista económico, R. microplus genera notables pérdidas en la ganadería bovina y ovina debido a la anemia que provoca en los animales por la succión de la sangre y por la trasmisión de agentes patógenos causantes de enfermedades como la babesiosis y la anaplasmosis (18).
En Perú, R. microplus se ha reportado como un parásito del ganado y, en Brasil, R. sanguineus se considera un parásito de perros, así como potencial vector de L. infantum (19-21) y L. braziliensis (20). Debe señalarse, además, que en estudios anteriores en Italia y Brasil se encontraron porcentajes más altos de kDNA (10,2 y 12,3 %, respectivamente) de L. infantum en R. sanguineus en comparación con los porcentajes encontrados en especies de Lutzomyia (<1 %), que son los vectores naturales (2,7).
En el caso de este estudio, el porcentaje de kDNA del parásito fue de 7 % (3/41), lo cual confirma que una gran proporción de garrapatas puede infestarse con Leishmania spp. Con la HRM-PCR se pudo identificar la especie patógena L. guyanensis. Esta prueba se validó (HRM-PCR Vs. secuenciación) en muestras humanas en el Laboratorio de Biología Molecular y Biotecnología del Instituto Nacional de Salud de Perú (manuscrito en preparación para publicación).
La PCR del kDNA para detectar Leishmania ( Viannia) spp. y la HRM-PCR para identificar la especie del parásito, se emplearon en el intento de amplificar el citocromo b para su posterior secuenciación, aunque sin éxito. El citocromo b se localiza en el maxicírculo del cinetoplasto con un menor número de copias que el minicírculo, también localizado en este (22,23), y se empleó como sección de molde para la amplificación mediante los dos tipos de PCR. El fallido intento de secuenciación usando el citocromo b se explicaría por el bajo número de parásitos en R. microplus. En el caso de las muestras con los códigos 75 y 81, aunque fueron positivas en la PCR del kDNA, presentaron bandas muy tenues, lo cual hace presumir la poca cantidad del genoma presente en el parásito. Por la misma razón, no fue posible identificar la especie de parásito en estas muestras mediante HRM- PCR.
En este estudio las garrapatas infectadas con Leishmania spp. eran machos, lo cual coincide con los resultados de un estudio anterior en el que las garrapatas machos se encontraron infestados naturalmente con L. infantum en mayor proporción que las hembras. Ello podría explicarse por la necesidad que tienen los machos de moverse con el fin de encontrar pareja y alimento (2).
Este estudio es el primero en detectar e identificar molecularmente la especie L. guyanensis en ejemplares de R. microplus provenientes de un animal silvestre, el pecarí de collar. Aunque existe la posibilidad de que R. microplus pueda haberse contaminado al succionar sangre de otro animal diferente a P. tajacu, es de suma importancia investigar su papel como probable huésped o reservorio de Leishmania.
Los resultados de esta investigación apoyan la hipótesis de la posible trasmisión de especies patógenas de Leishmania al ser humano por la picadura de garrapatas alojadas en mamíferos silvestres de su consumo. Además, es importante señalar el peligro potencial que representa para la salud pública el incremento de la caza de cerdos silvestres y su crianza para alimento humano en granjas.
Futuras investigaciones sobre las garrapatas y su relación con mamíferos silvestres permitirán dar nuevas luces sobre la epidemiología de la leishmaniasis.
A Andrés Lescano, Maxy De Los Santos, Jocelyn Pérez y Leonardo Mendoza, por sus recomendaciones e importantes contribuciones.
Los autores declaran no tener conflicto de intereses con respecto a los contenidos del manuscrito.
Esta investigación fue financiada por la Organización Trees de Inglaterra.
Correspondencia:
Jesús Eduardo Rojas, km 19, Panamericana Sur, Villa El Salvador, Lima, Perú
Teléfono: (511) (99) 363 8840
1. Dantas-Torres F. Canine leishmaniosis in South America. Parasit Vectors. 2009;2(Suppl.1):1-8. https://doi.org/10.1186/1756-3305-2-S1-S1
2. Solano-Gallego L, Rossi L, Scroccaro A, Montarsi F, Caldin M, Furlanello T,et al. Detection of Leishmania infantum DNA mainly in Rhipicephalus sanguineus male ticks removed from dogs living in endemic areas of canine leishmaniosis. Parasit Vectors. 2012;5:98. https://doi.org/10.1186/1756-3305-5-98
3. Colombo FA, Odorizzi RM, Laurenti MD, Galati EA, Canavez F, Pereira-Chioccola VL. Detection of Leishmania (Leishmania) infantum RNA in fleas and ticks collected from naturally infected dogs. Parasitol Res. 2011;2:267-74. https://doi.org/10.1007/s00436-010-2247-6
4. Dantas-Torres F, Latrofa MS, Otranto D. Quantification of Leishmania infantum DNA in females, eggs, and larvae of Rhipicephalus sanguineus. Parasit Vectors. 2011;4:56. https://doi.org/10.1186/1756-3305-4-56
5. Dantas-Torres F. Ticks as vectors of Leishmania parasites.
Trends Parasitol. 2011;27:155-9. https://doi.org/10.1016/j.pt.2010.12.006
6. Otranto D, Testini G, Dantas-Torres F, Latrofa MS, Vissotto de Paiva Diniz PP, de Caprariis D,et al. Diagnosis of canine vector-borne diseases in young dogs: A longitudinal study. J Clin Microbiol. 2010;48:3316-24. https://doi.org/10.1128/JCM.00379-10
7. Dantas-Torres F, Testini LV, de Paiva-Cavalcanti M, Figueredo LA, Stanneck D, Mencke N,et al. Detection of Leishmania infantum in Rhipicephalus sanguineus ticks from Brazil and Italy. Parasitol Res. 2010;106:857-60. https://doi.org/10.1007/s00436-010-1722-4
8. Sabogal S. Filogeografía y conservación genética del pecarí de collar, Pecari tajacu, en cuatro departamentos de Colombia (tesis). Bogotá: Universidad Nacional de Colombia; 2010.
9. Barros-Battesti D, Arzua M, Bechara H. Carrapatos de Importância Medico-Veterinaria da Região Neotropical: Um Guia Ilustrado para Identificação de Espécies. 10ma edição. Sao Paulo: Butantan Publicação; 2006. p. 223.
10. QIAGEN. Gentra®, Puregene® (QIAGEN Group), 2007-2010. Fecha de consulta: 9 de junio de 2017. Disponible en: https://www.qiagen.com/us/shop/sample-technologies/dna/genomicdna/gentra-puregene-tissue-kit/#orderinginformation.
11. López M, Inga R, Cangalaya M, Echevarría J, Llanos Cuentas A.Diagnosis the Leishmania using the polimerase chain reaction: A simplified procedure for field work. Am J Trop Med Hyg.1993;49:348-56. https://doi.org/10.4269/ajtmh.1993.49.348
12. Cabrera O, Munstermann L, Cárdenas R, Gutiérrez R, Ferro C. Definición de las condiciones de temperatura y almacenamiento adecuadas en la detección de ADN de Leishmania por PCR en flebotominos. Biomédica. 2002;22:296-302. https://doi.org/10.7705/biomedica.v22i3.1167
13. Labruna M, Romero M, Martins T, Tobler M, Ferrerira F.Ticks of the genus Amblyomma (Acari: Ixodidae) infesting tapirs (Tapirus terrestris) and peccaries (Tayassu pecari) in Perú. Syst Appl Acarol. 2010;15:109-12. https://doi.org/10.11158/saa.15.2.3
14. Otranto D, Dantas-Torres F.Canine and feline vector-borne diseases in Italy: Current situation and perspectives. Parasit Vectors. 2010;3:2. https://doi.org/10.1186/1756-3305-3-2
15. Rodríguez-Vivas R, Hodgkinson J, Trees A. Resistencia a los acaricidas en Rhipicephalus (Boophilus) microplus: situación actual y mecanismos de resistencia. Rev Mex Cienc Pecu. 2012;3:9-24.
16. Chen Z, Liu Q, Liu J, Xu B, Lv S, Xia S,et al. Tick-borne pathogens and associated co-infections in ticks collected from domestic animals in central China. Parasit Vectors. 2014;7:237. https://doi.org/10.1186/1756-3305-7-237
17. Madder M, Adehan S, De Deken R, Adehan R, Lokossou R. New foci of Rhipicephalus microplus in West Africa. Exp Appl Acarol. 2012;56:385-90. https://doi.org/10.1007/s10493-012-9522-4
18. Guerrero F, Andreotti R, Bendele K, Cunha R, MillerR, Yeater K,et al. Rhipicephalus (Boophilus) microplus aquaporin as an effective vaccine antigen to protect against cattle tick infestations. Parasit Vectors. 2014;7:475. https://doi.org/1010.1186/s13071-014-0475-9
19. Goncalvez L, Filgueira K, Ahid S, Pereira J, Mendes do Vale A, Machado R, et al.Study on coinfecting vectorborne pathogens in dogs and ticks in Rio Grande do Norte, Brazil. Rev Bras Parasitol Vet. 2014;23:407-12. https://doi.org/10.1590/S1984-29612014071
20. Silva de Morais R, Goncalves-de-Albuquerquea S, Pessoa e Silva R, Lemos P, Gaudêncio da Silva K, Brandão-Filho S,et al. Detection and quantification of Leishmania braziliensis in ectoparasites from dogs. Vet Parasitol. 2013;196:506-8. https://doi.org/10.1016/j.vetpar.2013.03.026
21. Campos J, Costa F. Participation of ticks in the infectious cycle of canine visceral leishmaniasis, in Teresina, Piauí, Brazil. Rev Inst Med Trop Sao Paulo. 2014;56:297-300. https://doi.org/10.1590/S0036-46652014000400005
22. Ceccarelli M, Galluzzi L, Migliazzo A, Magnani M. Detection and characterization of Leishmania (Leishmania) and Leishmania (Viannia) by SYBR green-based real-time PCR and high resolution melt analysis targeting kinetoplast minicircle DNA. PLoS One. 2014;9:e88845. https://doi.org/10.1371/journal.pone.0088845
23. Lopes E, Geraldo C, Marcili A, Silva R, Keid L, Oliverira T,et al.Performance of conventional PCRs based on primers directed to nuclear and mitochondrial genes for the detection and identification of Leishmania spp. Rev Inst Med Trop Sao Paulo. 2016;58:41. https://doi.org/10.1590/S1678-9946201658041