Biomédica 2017;37(Supl.2):167-79
doi: https://doi.org/10.7705/biomedica.v34i2.3559
ARTÍCULO ORIGINAL
1 Laboratorio de Biotecnología y Biología Molecular, Instituto Nacional de Salud, Lima, Perú
2 Laboratorio de Investigaciones en Parasitología Tropical, Universidad del Tolima, Ibagué, Colombia
3 Dirección Subregional de Salud de Cutervo, DIRESA Cajamarca, Cajamarca, Perú
4 Laboratorio de Entomología, Laboratorio de Referencia Regional de la Dirección de la Red de Salud Bagua, DIRESA Amazonas, Amazonas, Perú
5 área de Vigilancia y Control Vectorial, Unidad de Salud Ambiental, Dirección de la Red de Salud de Utcubamba, DIRESA Amazonas, Amazonas, Perú
6 Departamento de Microbiología y Patología, Facultad de Medicina, Universidad Nacional San Agustín, Arequipa, Perú
7 Laboratorio de Entomología, Laboratorio de Referencia Regional de Salud Pública, DIRESA Ucayali, Ucayali, Perú
8 Instituto de Medicina Tropical “Daniel A. Carrión”, Universidad Nacional Mayor de San Marcos, Lima, Perú
9 área de Entomología, Laboratorio de Referencia Regional de Amazonas, DIRESA Amazonas, Amazonas, Perú
10 Laboratorio de Entomología, Instituto Nacional de Salud, Lima, Perú
11 Sección de Entomología, Instituto de Medicina Tropical “Daniel A. Carrión” Universidad Nacional Mayor de San Marcos, Lima, Perú
12 Departamento Académico de Microbiología Médica, Facultad de Medicina Humana, Universidad Nacional Mayor de San Marcos, Lima, Perú
Contribución de los autores:
Carlos P. Padilla y Uriel Alvarado: procesamiento de muestras, detección molecular de las unidades discretas de tipificación y análisis de datos
Julio César Carranza: mantenimiento de las unidades discretas de tipificación de referencia
Gladis Ventura: procesamiento de muestras
Deysi Luna-Caipo, Marcial Suárez, José R. Tuñoque-Valdera, Luis A. Fachin y Lizandro Gonzáles: captura de triatominos e interacción con las personas de las diversas localidades de estudio
Nancy Ruelas-Llerena: trabajo de laboratorio, captura e identificación de triatominos, interacción con las personas de las diversas localidades de estudio de las provincias de Arequipa
Alina Huiza: trabajo de laboratorio e identificación de triatominos
Gustavo Adolfo Vallejo y Abraham G. Cáceres: diseño del proyecto y análisis de datos
Abraham G. Cáceres: captura e identificación de triatominos
Todos los autores participaron en la escritura del manuscrito.
Recibido: 18/08/16; aceptado: 18/08/17
Introducción. Trypanosoma cruzi se ha dividido en seis unidades taxonómicas discretas (Discreet Typing Units, DTU) denominadas TcI, TcII, TcIII, TcIV, TcV y TcVI. Aún se desconocen los factores determinantes de la dinámica de la transmisión vectorial de los genotipos de T. cruzi en las diferentes regiones geográficas de distribución de la enfermedad de Chagas en Perú.
Objetivo. Detectar y tipificar las unidades taxonómicas discretas de T. cruzi en las heces de siete especies de triatominos (Panstrongylus chinai, P. geniculatus, P. herreri, Rhodnius robustus, R. pictipes, Triatoma carrioni y T. infestans), capturados en ocho departamentos de diferentes regiones naturales de Perú.
Materiales y métodos. Se examinaron 197 insectos para la detección de tripanosomas. Se extrajo el ADN del contenido intestinal de cada insecto y se amplificó mediante reacción en cadena de la polimerasa (PCR) de los genes kDNA, SL-IR, 24Sa rRNA y 18Sa RNA para detectar las DTU de T. cruzi.
Resultados. Se detectaron cinco infecciones con T. rangeli y 113 con T. cruzi. De estas últimas, fue posible identificar 95 de TcI (dos en P. chinai, una en P. geniculatus, 68 en P. herreri, cuatro en R. pictipes, siete en R. robustus, una en T. carrioni, y 12 en T. infestans); cinco de TcII (cuatro en P. herreri, una en T. infestans); cuatro de TcIII (tres en P. herreri, una en R. robustus) y cuatro infecciones de TcIV en P. herreri.
Conclusión. Este es el primer trabajo de caracterización a gran escala de T. cruzi en el intestino de vectores de importancia epidemiológica en Perú, orientado a generar información básica que permita entender la dinámica de la transmisión vectorial de T. cruzi en esta región del continente.
Palabras clave: Trypanosoma cruzi; Trypanosoma rangeli; Triatominae; genotipo; Perú.
doi: https://doi.org/10.7705/biomedica.v34i2.3559
Identifying Trypanosoma cruzi discreet typing units in triatomines collected in different natural regions of Perú
Introduction: Trypanosoma cruzi has been divided by international consensus into six discrete typing units (DTU): TcI, TcII, TcIII, TcIV, TcV y TcVI. The factors determining the dynamics of T. cruzi genotypes vector transmission of Chagas’ disease in the different geographical regions of Perú are still unknown.
Objective: To detect and type T. cruzi DTUs from the faeces of seven species of triatomines (Panstrongylus chinai, P. geniculatus, P. herreri, Rhodnius robustus, R. pictipes, Triatoma carrioni and T. infestans) captured in eight departments from different natural regions of Perú.
Materials and methods: We examined 197 insects for detecting trypanosomes. DNA was extracted from each insect intestinal contents and PCR amplification of kDNA, SL-IR, 24Sa rRNA and 18Sa RNAwas performed for detecting T. cruzi DTUs.
Results: Five T. rangeli and 113 T. cruzi infections were detected; 95 of the latter were identified as TcI (two in P. chinai, one in P. geniculatus, 68 in P. herreri, four in R. pictipes, seven in R. robustus, one in T. carrioni, 12 in T. infestans), five as TcII (four in P. herreri, one in T. infestans), four as TcIII (three in P. herreri, one in R. robustus) and four TcIV infections in P. herreri.
Conclusions: This is the first study which has attempted a large-scale characterization of T. cruzi found in the intestine of epidemiologically important vectors in Perú, thus providing basic information that will facilitate a better understanding of the dynamics of T. cruzi vector transmission in Perú.
Key words: Trypanosoma cruzi; Trypanosoma rangeli; Triatominae; genotype; triatomines; Perú.
doi: https://doi.org/10.7705/biomedica.v34i2.3559
La enfermedad de Chagas es una zoonosis causada por Trypanosoma cruzi presente en 22 países de Latinoamérica, donde se estima que alrededor de ocho millones de personas están infectadas y 25 millones viven en áreas de riesgo (1). La transmisión de T. cruzi ocurre principalmente mediante la transmisión vectorial por contacto de las heces contaminadas de los triatominos con el huésped. Otras formas de transmisión se presentan por transfusión de sangre contaminada, trasplante de órganos infectados, transmisión congénita, accidentes de laboratorio y por vía oral debida a la ingestión de alimentos contaminados con heces del vector (1,2). Trypanosoma cruzi ha sido dividido en seis unidades discretas de tipificación (Discreet Typing Units, DTU): TcI, TcII, TcIII,TcIV, TcV y TcVI (3,4). Además, se han descrito nuevos genotipos como el Tc Bat, asociado con murciélagos en Brasil, Panamá y Colombia (5-8).
Se ha observado una distribución geográfica heterogénea de estos genotipos, así, por ejemplo, la TcI se ha detectado predominantemente en humanos, reservorios y vectores en Centroamérica, Colombia y Venezuela, en tanto que la TcII, la TcV y la TcVI son más comunes en los países del Cono Sur del continente americano (9-11). Al parecer, es posible encontrar todos los genotipos a lo largo del continente americano, pero con predominio de algunos de ellos en diferentes regiones geográficas. Pocas correlaciones se han observado entre la distribución de las DTU de T. cruzi y las características clínicas de la enfermedad (12,13). Asimismo, se han observado diferencias en la distribución de los genotipos de T. cruzi en los reservorios domésticos, por ejemplo, los perros en la región norte de América se han encontrado infectados predominantemente con TcI y, en el sur del continente, con TcIV, TcV y TcVI (14). Con relación a los reservorios silvestres, se ha observado que la mayoría de los Didelphis marsupialis y los reservorios arbóreos se encuentran infectados con TcI (15). Por ello, se ha planteado la hipótesis de que los reservorios arbóreos estarían infectados con TcI y los reservorios terrestres con las unidades TcII a TcVI (16). Sin embargo, se han encontrado varios reservorios arbóreos, como Monodelphis brevicaudata, Philander frenatus y Didelphis aurita, infectados con TcII, TcIV y TcII, respectivamente (5,10).
En cuanto a los genotipos de T. cruzi aislados de triatominos, en varios estudios se ha demostrado la asociación de genotipos de T. cruzi en algunas especies de vectores, por ejemplo, se ha encontrado predominancia de TcI en el intestino de Rhodnius prolixus, R. colombiensis y R. pallescens en vectores domésticos y silvestres en Colombia, con prevalencias de 2,1 a 78,6 % (17). Además, se ha observado que la TcI se encuentra asociada a los ciclos domésticos y silvestres en Colombia, Venezuela y en algunos países de Centroamérica, y a los ciclos selváticos en la Amazonia, en tanto que TcII, TcV y TcVI se han relacionado con ciclos domiciliarios en los países del Cono Sur de Suramérica. Los linajes TcIII y TcIV son los más raros, y se han relacionado con la transmisión selvática, con pocos reportes de infección en humanos (18).
A pesar de los estudios sobre la epidemiología molecular de T. cruzi, aún existen varios interrogantes sobre los factores que determinan la dinámica de transmisión de los genotipos de T. cruzi en las diferentes regiones endémicas de la enfermedad de Chagas. Asimismo, en los estudios sobre la interacción entre parásito y vector se ha señalado la presencia de factores de la respuesta inmunitaria del insecto (19) o de la microbiota intestinal, que estarían facilitando o impidiendo la transmisión de los genotipos de T. cruzi (20,21).
En Perú, se han documentado casos de enfermedad de Chagas en varias localidades de los departamentos de Arequipa, Moquegua, Madre de Dios, Lambayeque, Ica, Tacna, Ucayali, Loreto, San Martín, Cajamarca, Pasco y Amazonas (22). En un estudio sobre seroprevalencia de T. cruzi en humanos, perros y cobayos, se encontraron porcentajes de 14,9 % (IC95% 12,2-18,0), 19,8 % (IC95% 12,7-28,7) y 3,3 % (IC95% 1,4-6,9), respectivamente (23).
Se han descrito 18 especies de triatominos en Perú, de las cuales Triatoma infestans, T. carrioni, Panstrongylus herreri, P. chinai, P. geniculatus y R. ecuadoriensis están involucradas en la transmisión de T. cruzi (24-29). En este país, son escasos los estudios sobre caracterización molecular de los tripanosomas detectados en triatominos; por esta razón, el propósito del presente estudio fue detectar y tipificar las DTU de T. cruzi en las heces de siete especies de triatominos capturados en ocho departamentos localizados en diferentes regiones naturales de Perú. Se decidió por la detección y caracterización directa de los tripanosomas presentes en las heces para evitar la selección de cepas o DTU cuando los parásitos se llevan a medios de cultivo o se aíslan en animales de laboratorio (30).
Este es el primer estudio de caracterización a gran escala de T. cruzi en el intestino de vectores de importancia epidemiológica en Perú con el propósito de generar información básica que permita entender la dinámica de la transmisión vectorial de T. cruzi en esta región del continente.
Materiales y métodos
Material entomológico
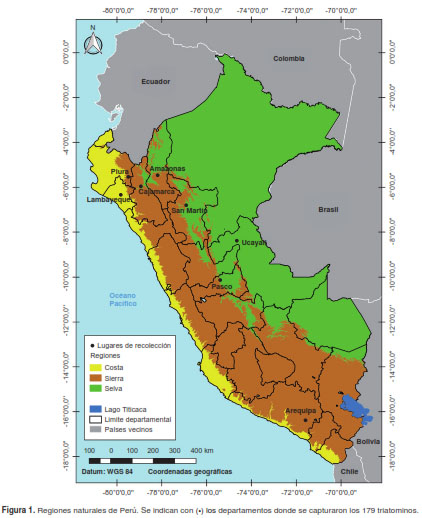
Las capturas de los triatominos se hicieron entre 1995 y 2014 en el interior de las viviendas de áreas urbanas y rurales de ocho departamentos de Perú (Amazonas, Arequipa, Cajamarca, Lambayeque, San Martín, Pasco, Piura y Ucayali), ubicadas en las regiones naturales de Costa, Sierra y Selva (figura 1).

La búsqueda de los triatominos se hizo entre las 08:30 y las 19:00 horas; el tiempo de recolección en cada vivienda fue de 15 a 20 minutos y, para ello, se emplearon linterna de mano, vaso recolector y pinzas de 20 cm de largo.
En grietas, agujeros y hendiduras de las paredes internas de los dormitorios y de la cocina, el come dor y el cuyero, se recolectaron ejemplares adultos vivos y muertos, así como huevos, ninfas y exuvias en el caso de P. herreri.
Los ejemplares vivos (adultos y ninfas) se colocaron en vasos recolectores rotulados, recubiertos con papel filtro plegable en su interior; los huevos se pusieron en otros vasos rotulados con una porción de algodón en su interior y, los ejemplares muertos, en frascos pequeños con etanol al 90 %. El material se llevó al Laboratorio de Entomología del Instituto Nacional de Salud y a la Sección de Entomología del Instituto de Medicina Tropical “Daniel A. Carrión” de la Facultad de Medicina de la Universidad Nacional Mayor de San Marcos. La identificación de las especies se confirmó mediante claves taxonómicas (25,31).
Detección molecular de las DTU de T. cruzi
A partir del contenido intestinal de cada triatomino, se extrajo el ADN utilizando el estuche comercial PureLink Genomic DNA minikit ® (Thermo Fisher Scientific, Waltham, MA, USA). Para confirmar la presencia de ADN de T. cruzi, se utilizó una PCR convencional con los iniciadores S35/S36, previamente documentados (32).
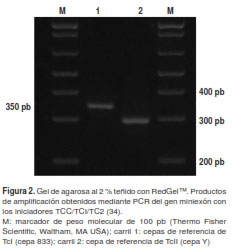
Las muestras positivas se procesaron para determinar el genotipo de T. cruzi mediante el protocolo reportado por Brisse, et al. (33): se hizo primero una PCR dirigida a la región del gen miniexón (34) para diferenciar la TcI (350 bp) del subgrupo de las TcII, TcV y TcVI (300 pb); la TcIII y la TcIV no generan ningún producto. Luego se hizo otra PCR para la región 24Sa rRNA, cuyos productos son la TcV (110 pb), la TcII y la TcVI (125 pb), la TcIII (110 pb) y la TcIV (120, 125 130 pb). Por último, para diferenciar los linajes TcII y TcVI, se hizo una tercera PCR para la región 18Sa rRNA: la TcII arrojó un producto de 165 pb y la TcVI no generó producto, aunque algunas cepas produjeron una banda tenue de 165 pb.
Se utilizaron seis cepas de referencia internacional correspondientes a las DTU para identificar los genotipos presentes en cada vector (TcI, cepa 833; TcII, cepa Y; TcIII, cepa MT3663; TcIV, cepa José Julio; TcV, cepa 967, y TcVI, cepa CL Brener) (figura 2 y figura 3). Los productos de amplificación se visualizaron en geles de agarosa al 2 % y en geles de poliacrilamida al 6 %, teñidos con Red Gel™ y nitrato de plata, respectivamente.
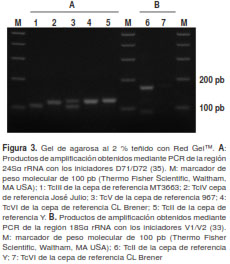
Resultados
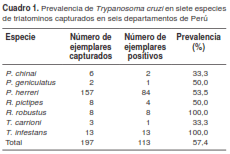
De los ocho departamentos donde se capturaron las siete especies de triatominos, solamente en seis se confirmó la presencia de T. cruzi (Amazonas, Arequipa, Cajamarca, Lambayeque, Pasco y Ucayali). En dichos departamentos, la prevalencia de T. cruzi fluctuó entre 33,3 y 100 % (cuadro 1).
Se procesaron 197 muestras de heces de triatominos utilizando los iniciadores S35/S36, y se detectaron 113 positivas para T. cruzi y cinco para T. rangeli (32). Trypanosoma rangeli sensu lato se detectó en el contenido intestinal de cuatro ejemplares de P. herreri capturados en el departamento de Amazonas y en un ejemplar de T. carrioni procedente del departamento de Cajamarca.
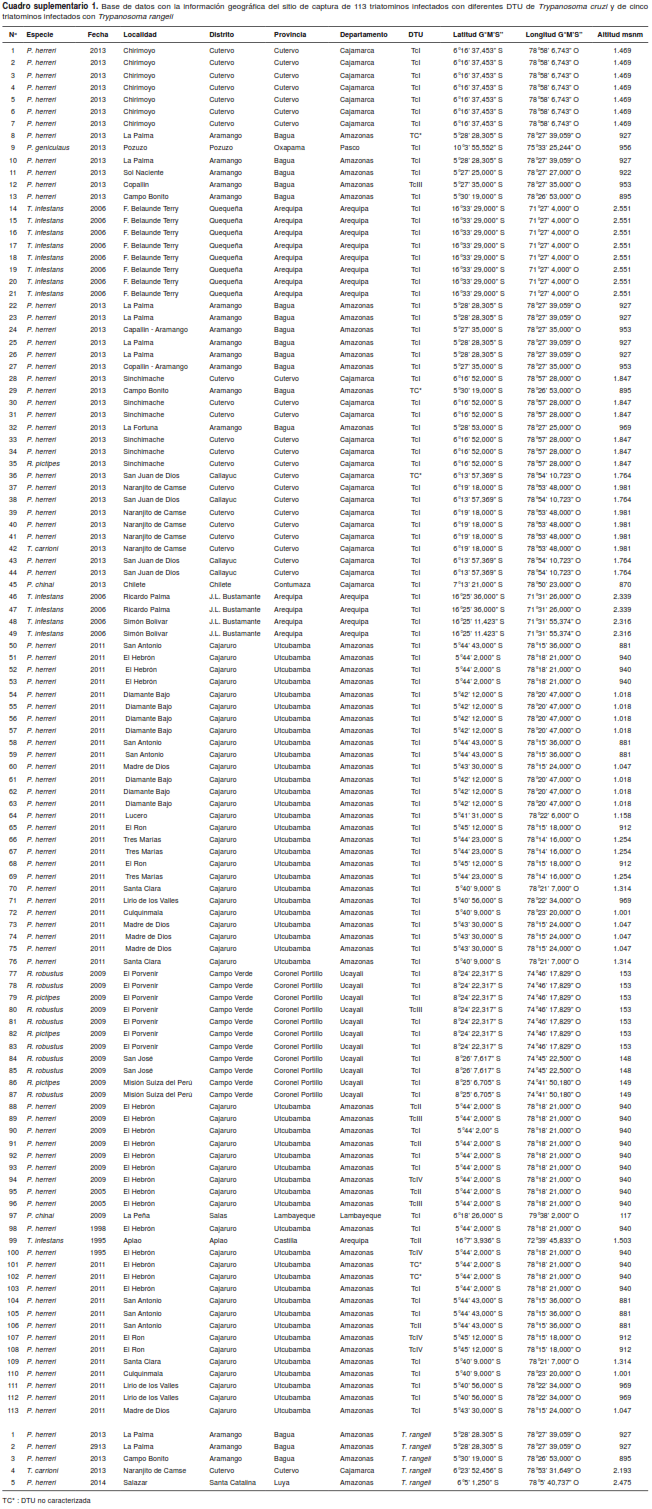
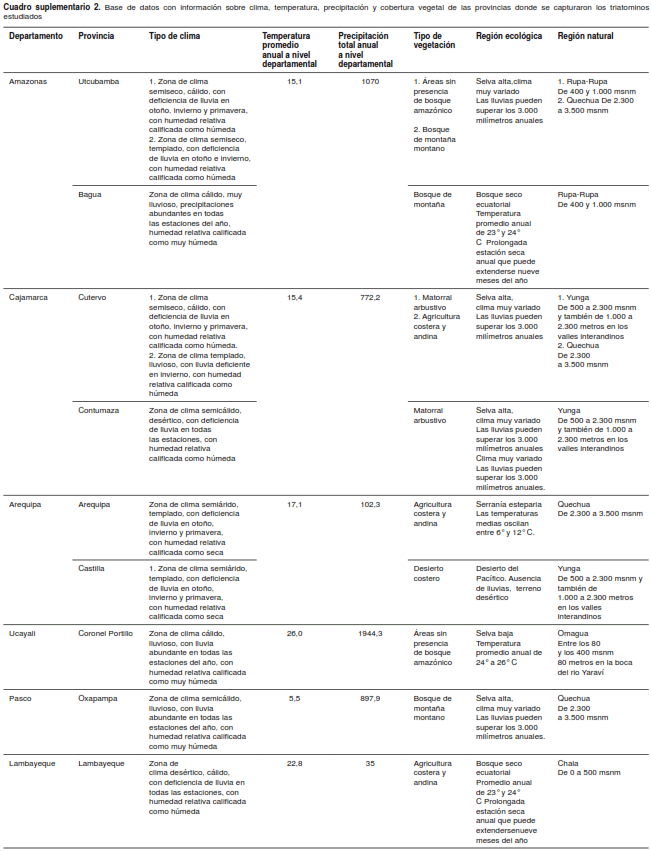
En el cuadro suplementario 1, disponible en https://doi.org/10.7705/biomedica.v34i2.3559, se presenta la información sobre especie, fecha de captura, localidad, distrito, provincia, departamento, DTU detectada, latitud, longitud y altura sobre el nivel del mar de los 113 triatominos positivos para T. cruzi y los cinco positivos para T. rangeli. Además, en el cuadro suplementario 2, disponible en https://doi.org/10.7705/biomedica.v34i2.3559, se presenta la información sobre el tipo de clima, temperatura promedio anual, precipitación anual departamental, tipo de vegetación, región ecológica y región natural de las provincias donde fueron capturados los triatominos, información obtenida del Servicio Nacional de Meteorología e Hidrología (SENAMHI), Lima-Perú, con respecto a los años 2003 a 2015, y del Ministerio del Ambiente, Dirección General de Evaluación, Valoración y Financiamiento del Patrimonio Natural: Lima-MINAM, para el 2015.
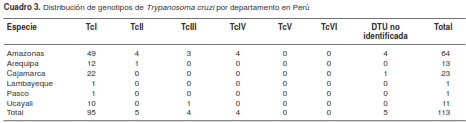
En 108 de las 113 muestras positivas para T. cruzi procesadas mediante PCR S35/S36 fue posible caracterizar las DTU utilizando amplificación de los genes miniexón, 24Sa rRNA y 18Sa rRNA. Las DTU caracterizadas presentaron la siguiente distribución: 95 de TcI, cinco deTcII, cuatro de TcIII, cuatro de TcIV y cinco cuyas DTU no fueron identificadas (cuadro 2). Se observó una predominancia de la TcI en las siete especies de triatominos analizadas (T. infestans, P. herreri, P. chinai, P. geniculatus, R. pictipes, R. robustus y T. carrioni), cuyos ejemplares se capturaron en los departamentos de Amazonas, Arequipa, Cajamarca, Lambayeque, Pasco y Ucayali (figura 4).
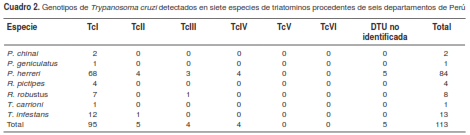
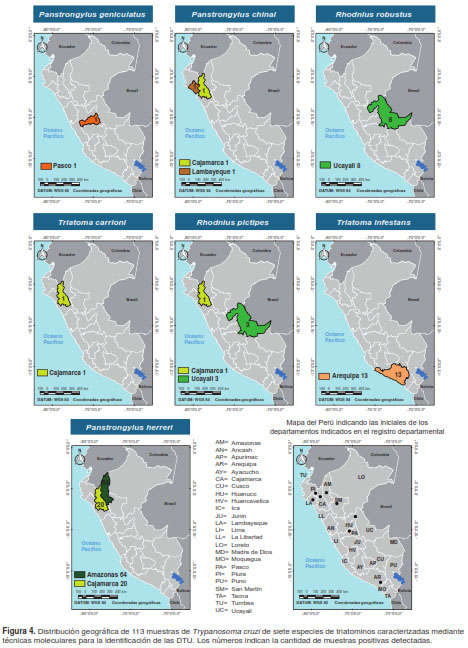
La mayoría de las muestras procedentes de Arequipa correspondió a la TcI (12/13 muestras) y, además, se detectó una muestra de TcII. Por otro lado, en Amazonas se detectó la mayoría de las DTU, así: 49 de TcI, cuatro de TcII, tres de TcIII y cuatro de TcIV. En Cajamarca y en Ucayali, se detectó la TcI en 22 y 10 muestras, respectivamente, y la TcIII se detectó en una muestra procedente de Ucayali (cuadro 3). No se detectaron genotipos correspondientes a TcV y TcVI.
Discusión
En el tubo digestivo de 118 (60 %) de los 197 triatominos procesados, se detectaron tripanosomas, de los cuales 113 correspondieron a T. cruzi y cinco a T. rangeli. En 108 de las muestras positivas para T. cruzi, se logró identificar la correspondiente DTU, y cinco se determinaron como T. cruzi sensu lato.
En estudios previos se ha reportado que T. rangeli se puede discriminar en dos grupos, KP1(+) y KP1(-), con base en la presencia o ausencia del minicírculo KP1 del ADN del cinetoplasto (kDNA) (35). Además, el análisis de la subunidad pequeña del ARN ribosómico (SSU-rRNA) ha permitido identificar cinco linajes (A, B, C, D, y E) en Centroamérica y Suramérica (36,37). En el caso de las cinco muestras de T. rangeli,estas se determinaron como T. rangeli sensu lato, sin identificar el linaje correspondiente debido a la escasa concentración del ADN del parásito. La presencia de T. rangeli se ha reportado previamente en Perú (28,38) y algunos aislamientos de R. ecuadoriensis en el departamento de Cajamarca se caracterizaron como T. rangeli KP1(-)/C (39,40). En este último estudio, los investigadores informaron que en las especies de Rhodnius de la cordillera de los Andes (R. pallescens, R. colombiensis, R. ecuadoriensis) circula el genotipo T. rangeli KP1(-)/C. En estudios los investigadores informaron que en las especies de Rhodnius de la cordillera de los Andes (R. pallescens, R. colombiensis, R. ecuadoriensis) circula el genotipo T. rangeli KP1(-)/C. En estudios recientes en las provincias de Loja y Manabí en Ecuador, se reportó la presencia de T. rangeli KP1(-)/C en el intestino de R. ecuadoriensis, P. howardi y P. rufotuberculatus (41), lo cual indica que es posible detectar la presencia de T. rangeli en el intestino de especies diferentes a hodnius, tal como se documentó previamente en P. megistus en Santa Catarina, Brasil (35).
En el presente estudio, se reportó la presencia de T. rangeli en el intestino de cuatro ejemplares de P. herreri recolectados en Amazonas y en un ejemplar de T. carrioni procedente de Cajamarca. La presencia de T. rangeli en el intestino de los triatominos es muy frecuente en varias regiones de Latinoamérica, aunque la invasión de las glándulas salivales solo ocurre en las especies del género Rhodnius (42). Los genotipos de T. rangeli detectados en el presente trabajo en el intestino de P. herreri y T. carrioni podrían corresponder a T. rangeli KP1(-) (C,D,E) o a T. rangeli KP1(+) (A,B). Para confirmar estas posibilidades será necesario caracterizar el parásito presente en el intestino de estos vectores con la finalidad de confirmar el tipo de linaje que circula en ellos.
En un estudio previo en Perú se abordó la detección de las DTU de T. cruzi en un número reducido de muestras (16 aislamientos), por lo cual sus resultados no fueron concluyentes (43). En el presente estudio, en 108 de las muestras positivas para T. cruzi se logró identificar la correspondiente DTU y cinco se determinaron como T. cruzi sensu lato. Se confirmó, asimismo, que la DTU TcI (95/108=88%) predominó en las especies de los triatominos recolectados en los seis departamentos de Perú (cuadro 3). Esta situación es similar en los países centroamericanos y en los países de la región norte de Suramérica, en donde predomina T. cruzi I (TcI) sobre T. cruzi II (TcII), T. cruzi III (TcIII) y T. cruzi IV (TcIV) (17,44-47).
En un estudio en Venezuela, el 94,1 % de 778 aislamientos correspondió a la TcI, mientras que la TcIV y la TcIII se detectaron en 3,1 y 2,8 % de ellos, respectivamente. En aislamientos humanos se detectaron la TcI y la TcIV en 79 y 21 % de ellos, respectivamente, pero no se detectó la TcIII, en tanto que en los aislamientos de triatominos se detectaron tres linajes (TcI, TcIV y TcIII) (46). En Bolivia, 99,1 % de 333 muestras del contenido intestinal de T. infestans correspondió a la TcI (45), y en Chile este fue el linaje más prevalente en muestras del contenido intestinal de T. infestans (97,6 %), detectado en 100 % de los ejemplares recolectados en el interior de las viviendas y en 93,3 % de los ejemplares selváticos (47). La predominancia de la TcI podría estar asociada a una mayor capacidad de los vectores para transmitirla, similar a lo que se ha demostrado para T. infestans, especie que tiene una mayor capacidad de transmitir la TcI que la TcV y la TcII (48); asimismo, el linaje TcI tiene mayor eficiencia para infectar huéspedes (49).
Es importante señalar que en este estudio se recolectaron en algunas localidades de las provincias de Bagua y Utcubamba, departamento de Amazonas, 101 ejemplares de P. herreri en el interior de las viviendas, de los cuales 64 fueron positivos para T. cruzi (64/101; 63,4 %) y cuatro para T. rangeli (4/101; 4 %). Esta región (Selva) presentó la mayor variabilidad de las DTU de T. cruzi: TcI (49/64; 76,6 %), TcII (4/64; 6,25 %), TcIII (3/64; 4,7 %), TcIV (4/64; 6,25 %) y algunas no determinadas (5/64; 7,8 %). En estas mismas localidades de las dos provincias (Amazonas), se capturaron en años anteriores ejemplares de P. herreri en el peridomicilio (27).
La variabilidad de las DTU en el departamento de Amazonas podría explicarse por las características y la ubicación de las viviendas de las diversas localidades; por ejemplo, las viviendas de la localidad de El Hebrón, distrito de Cajaruro, provincia de Utcubamba (figura 5), se encontraban cerca de la floresta, y 90 % de ellas estaban construidas con material favorable para la colonización de triatominos (paredes de adobe, techo de calamina, sin piso asfaltado y paredes externas e internas sin estucar). Asimismo, en el interior de las viviendas había cuyes (Cavia porcellus) y en los alrededores con frecuencia se observaban perros y gatos, así como corrales para aves y cerdos. Precisamente en esta localidad se detectaron cinco DTU de TcI, tres de TcII, dos de TcIII y dos de TcIV, lo cual indica la gran variabilidad de las DTU de T. cruzi en esta región. Sin duda, en la floresta de El Hebrón los animales silvestres visitan durante la noche los alrededores de las viviendas con la finalidad de alimentarse de frutas y ubicar sus presas. En los alrededores de las viviendas se ha observado la presencia de P. herreri y, por lo tanto, existe la probabilidad de que estos triatominos se alimenten tanto de animales silvestres como de los domésticos.

En el presente estudio, se capturaron ejemplares de P. herreri en el interior de las viviendas, pero en estudios previos se han capturado ejemplares de esta especie también en el peridomicilio (27). Estas observaciones sugieren la coexistencia en esta zona de los ciclos de transmisión doméstica, peridoméstica y silvestre; no obstante, es necesario verificar esta hipótesis en futuros proyectos. Los resultados de este estudio en la selva amazónica peruana coinciden con los de otros estudios de caracterización de T. cruzi aislado de vectores y reservorios de la Amazonia brasilera, donde se ha documentado la presencia de T. cruzi I, T. cruzi III y T. cruzi IV (50,51).
En el departamento de Ucayali (Selva), se recolectaron en el interior de las viviendas ocho ejemplares de R. robustus y tres de R. pictipes. Estas dos especies son estrictamente silvestres, por lo que este hecho constituye una evidencia de su comportamiento intrusivo de las viviendas humanas. Las viviendas de las localidades de El Porvenir-Cashibo y San José, distrito de Campo Verde, provincia de Coronel Portillo, se ubicaban en la floresta de bosque secundario (a unos 5 a 10 m), poblado de palmeras, árboles y arbustos (figura 6), y sus paredes eran de madera, el techo de hojas de palmeras y el piso de tierra. Cada vivienda constaba de dos a tres ambientes; en los alrededores se criaban aves de corral y cerdos, y en el interior de las viviendas, cuyes. Sobre los árboles circundantes ocasionalmente se observó la presencia de animales silvestres (monos tití).

Por otro lado, algunas viviendas de la localidad de Misión Suiza del Perú, distrito de Campo Verde, provincia de Coronel Portillo (Amazonas), se ubicaban en las cercanías o a 100 m del bosque secundario, caracterizado por la presencia de palmeras, árboles y unos pocos arbustos. Las viviendas allí también eran de materiales favorables para los triatominos (paredes de madera, la mayoría con techo de hojas de palmeras y ramas de árboles, y pocas con techo de eternit o calamina) (figura 7), y se acostumbraba la cría de aves de corral. Por las características de las viviendas y por las especies de triatominos capturados en esas localidades, se puede concluir que no se daba el ciclo de transmisión doméstica o peridoméstica, y que el ciclo silvestre llegaba hasta las viviendas humanas con la intrusión de R. robustus y R. pictipes. En la localidad de El Porvenir-Cashibo, se detectó la presencia de TcI en siete ejemplares de R. robustus y en tres de R. pictipes,y en uno de R. robustus,la de TcIII.

En el departamento de Cajamarca (Sierra), se recolectaron 54 ejemplares de P. herreri, de los cuales 20 (20/54; 37,04 %) resultaron positivos para T. cruzi, y en 19 (19/20; 95 %) se identificó la TcI, en tanto que en un ejemplar (1/20; 5 %) no se logró identificar la DTU. Estos resultados sugieren que en las cuatro localidades de los dos distritos de la provincia de Cutervo (Cajamarca), de donde procedían los triatominos, la transmisión de T. cruzi era doméstica. Panstrongylus herreri es un triatomino doméstico, pues todo su ciclo biológico lo desarrolla en el interior de las viviendas (27), pero también hay presencia de animales domésticos, como cuyes, perros y gatos, que constantemente se desplazan de un ambiente a otro de la vivienda donde se encuentran los estadios inmaduros y adultos de P. herreri que así tienen la oportunidad de alimentarse de dichos animales.
En la misma provincia de Cutervo (Cajamarca), pero en localidades pertenecientes a distritos diferentes, en numerosas oportunidades se han capturado ejemplares de P. herreri en ambientes domésticos, peridomésticos y silvestres, aunque en estos dos últimos ambientes el número de las capturas fue menor que en el interior de las viviendas; asimismo, se ha informado la presencia de tripanosomas en las heces de P. herreri (Sara Castro y Deysi LunaCaipo, comunicación personal, 15 de marzo de 2016). Por ello, es necesario y prioritario evaluar los triatominos existentes en los ambientes peridomésticos y silvestres para determinar con exactitud el tipo de transmisión del parásito en las áreas de estudio y, a la vez, determinar las DTU circulantes.
Por las limitaciones del muestreo, en este estudio solo se examinó un ejemplar de P. chinai proveniente de la costa norte, de la localidad de La Peña, distrito de Salas, provincia de Lambayeque (Lambayeque), y en él se detectó T. cruzi I. Las referencias bibliográficas indican que se han observado T. cruzi mediante microscopía en ejemplares de P. chinai y R. ecuadoriensis capturados en Tumbes, Piura, Lambayeque, La Libertad y Ancash en la costa norte (52-55). Se han encontrado varios ejemplares de R. ecuadoriensis procedentes de la costa de Piura y de La Libertad infectados naturalmente con T. rangeli (28,38,56) y, en estudios posteriores con cepas de T. rangeli aisladas de ejemplares procedentes del departamento de La Libertad, se encontró el genotipo T. rangeli KP1 (-)/C (39,40).
En la costa sur (Ica, Arequipa, Moquegua y Tacna), T. infestans es el único triatomino presente. En algunas localidades de la costa de las provincias de Ica, Palpa y Nazca, departamento de Ica, se recolectan con frecuencia ejemplares de T. infestans infectados naturalmente con T. cruzi (57); sin embargo, en los estudios no se menciona la caracterización de las DTU.
Entre 2012 y 2014, se capturaron ejemplares de T. infestans en el domicilio y el peridomicilio, en localidades de la costa de la provincia de Caraveli, departamento de Arequipa; sin embargo, no se ha detectado T. cruzi en estos especímenes mediante microscopía (Nancy Ruelas-Llerena, comunicación personal, marzo de 2016, y Fernando Málaga Chávez, comunicación personal, 8 de abril de 2016). Esto mismo sucede con los ejemplares de T. infestans de la costa de los departamentos de Moquegua y Tacna (Miriam Guzmán Loayza y Javier Villanueva Roque, comunicación personal, 10 de abril de 2016).
En estudios recientes sobre la variabilidad de T. cruzi I en Colombia, se detectaron dos genotipos claramente diferenciados denominados T. cruzi I doméstico (asociado a R. prolixus doméstico) y T. cruzi I selvático (asociado a ejemplares silvestres de R. pallescens y R. colombiensis) (58). Esta misma diferenciación molecular se observó en 101 aislamientos de T. cruzi I en ese país, pues en 70 % de ellos se encontró la TcI selvática y, en 30 %, la TcI doméstica (59). El genotipo selvático se ha asociado con los casos agudos de transmisión vectorial en áreas selváticas y en casos de transmisión oral por contaminación de alimentos (58). Aunque en Perú se documentó un primer caso de enfermedad aguda de Chagas por transmisión vectorial de T. cruzi, en el cual se implicó P. geniculatus (29), es muy importante confirmar si en el país están presentes estos mismos genotipos de T. cruzi I, denominados T. cruzi I doméstico y T. cruzi I selvático (58), lo cual permitirá obtener información sobre la epidemiología de T. cruzi en Perú y confirmar si eventuales casos de enfermedad de Chagas aguda pudieran estar asociados a los genotipos silvestres de T. cruzi I, como ya se reportó en Colombia (58).
En este trabajo se documentó la presencia de T. cruzi en vectores procedentes de zonas endémicas de Perú, entre ellos, P. herreri, T. infestans, P. chinai, P. geniculatus, R. pictipes, R. robustus y T. carrioni. Las DTU presentes en el intestino de estos vectores de importancia epidemiológica se caracterizaron y se determinó que la TcI fue la más abundante, y que la TcII, la TcIII y la TcIV fueron menos frecuentes. La información presentada contribuye a una mejor comprensión de la dinámica de transmisión vectorial de T. cruzi en Perú.
Al Consejo Nacional de Ciencia, Tecnología e Innovación Tecnológica de Perú (CONCYTEC), por la subvención económica proporcionada a Carlos Padilla para su pasantía en el Laboratorio de Investigaciones en Parasitología Tropical de la Universidad del Tolima, Ibagué, Colombia, donde se hicieron los estudios moleculares para la tipificación de las DTU de T. cruzi. Al Departamento Administrativo de Ciencia, Tecnología e Innovación de Colombia (Colciencias) y al Fondo de Investigaciones de la Universidad del Tolima en ese país, por la financiación parcial de la tipificación de T. cruzi.
Los autores declaramos que no existen conflictos de intereses.
El presente trabajo fue financiado por el Consejo Nacional de Ciencia, Tecnología e Innovación
Tecnológica de Perú (CONCYTEC), por el Departamento Administrativo de Ciencia, Tecnología e Innovación, Colciencias, de Colombia (contrato 110551929038), y por el Fondo de Investigaciones de la Universidad del Tolima de ese país.
Correspondencia:
Abraham G. Cáceres, Laboratorio de Entomología, Instituto Nacional de Salud, Calle Capac Yupanqui N° 1400, Jesús María, Lima, Perú
Teléfono: (051) 748 1111, anexo 1548
acaceres31@hotmail.com; acaceres31@hotmail.com
Referencias
1. World Health Organization. Chagas disease (American trypanosomiasis). Fecha de consulta: 10 de enero de 2015. Disponible en: http://www.who.int/mediacentre/factsheets/fs340/en/.
2. Rueda K, Trujillo JE, Carranza JC, Vallejo GA. Transmisión oral de Trypanosoma cruzi: una nueva situación epidemiológica de la enfermedad de Chagas en Colombia y otros países suramericanos. Biomédica. 2014;34:631-41. https://doi.org/10.7705/biomedica.v34i4.2204
3. Zingales B, Andrade SG, Briones MR, Campbell DA, Chiari E, Fernandes O, et al. A new consensus for Trypanosoma cruzi intraspecific nomenclature: Second revision meeting recommendsTcI to TcVI. Mem Inst Oswaldo Cruz. 2009;104:1051-4. https://doi.org/10.1590/S007402762009000700021
4. Zingales B, Miles MA, Campbell DA, Tibayrenc M, Macedo AM, Teixeira MM, et al. The revised Trypanosoma cruzi subspecific nomenclature: Rationale, epidemiological relevance and research applications. Infect Genet Evol. 2012;12:240-53. https://doi.org/10.1016/j.meegid.2011.12.009
5. Marcili A, Lima L, Cavazzana MJ, Junqueira AC, Veludo HH, Maia da Silva F, et al. A new genotype of Trypanosoma cruzi associated with bats evidenced by phylogenetic analyses using SSU rDNA, cytochrome b and histone H2B genes and genotyping based on ITS1 rDNA. Parasitology. 2009;136:641-55. https://doi.org/10.1017/S0031182009005861
6. Pinto CM, Kalko EK, Cottontail I, Wellinghausen N, Cottontail VM. TcBat a bat-exclusive lineage of Trypanosoma cruzi in the Panamá Canal Zone, with comments on its classification and the use of the 18S rRNA gene for lineage identification. Infect Genet Evol. 2012;12:1328-32. https://doi.org/10.1016/j.meegid.2012.04.013
7. Ramírez JD, Tapia-Calle G, Muñoz-Cruz G, Poveda C, Rendón LM, Hincapié E, et al. Trypanosome species in neotropical bats: Biological, evolutionary and epidemiological implications. Infect Genet Evol. 2014;22:250-6. https://doi.org/10.1016/j.meegid.2013.06.022
8. Lima L, Espinosa-álvarez O, Ortiz PA, Trejo-Varón JA, Carranza JC, Pinto CM, et al. Genetic diversity of Trypanosoma cruzi in bats, and multilocus phylogenetic and phylogeographical analyses supporting Tcbat as an independent DTU (discrete typing unit). Acta Trop. 2015;151:166-77. https://doi.org/10.1016/j.actatropica.2015.07.015
9. Herrera C, Bargues MD, Fajardo A, Montilla M, Triana O, Vallejo GA, et al. Identifying four Trypanosoma cruzi I isolate haplotypes from different geographic regions of Colombia. Infect Genet Evol. 2007;7:535-9. https://doi.org/10.1016/j.meegid.2006.12.003
10. Llewellyn MS, Miles MA, Carrasco HJ, Lewis MD, Yeo M, Vargas J, et al. Genome-scale multilocus microsatellite typing of Trypanosoma cruzi discrete typing unit I reveals phylogeographic structure and specific genotypes linked to human infection. PLoS Pathog. 2009;5:e1000410. https://doi.org/10.1371/journal.ppat.1000410
11. Ramírez JD, Guhl F, Rendón LM, Rosas F, Marín-Neto JA, Morillo CA. Chagas cardiomyopathy manifestations and Trypanosoma cruzi genotypes circulating in chronic chagasic patients. PLoS Negl Trop Dis. 2010;4:e899. https://doi.org/10.1371/journal.pntd.0000899
12. Mantilla JC, Zafra GA, Macedo AM, González CI. Mixed infection of Trypanosoma cruzi I and II in a Colombian cardiomyopathic patient. Hum Pathol. 2010;41:610-3. https://doi.org/10.1016/j.humpath.2009.11.005
13. Rassi A Jr, Rassi A, Marcondes de Rezende J. American trypanosomiasis (Chagas disease). Infect Dis Clin North Am. 2012;26:275-91. https://doi.org/10.1016/j.idc.2012.03.002
14. Monje-Rumi MM, Pérez-Brandán C, Ragone PG, Tomasini N, Lauthier JJ, Alberti-D’Amato AM, et al. Trypanosoma cruzi diversity in the Gran Chaco: Mixed infections and differential host distribution of TcV and TcVI. Infect Genet Evol. 2015;29:53-9. https://doi.org/10.1016/j.meegid.2014.11.001
15. Cura CI, Mejía-Jaramillo AM, Duffy T, Burgos JM, Rodriguero M, Cardinal MV, et al. Trypanosoma cruzi I genotypes in different geographical regions and transmission cycles based on a microsatellite motif of the intergenic spacer of spliced-leader genes. Int J Parasitol. 2010;40:1599607. https://doi.org/10.1016/j.ijpara.2010.06.006
16. Yeo M, Acosta N, Llewellyn M, Sánchez H, Adamson S, Miles GA, et al. Origins of Chagas disease: Didelphis species are natural hosts of Trypanosoma cruzi I and armadillos hosts of Trypanosoma cruzi II, including hybrids. Int J Parasitol. 2005;35:225-33. https://doi.org/10.1016/j.ijpara.2004.10.024
17. Vallejo GA, Guhl F, Carranza JC, Herrera C, Urrea DA, Falla A, et al. Trypanosoma cruzi population variability in Colombia: Possible co-evolution in different vector species. Rev Soc Bras Med Trop. 2009;42(Supl.II):27-34.
18. Wiesenberger SJ, Barnabé C, Campbell DA, Sturm NR. Two hybridization events define the population structure of Trypanosoma cruzi. Genetics. 2005;171:527-43. https://doi.org/10.1534/genetics.104.038745
19. García ES, Genta FA, Azambuja P, Schaub GA. Interactions between intestinal compounds of triatomines and Trypanosoma cruzi. Trends Parasitol. 2010;20:499-505. https://doi.org/10.1016/j.pt.2010.07.003
20. Vallejo GA, Guhl F, Schaub GA. Triatominae-Trypanosoma cruzi-T. rangeli: Vector-parasite interactions. Acta Trop. 2009;110:137-47. https://doi.org/10.1016/j.actatropica.2008.10.001
21. Castro DP, Moraes CS, González MS, Ratcliffe NA, Azambuja P, García ES. Trypanosoma cruzi immune response modulation decreases microbiota in Rhodnius prolixus gut and is crucial for parasite survival and development. PLoS One. 2012;7:e36591. https://doi.org/10.1371/journal.pone.0036591
22. Centro Nacional de Epidemiología, Prevención y Control de Enfermedades Dr. Daniel Olaechea. Boletín epidemiológico. Semana epidemiológica 52 de 2014. Fecha de consulta: 15 de enero de 2015. Disponible en: http://www.dge.gob.pe/portal/docs/vigilancia/boletines/2014/52.pdf
23. Alroy KA, Huang C, Gilman RH, Quispe-Machaca VR, Marks MA, Ancca-Juárez J, et al. Prevalence and transmission of Trypanosoma cruzi in people of rural communities of the high jungle of northern Perú. PLoS Negl Trop Dis. 2015;9:e0003779. https://doi.org/10.1371/journal.pntd.0003779
24. Chávez J. Contribución al estudio de los triatominos del Perú: distribución geográfica, nomenclatura y notas taxonómicas.An Fac Med Lima. 2006;67:65-76.
25. Elliot A, Cáceres I, Guillén Z, Nakashima I. Identificación de los chinches triatominos (Hemiptera, Reduviidae) conocidos del Perú. Rev Per Ent. 1988;31:18-20.
26. Guillén Z, Cáceres I, Elliot A, Ramírez J. Triatominos del norte peruano y su importancia como vectores de Trypanosoma spp. Rev Per Ent. 1989;31:25-30.
27. Cáceres GA, Troyes L, Gonzáles-Pérez A, Llontop E, Bonilla C, Murias E, et al. Enfermedad de Chagas en la región nororiental del Perú. I. Triatominos (Hemiptera, Reduviidae) presentes en Cajamarca y Amazonas. Rev Peru Med Exp Salud Pública. 2002;19:17-23.
28. Cuba CA, Abad-Franch F, Roldán J, Vargas F, Pollack L, Miles MA. The triatomines of northern Perú, with emphasis on the ecology and infection by trypanosomes of Rhodnius ecuadoriensis (Triatominae). Mem Inst Oswaldo Cruz. 2002;97:175-183. https://doi.org/10.1590/S007402762002000200005
29. Vega S, Mendoza A, Cabrera R, Cáceres GA, Campos E, Ancca J, et al. Primer caso de enfermedad de Chagas aguda en la Selva Central del Perú: investigación de colaterales, vectores y reservorios. Rev Peru Med Exp Salud Pública. 2006;23:288-92.
30. Macedo AM, Oliveira RP, Pena SD. Chagas disease: Role of parasite genetic variation in pathogenesis. Expert Rev Mol Med. 2002;4:1-16. https://doi.org/10.1017/S1462399402004118
31. Lent H, Wygodzinsky P. Revision of the Triatominae (Hemiptera, Reduviidae), and their significance as vectors of Chagas disease. Bull Am Mus Nat His.1979;163:123-520.
32. Vallejo GA, Guhl F, Chiari E, Macedo AM. Speciesspecific detection of Trypanosoma cruzi and Trypanosoma rangeli in vector and mammalian hosts by polymerase chain reaction amplification of kinetoplast minicircle DNA. Acta Trop. 1999;72:203-12. https://doi.org/10.1016/S0001706X(98)00085-0
33. Brisse S, Verhoef J, Tibayrenc M. Characterisation of large and small subunit rRNA and mini-exon genes further supports the distinction of six Trypanosoma cruzi lineages. Int J Parasitol. 2001;31:1218-26. https://doi.org/10.1016/S0020-7519(01)00238-7
34. Souto RP, Fernandes O, Macedo AM, Campbell DA, Zingales B. DNA markers define two major phylogenetic lineages of Trypanosoma cruzi. Mol Biochem Parasitol. 1996;83:141-52. https://doi.org/10.1016/S0166-6851(96)02755-7
35. Vallejo GA, Guhl F, Carranza JC, Lozano LE, Sánchez JL, Jaramillo JC, et al. KDNA markers define two major Trypanosoma rangeli lineages in Latin-America. Acta Tropica. 2002;81:77-82. https://doi.org/10.1016/S0001-706X (01)00186-3
36. Maia Da Silva F, Junqueira AC, Campaner M, Rodrigues AC, Crisante G, Ramirez LE, et al. Comparative phylogeography of Trypanosoma rangeli and Rhodnius (Hemiptera: Reduviidae) supports a long coexistence of parasite lineages and their sympatric vectors. Mol Ecol. 2007;16:3361-73. https://doi.org/10.1111/j.1365-294X.2007.03371.x
37. Maia da Silva F, Marcili A, Lima L, Cavazzana M Jr, Ortiz PA, Campaner M, et al. Trypanosoma rangeli isolates of bats from Central Brazil: Genotyping and phylogenetic analysis enable description of a new lineage using spliced-leader gene sequences. Acta Trop. 2009;109:199-207. https://doi.org/10.1016/j.actatropica.2008.11.005
38. Cuba CA, Vallejo GA, Gurgel-Gonçalves R. Triatomines (Hemiptera, Reduviidae) prevalent in the northwest of Perú: Species with epidemiological vectorial capacity. Parasitol Latinoam. 2007;62:154-64. https://doi.org/10.4067/S071777122007000200009
39. Urrea DA, Carranza JC, Cuba-Cuba CA, GurgelGonçalves R, Guhl F, Schofield CJ, et al. Molecular characterisation of Trypanosoma rangeli strains isolated from Rhodnius ecuadoriensis in Perú, R. colombiensis in Colombia and R. pallescens in Panamá, supports a coevolutionary association between parasites and vectors. Infect Genet Evol. 2005;5:123-9. https://doi.org/10.1016/j.meegid.2004.07.005
40. Urrea DA, Herrera CP, Falla A, Carranza JC, Cuba-Cuba C, Triana-Chávez O, et al. Sequence analysis of the splicedleader intergenic region (SL-IR) and random amplified polymorphic DNA (RAPD) of Trypanosoma rangeli strains isolated from Rhodnius ecuadoriensis, R. colombiensis, R. pallescens and R. prolixus suggests a degree of co-evolution between parasites and vectors. Acta Trop. 2011;120:59-66. https://doi.org/10.1016/j.actatropica.2011.05.016
41. Ocaña-Mayorga S, Aguirre-Villacis F, Pinto C, Vallejo GA, Grijalva M. High prevalence and low genetic diversity of Trypanosoma rangeli in sylvatic R. ecuadoriensis and mammal hosts. Vector Borne Zoonotic Dis. 2015;15:732-42. https://doi.org/10.1089/vbz.2015.1794
42. Vallejo GA, Suárez Y, Olaya JL, Gutiérrez SA, Carranza JC. Trypanosoma rangeli: un protozoo infectivo y no patógeno para el humano que contribuye al entendimiento de la transmisión vectorial y la infección por Trypanosoma cruzi, agente causal de la enfermedad de Chagas. Rev Acad Colomb Cienc Ex Fis Nat. 2015;39:111-22.
43. Ancca J, Vega S, Pinto J, Naquira C. Caracterización genética de Trypanosoma cruzi aislados de triatominos de áreas endémicas a la enfermedad de Chagas en el Perú. Rev Ibero Latinoam Parasitol. 2009;1:8-14.
44. Guhl F, Ramírez JD. Retrospective molecular integrated epidemiology of Chagas disease in Colombia. Infect Genet Evol. 2013;20:148-54. https://doi.org/10.1016/j.meegid.2013.08.028
45. Brenière SF, Aliaga C, Waleckx E, Buitrago R, Salas R, Barnabé C, et al. Genetic characterization of Trypanosoma cruzi DTUs in wild Triatoma infestans from Bolivia: Predominance of TcI. PLoS Negl Trop Dis. 2012;6:e1650. https://doi.org/10.1371/journal.pntd.0001650
46. Carrasco HJ, Segovia M, Llewellyn MS, Morocoima A, Urdaneta-Morales S, Martínez C, et al. Geographical distribution of Trypanosoma cruzi genotypes in Venezuela. PLoS Negl Trop Dis. 2012;6:e1707. https://doi.org/10.1371/journal.pntd.0001707
47. Bacigalupo A, Segovia V, García A, Botto-Mahan C, Ortiz S, Solari A, et al. Differential pattern of infection of sylvatic nymphs and domiciliary adults of Triatoma infestans with Trypanosoma cruzi genotypes in Chile. Am J Trop Med Hyg. 2012;87:473-80. https://doi.org/10.4269/ajtmh.2012.11-0237
48. de Lana M, da Silveira Pinto A, Barnabé C, Quesney V, Noël S, Tibayrenc M. Trypanosoma cruzi: Compared vectorial transmissibility of three major clonal genotypes by Triatoma infestans. Exp Parasitol. 1998;90:20-5. https://doi.org/10.1006/expr.1998.4304
49. Galuppo S, Bacigalupo A, García A, Ortiz S, Coronado X, Cattan PE, et al. Predominance of Trypanosoma cruzi genotypes in two reservoirs infected by sylvatic Triatoma infestans of an endemic area of Chile. Acta Trop. 2009;111:90-3. https://doi.org/10.1016/j.actatropica.2009.02.010
50. Monteiro WM, Magalhães LK, Santana Filho FS, Borborema M, Silveira H, Barbosa MD. Trypanosoma cruzi TcIII/Z3 genotype as agent of an outbreak of Chagas disease in the Brazilian Western Amazonia. Trop Med Int Health. 2010;15:1049-51. https://doi.org/10.1111/j.1365-3156.2010.02577
51. Monteiro WM, Teston AP, Gruendling AP, dos Reis D, Gomes ML, de Araújo SM, et al. Trypanosoma cruzi I and IV stocks from Brazilian Amazon are divergent in terms of biological and medical properties in mice. PLoS Negl Trop Dis. 2013.7: e2069. https://doi.org/10.1371/journal.pntd.0002069
52. Calderón-Falero G. Chinches triatominos (Hemiptera: Reduviidae) de la región Grau, Perú. Rev Per Ent. 1996;38:19-22.
53. Guillén Z, Cáceres I, Elliot A. Los triatominos (Hemiptera, Reduviide) de la zona Centro del Perú. Rev Per Med Trop UNMSM. 1992;6:89-91.
54. Jara CA, Escalante H, Roldán J, Díaz-Lima E. Distribución y frecuencia de infección por Trypanosoma cruzi de triatominos y Ovis aries en el Valle de Chamán, La Libertad, Perú. Sciendo. 1998;1:23-31.
55. Llanos B. Hallazgo en el Perú de Rhodnius ecuadoriensis Lent & León, 1958, naturalmente infectado por Trypanosoma cruzi. Nota preliminar. Arch Per Pat Clin. 1961;15:133-40.
56. Cuba-Cuba AC, Vargas F, Roldán J, Ampuero C. Domestic Rhodnius ecuadoriensis (Hemiptera, Reduviidad) infestation in Northern Perú: A comparative trial of detection methods during a six-month follow-up. Rev Inst Med Trop Sao Paulo. 2003;45:85-90. https://doi.org/10.1590/S003646652003000200006
57. Solís H, De Carvalho E, Ferreira C, Casanova C, Huamán A, Mendoza V. Contribución al estudio de la epidemiología de la enfermedad de Chagas en tres localidades de la zona Sur del Perú. An Fac Med Lima. 2003;64:223-7.
58. Villa LM, Guhl F, Zabala D, Ramírez JD, Urrea DA, Hernández DC, et al. The identification of two Trypanosoma cruzi I genotypes from domestic and sylvatic transmission cycles in Colombia based on a single polymerase chain reaction amplification of the spliced-leader intergenic region. Mem Inst Oswaldo Cruz. 2013;108:932-35. https://doi.org/10.1590/0074-0276130201
59. León CM, Hernández C, Montilla M, Ramírez JD. Retrospective distribution of Trypanosoma cruzi I genotypes in Colombia. Mem Inst Oswaldo Cruz. 2015;110:387-93. https://doi.org/10.1590/0074-02760140402