
doi: http://dx.doi.org/10.7705/biomedica.v33i2.774
ARTÍCULO ORIGINAL
Beatriz Millán 1 , David Castro 1 , María Araque 1 , Bárbara Ghiglione 2 , Gabriel Gutkind 2
1 Laboratorio de Microbiología Molecular, Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Mérida, Venezuela
2 Cátedra de Microbiología, Facultad de Farmacia y Bioquímica, Universidad de Buenos Aires, Buenos Aires, Argentina
Institución donde se llevó a cabo el trabajo: Laboratorio de Microbiología Molecular, Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Mérida, Venezuela.
Recibido: 13/07/12; aceptado:20/12/12
Introducción. Las secuencias de inserción tales como IS CR1 promueven la captura, transposición y expresión de los genes bla CTX-M, facilitando, de esta manera, su diseminación rápida en la población bacteriana.
Objetivo. Se determinó la presencia del elemento IS CR1 y su asociación con genes bla CTX-M-1 y bla CTX-M-2 en plásmidos de diferentes grupos de incompatibilidad en Klebsiella pneumoniae de origen hospitalario.
Materiales y métodos. Se aislaron tres cepas de K. pneumoniae con sensibilidad disminuida a cefalosporinas de amplio espectro, de neonatos con septicemia hospitalaria. La presencia de β -lactamasas de espectro expandido (BLEE) fue determinada fenotípicamente. Los plásmidos se aislaron y clasificaron según grupos de incompatibilidad por tipificación del replicón por reacción en cadena de la polimerasa (PCR). Los genes bla BLEE y su asociación a IS CR1 se determinaron por PCR y secuenciación directa, usando varios juegos de iniciadores.Resultados. Todas las cepas demostraron un perfil fenotípico indicativo de producción de BLEE, transferibles por conjugación. Los ensayos de PCR para para cefotaximasas (CTX-M) y el análisis de la secuenciación, revelaron que las cepas portaban genes bla CTX-M-1 y bla CTX-M-2. Estos genes se encontraron en plásmidos conjugados de 150 kb, aproximadamente, relacionados con los grupos IncN e IncFIIA, respectivamente. IS CR1 se encontró ‘aguas arriba' ( upstream ) y asociado con los genes bla CTX-M-1 y bla CTX-M-2.
Conclusión. Este es el primer reporte realizado en Venezuela donde la presencia de IS CR1 está estrechamente asociada con la movilización de los genes bla CTX-M-1 y bla CTX-M-2 en plásmidos conjugativos IncN y IncFIIA en cepas de K. pneumoniae que circulan en una Unidad de Alto Riesgo Neonatal.
Palabras clave: Klebsiella pneumoniae , plásmidos, infección hospitalaria, beta-lactamasas, cefotaxima, elementos transponibles de ADN.
doi: http://dx.doi.org/10.7705/biomedica.v33i2.774
IS CR1 associated with bla CTX-M-1 y bla CTX-M-2 genes in IncN and IncFIIA plasmids isolated from Klebsiella pneumoniae of nosocomial origin in Mérida, Venezuela
Introduction: Insertion sequences such as IS CR1 promote capture, transposition and expression of bla CTX-M genes. Thus, gene dissemination in bacterial populations occurs rapidly.
Objective: To determine the presence of IS CR1 sequence genes and their association with bla CTX-M-1 and bla CTX-M-2 on plasmids IncN and IncFIIA from K. pneumoniae of nosocomial origin, was determined.
Materials and methods: Three strains of K. pneumoniae with reduced susceptibility to extendedspectrum cephalosporins were isolated from neonatal sepsis cases of nosocomial origin. Phenotypic tests showed the presence of ESBLs. Plasmids were isolated and classified according to incompatibility groups by PCR replicon typing. Detection and association of IS CR1 with bla CTX-M genes were determined by PCR and direct sequencing through the use of several sets of PCR primers.
Results: All strains showed phenotypic profile consistent with ESBL-producing transferred by conjugation. PCR amplification assay for CTX-M together with sequencing analysis revealed that strains carrying bla CTX-M-1 y bla CTX-M-2 genes were localized in plasmids of approximately 150 kb related to IncN and IncFIIA groups, respectively. IS CR1 was found upstream and associated with bla CTX-M-1 y bla CTX-M-2 genes.
Conclusion. Thus far, this is the first Venezuelan report, in which IS CR1 presence is closely related to bla CTX-M-1 y bla CTX-M-2 gene mobilization in IncN and IncFIIA conjugative plasmids located in K. pneumonaiae strains circulating at a neonatal high risk unit.
Key words: Klebsiella pneumoniae , plasmids, cross infection, beta-lactamases, cefotaxime, DNA transposable elements.
doi: http://dx.doi.org/10.7705/biomedica.v33i2.774
La epidemiología de las ß -lactamasas de espectro extendido (BLEE) ha cambiado drásticamente en los últimos años. Hasta finales de la década de los años 90, las BLEE de tipo TEM y SHV fueron las más frecuentemente asociadas con Klebsiella pneumoniae en todo el mundo (1). Sin embargo, en la actualidad las ß -lactamasas TEM y SHV han disminuido su prevalencia notablemente, mientras que la diseminación de las BLEE de tipo cefotaximasas (CTX-M) se han incrementado en todos los continentes, en ambientes hospitalarios y extrahospitalarios, en humanos y animales; también se ha aislado en una gran variedad de especies de enterobacterias (1,2). La primera descripción de CTX-M fue hecha en 1989 y hasta el presente se conocen un poco más de 120 variantes genéticas (www.lahey.org/studies). Estas variantes se clasifican en cinco subgrupos principales según la homología de sus aminoácidos: CTX-M-1, CTXM- 2, CTX-M-8, CTX-M-9 y CTX-M-25 (1-3).
En la rápida diseminación de los genes bla CTX-M se han involucrado diversos elementos genéticos, como las secuencias de inserción IS CR1, IS 26, IS 903e ISEcp1, las cuales potencialmente facilitan la captura, transposición y expresión de estos genes (1,3,4). La secuencia IS CR1 se asocia estrechamente con integrones de clase 1 y se le atribuye la particularidad de movilizar, además de genes bla BLEE , otros factores determinantes de resistencia, como qnr y dfr10 , mediante mecanismos de transposición denominados rolling-circle (5-7).
Por otra parte, los plásmidos contribuyen de forma importante en la diseminación horizontal de genes de resistencia. Recientemente, se ha incrementado el interés por caracterizar los plásmidos de acuerdo con sus grupos de incompatibilidad (Inc), y su relación con la diseminación y distribución de BLEE (8). En cepas de origen clínico, los genes bla CTX-M se localizan en plásmidos grandes (40-350 kb) y transferibles; de acuerdo con su maquinaria de replicación, se clasifican comúnmente dentro de los grupos de incompatibilidad IncF, I, N, P y, con menor frecuencia, en los grupos IncA/C y L/M (8,9). El análisis del ambiente genético de los genes bla CTX-M puede revelar detalles interesantes relacionados con la adquisición, movilización y diseminación de estas ß -lactamasas.
Recientemente, Millán, et al . (10), reportaron la presencia de un plásmido conjugable del grupo IncF/FIB en Citrobacter freundii productor de BLEE de tipo CTX-M-14. Sin embargo, en Venezuela los estudios relacionados con el contexto genético de los genes codificantes para BLEE, especialmente los referentes a CTX-M, son escasos. Por tal motivo, en este trabajo determinamos la presencia de la secuencia IS CR1 y su asociación con genes bla CTXM- 1 y bla CTX-M-2 en plásmidos de diferentes grupos de incompatibilidad en cepas de K. pneumoniae de origen hospitalario.
Materiales y métodos
Cepas bacterianas
Se estudiaron tres cepas de K. pneumoniae (LMMKp27, LMMKp28 y LMMKp29) con disminución de la sensibilidad a las cefalosporinas de amplio espectro, pertenecientes a la colección del Laboratorio Bacteriología Clínica "Dr. Roberto Gabaldón" de la Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Mérida, Venezuela. Estas cepas relacionadas clonalmente se aislaron durante un brote de septicemia hospitalaria ocurrido en el año 2010, en neonatos recluidos en la Unidad de Alto Riesgo Neonatal del Instituto Autónomo Hospital Universitario de Los Andes, Mérida, Venezuela (Ghiglione B, Milllán B, González A, Gutkind G, Araque M. Klebsiella pneumoniae productora de CTX-M-2 aislada en una unidad de alto riesgo neonatal en Venezuela. Actas del XX Congreso Latinoamericano de Microbiología. Septiembre 27-30, 2010, Montevideo, Uruguay).
Detección fenotípica de BLEE y determinación de la sensibilidad antimicrobiana
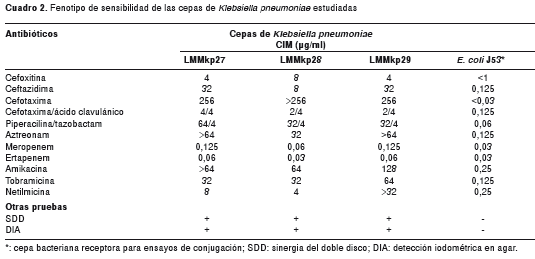
Inicialmente, se hizo la evaluación fenotípica de las cepas para detectar ß -lactamasas de espectro expandido (BLEE), mediante el ensayo de sinergia del doble disco (11). Posteriormente, se determinó la concentración inhibitoria mínima (CIM), mediante el método de dilución en agar, de acuerdo con lo establecido por el Clinical and Laboratory Standards Institute (CLSI) (11). Los antimicrobianos ensayados (Valmorca, Mérida, Venezuela) fueron: cefoxitina, ceftazidima, cefotaxima, aztreonam, cefotaxima/ ácido clavulánico, piperacilina/tazobactam, meropenem, ertapenem, amikacina, tobramicina y netilmicina. Se obtuvieron extractos crudos de cada cepa por sonicación y la actividad de las ß -lactamasas se confirmó mediante el método de detección iodométrica en agar usando como sustrato cefotaxima (12,13). Para estos ensayos se utilizaron las cepas Escherichia coli ATCC 25922 (BLEE -) y K. pneumoniae LMM60 (BLEE +) como controles negativo y positivo, respectivamente.
Transferencia por conjugación de genes que codifican para BLEE
Los factores determinantes de resistencia BLEE de las cepas de K. pneumoniae se transfirieron utilizando E. coli J53 resistente a la azida como cepa receptora. Las cepas donadoras y la receptora se inocularon en caldo Mueller-Hinton (BBL) y se incubaron a 37 °C en agitación, hasta alcanzar una densidad óptica entre 0,6-0,7 y 600 nm. Los cultivos se mezclaron en una proporción de 1:10 a favor de la cepa receptora y se incubaron durante toda la noche a 37 °C. A partir de este cultivo, se inoculó una alícuota 100 µl en placas de agar Mueller- Hinton con suplemento de 100 µg/ml de azida sódica (Sigma) y 2 µg/ml de cefotaxima. El fenotipo de las células obtenidas por transconjugación se verificó mediante las pruebas de sensibilidad de detección iodométrica en agar y PCR.
Extracción y análisis de plásmidos
Los plásmidos de las células obtenidas por transconjugación se extrajeron mediante lisis alcalina (14), se visualizaron en geles de agarosa al 0,8 %, se tiñeron con 50 µg/ml bromuro de etidio (Sigma) y se fotografiaron con el UVP Biodoc-It System . El tamaño de los plásmidos se determinó por comparación visual con otros de peso molecular conocidos: pLM07/20 (150kb) y pKAM542 (87kb) (10,15).
Los plásmidos se clasificaron preliminarmente de acuerdo con sus grupos de incompatibilidad y según el esquema de tipificación del replicón por PCR descrito por Carattoli, et al . (16).
Extracción del ADN genómico de las cepas de Klebsiella pneumoniae y sus respectivas células obtenidas por transconjugación
A partir de cultivos frescos en agar Mueller-Hinton, varias colonias de las cepas estudiadas se suspendieron nuevamente en 200 µl de agua bidestilada estéril ajustándose a una turbidez equivalente al patrón Nº 2 de McFarland. Esta suspensión fue colocada a ebullición por 10 minutos y se centrifugó a 13.000 rpm durante cinco minutos. Seguidamente, se transfirió el sobrenadante que contenía el ADN a un tubo Eppendorf® estéril, el cual se mantuvo a -20 °C hasta el momento de su uso.
Detección de genes bla BLEE
La detección de genes codificantes para TEM, SHV y CTX-M en las cepas estudiadas, células obtenidas por transconjugación y plásmidos, se hizo utilizando los iniciadores y condiciones de amplificación previamente descritas (cuadro 1). Las amplificaciones se llevaron a cabo en un volumen final de 25 µl y la mezcla estuvo compuesta por 2,5 µl del solución tampón de reacción (10X), 1,25 µl de MgCl 2 (50 Mm) , 1,5 de dNTP (10 mM; Fudaim, Venezuela), 2,5 µl de cada iniciador (10 pmol/ µl), 0,3 µl de la Taq polimerasa (5 U/µl; Fundaim, Venezuela), 12,45 µl de agua bidestilada ultrapura y 2 µl del ADN extraído. Las amplificaciones por PCR se realizaron en un termociclador Perkin Elmer. Los productos obtenidos se observaron en geles de agarosa al 1 %, teñidos con 50 µg/ml de bromuro de etidio, y se fotografiaron con el UVP Biodoc-It System . Como marcador de tamaño molecular, se utilizó una escalera de 100 pb (Bioneer).

Detección y asociación de ISCR1 con los genes bla CTX-M-1 y bla CTX-M-2
Inicialmente, se verificó la presencia del elemento ISCR1 por PCR, y su asociación con los genes bla CTXM-1 y bla CTX-M-2 se determinó mediante la combinación de varios juegos de iniciadores, siguiendo la estrategia del mapeo por PCR (cuadro 1).
Secuenciación de los productos amplificados
Todos los productos obtenidos a partir de las amplificaciones por PCR se purificaron utilizando el kit PCR-Accuprep (Bioneer) y las secuencias nucleotídicas fueron determinadas por el Centro de Secuenciación y Procesamiento de Ácidos Nucleicos (CeSAAN) del Instituto Venezolano de Investigaciones Científicas (IVIC), Caracas, Venezuela, mediante electroforesis capilar en un secuenciador modelo ABI 3130X2 (Applied Biosystem), utilizando los mismos iniciadores para la PCR. Las secuencias nucleotídicas resultantes se analizaron mediante el uso del programa Basic Local Alignment Search Tool (BLAST) del National Center for Biotechnology Information (NCBI) y se compararon con las secuencias genéticas incluidas en las base de datos (www.ncbi.nlm.nih.gov/blast/).
Resultados
Todas las cepas de K. pneumoniae estudiadas demostraron un perfil típico que indicaba producción de BLEE de acuerdo con los resultados de la CIM (cefotaxima >256 µg/ml y ceftazidima 8-32 µg/ml). La restitución de la sensibilidad a las cefalosporinas se observó con el ácido clavulánico (2-4 µg/ml). Los fenotipos de resistencia de estas cepas coincidieron con lo encontrado en el ensayo de sinergia del doble disco y la detección iodométrica de la actividad enzimática de BLEE en agar.
Estas cepas mostraron resistencia asociada con, al menos, dos aminoglucósidos (cepas LMMkp27 y LMMkp28: AMK-TOB y LMMkp29: AMK-NET). Las tres cepas fueron sensibles a los antibióticos carbapenémicos con CIM entre 0,03 y 0,125 µg/ ml (cuadro 2). Todos los factores determinantes de resistencia se transfirieron por conjugación en una frecuencia de 10 -4 a 10 -6 células obtenidas por transconjugación/célula donadora. Las células obtenidas por transconjugación mostraron un patrón de resistencia similar al de las cepas donadoras y fueron positivas en el ensayo de sinergia del doble disco y el método de detección iodométrica en agar. Los marcadores de resistencia a los aminoglucósidos fueron cotransferidos con BLEE.
En el cuadro 3 se muestran las características genéticas de las células obtenidas por transconjugación obtenidas. Los ADN provenientes de las células donadoras, las células obtenidas por transconjugación y plásmidos fueron analizados para detectar genes codificantes para las BLEE de tipo TEM, SHV y CTX-M.
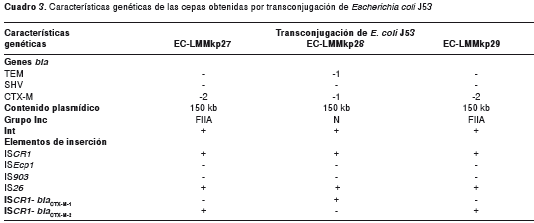
Las amplificaciones por PCR con iniciadores específicos para CTX-M y el análisis de la secuenciación, revelaron que las cepas LMMkp27 y LMMkp29 portaban genes bla CTX-M-2 , mientras que el bla CTX-M-1 asociado con bla TEM-1 fue detectado en la cepa LMMkp28. Los genes bla CTX-M-2 y bla CTX-M-1 se localizaron en plásmidos de tamaño aproximado a 150 kb, relacionados con los grupos IncFIIA e IncN, respectivamente.
Estos plásmidos se analizaron para detectar distintas secuencias de inserción (IS 26 , IS EcpI , IS 903 y IS CR1 ), además de otros genes asociadas con los genes bla , encontrándose que en todos los casos se detectó la secuencia IS CR1 (469 pb) y la IS 26 (500pb) . Con la estrategia de mapeo por PCR y utilizando los iniciadores 341F–blaUp en los pLMMkp27 y pLMMKp29, se obtuvo un amplicón de 2.100 pb. El análisis de la secuenciación determinó que la IS CR1 se encontraba a 260 pb aguas arriba y en la misma dirección del gen bla CTX-M-2 . En el caso del pLMMkp28, un amplicón de aproximadamente 2.700 pb se obtuvo con los iniciadores 341F–CTX-M-1R. De igual forma, se pudo deducir que el IS CR1 se encontraba aguas arriba y en el mismo sentido que el gen bla CTX-M-1 .
Discusión
Desde hace más de una década se ha reportado K. pneumoniae multirresistente productora de BLEE como causante de brotes de infección hospitalaria en neonatos recluidos en la Unidad de Alto Riesgo Neonatal del Instituto Autónomo Hospital Universitario de Los Andes (Mérida, Venezuela). Estos brotes se han caracterizado por una situación epidemiológica compleja, en la cual se han involucrado clones diferentes, presencia de distintas BLEE y sus combinaciones, así como diversas estructuras genéticas que portan genes bla BLEE (15,24,25). Actualmente, esta situación se mantiene.
Las tres cepas de K. pneumoniae analizadas en este estudio provenientes de neonatos con septicemia hospitalaria, aisladas en el año 2010, portaban genes que codifican para BLEE de tipo CTX-M; en dos de estas (LMMkp27 y LMMkp29) se detectaron genes bla CTX-M-2 , mientras que el bla CTXM-1 se encontró en la cepa LMMkp28. En ambos casos, la producción de las ß -lactamasas CTX-M se asociaron con resistencia a los amino glucósidos.
Algunos autores consideran este fenómeno de corresistencia en Enterobacteriaceae productoras de CTX-M, como uno de los factores que favorecen el proceso de selección de genes bla CTX-M , lo cual resulta en una alta incidencia y distribución en la población bacteriana (1,2).
En este contexto, los resultados de este estudio permitieron determinar que los genes bla CTXM-1 y bla CTX-M-2 se localizaron en plásmidos por conjugación de aproximadamente 150 kb, pertenecientes a los grupos de incompatibilidad IncN e IncFIIA, respectivamente.
Diestra, et al . (26) y Marcadé, et al . (27), reportaron resultados similares; ellos encontraron genes bla CTX-M-1 en plásmidos del grupo IncN en cepas de E. coli y K. pneumoniae de origen clínico. Sin embargo, Moodley y Guardabassi (28) y García-Fernández, et al . (29), afirman que los plásmidos IncN están involucrados en la transmisión de genes bla CTX-M-1 cuyo reservorio es principalmente animal. Este hallazgo puede ser sustentado por el uso y el abuso de cefalosporinas en la medicina veterinaria. Los plásmidos IncN se consideran de amplio espectro bacteriano. Por el contrario, los plásmidos IncF son de estrecho rango, limitándose especialmente a los miembros de la familia Enterobacteriaceae , pero su gran versatilidad de adaptación celular y evolución de sus mecanismos de replicación, los relacionan con la diseminación abrupta de las más relevantes variantes de BLEE de tipo CTX-M en humanos (30); esta afirmación es avalada por los resultados obtenidos en este trabajo.
Las BLEE de tipo CTX-Ms se derivan de las ß -lactamasas cromosómicas de especies de Kluyvera y la movilización de los genes bla CTX-M ocurre gracias a la asistencia de los llamados elementos de inserción, tales como IS CR1 (6). Esta secuencia de inserción anteriormente llamada orf513 , que codifica para una recombinasa hipotética y que suele encontrarse en integrones de clase 1, se ha asociado previamente con genes bla CTX-M-2 , bla CTX-M-9 y bla CTX-M-14 (2). En este estudio pudimos determinar la presencia del IS CR 1 a 260 pb aguas arriba y en el mismo sentido que el gen bla CTX-M-2. Por otra parte, se ha descrito que los genes bla CTX-M-1 están más frecuentemente asociados con la secuencia de inserción IS Ecp1 (19,31). Sin embargo, Su, et al ., (32) reportaron por primera vez genes bla CTXM-1 asociados con el elemento IS CR1, formando parte de un integrón de clase 1 en cepas de E. coli aisladas del hospital de la Universidad de Jiangsu en China. Una estructura genética semejante fue encontrada en la cepa LMMKp28 del presente estudio, donde el gen bla CTX-M-1 se asoció con el IS CR1. Estos resultados podrían estar indicando la presencia de una plataforma genética poco común, lo que requerirá una caracterización más detallada en estudios futuros. No obstante, los hallazgos obtenidos sugieren que el elemento de IS CR1 ha promovido los eventos de expresión y movilización de los genes bla CTX-M-1 y bla CTX-M-2 , así como los mecanismos de recombinación entre los diferentes plásmidos estudiados.
En conclusión, esta es la primera descripción realizada en Venezuela, en la cual la presencia de IS CR1 está estrechamente asociada con la movilización de los genes bla CTX-M-1 y bla CTX-M-2 en plásmidos por conjugación de diferentes grupos de incompatibilidad, IncN y IncFIIA, en cepas de K. pneumoniae que circulan en la Unidad de Alto Riesgo Neonatal del Instituto Autónomo Hospital Universitario de Los Andes. En este estudio se enfatiza la necesidad de aplicar políticas estrictas para el uso de los antibióticos e implementar medidas para la contención epidemiológica de los genes que codifican para las BLEE.
Los autores agradecen a Ana C. González, por su colaboración por el suministro de las cepas.
Los autores declaran no tener conflictos de intereses.
Este trabajo fue realizado dentro de las actividades del Proyecto Bacterialnet ALFA II NºII-531-FC-FA274 Millán B, Castro D, Araque M, et al . Biomédica 2013;33:268-75 FCD-FI y parcialmente financiado por el Consejo de Desarrollo Científico, Humanístico, Tecnológico y de las Artes (CDCHTA-ULA) de la Universidad de Los Mérida, Venezuela (CVI-ADG-FA-02-97).
Correspondencia: Beatriz Millán, Laboratorio de Microbiología Molecular, Departamento de Microbiología y Parasitología, Facultad de Farmacia y Bioanálisis, Universidad de Los Andes, Sector Campo de Oro, Mérida 5101, Venezuela. Telefax: (58 274) 240 3180
Referencias
1. Cantón R, González-Alba JM, Galán JC. CTX-M enzymes: origen and diffusion. Front Microbiol. 2012;3:10. http://dx.doi.org/103389/fmicb.2012.00110
2. Cantón R, Coque TM. The CTX-M ß -lactamase pandemic. Curr Opin Microbiol. 2006;9:466-75. http://dx.doi.org/10.1016/j.mib.2006.08.011
3. Lartigue MF, Poirel L, Normand P. Diversity of genetic enviromental of bla CTX-M genes. FEMS Microbiol Lett. 2004; 234:201-7. http://dx.doi.org/10.1016/j.femsle.2004.01.051
4. Navarro F, Miró E. Entorno genético de las BLEE: implicaciones en la transmisión. Enferm Infec Microbiol Clin. 2007;25(Suppl.2):11-7.
5. Toleman MA, Bennett PM, Walsh TR. ISCR elements: Novel gene-capturing systems of the 21 st century? Microbiol Mol Biol Rev. 2006;70:296-316. http://dx.doi.org/10.1128/MMBR.00048-05.
6. Lee Ch-H, Liu JW, Li ChCh, Chien ChCh, Tang YF, Su Lh. Spread of IS CR1 elements containing bla DHA-1 and multiple antimicrobial resistance genes leading to increase of flomoxef resistance in extended-spectrum- ß -lactamaseproducing Klebsiella pneumoniae . Antimicrob Agents Chemother. 2011;55:4058-63. http://dx.doi.org/10.1128/AAC.00259-11.
7. Chen YT, Liao TL, Liu YM, Lauderdale TL, Yan JJ, Tsai SF. Mobilization of qnrB2 and ISCR in plasmids. Antimicrob Agents Chemother. 2009;53:1235-37. http://dx.doi.org/10.1128/AAC.00970-08
8. Carattoli A. Plasmids in Gram negatives: Molecular typing of resistance plasmids. Int J Med Microbiol. 2011;301:654-58. http://dx.doi.org/10.1128/AAC.00970-08.
9. Carattoli A. Resistance plasmid families in Enterobacteriaceae . Antimicrob Agents Chemother. 2009;53:2227-38. http://dx.doi.org/10.1128/AAC.01707-08.
10. Millán B, Ghiglione B, Díaz T, Gutkind G, Araque M. CTXM- 14 ß -lactamase-producing Citrobacter freundii isolated in Venezuela. Ann Clin Microb Antimicrob. 2011;10:22. http://dx.doi.org/10.1186/1476-0711-10-22
11. Clinical and Laboratory Standards Institute (CLSI). Performance standards for antimicrobial susceptibility testing, 21th informational supplement. CLSI Document M100-S20. Wayne, Pennsylvania: Clinical and Laboratory Standards Institute; 2011.
12. Labia R, Barthélémy M. Beta-lactamase enzymogram: An agar adaptation of the iodometric method. Ann Microbiol 1979;30:295-304.
13. Rossi A, Lopardo H, Picandet AM, Mariño M, Galds M, Radice M, et al . Non-typhoid Salmonella spp. resistant to cefotaxime. J Antimicrobial Chemother. 1995;36:697-702. http://dx.doi.org/10.1093/jac/36.4.697
14. Birmboim HC, Doly J. A rapid alkaline extraction procedure for screening recombinant plasmid DNA. Nucleic Acid Res. 1979;7:1513-23.
15. Araque M, Rivera I. Simultaneous presence of bla TEM and bla SHV genes on a large conjugative plasmid carried by extended-spectrum ß -lactamase-producing Klebsiella pneumoniae. Am J Med Sci. 2004;327:118-22.
16. Carattoli A, Bertini A, Villa L, Falbo V, Hopkins KL, Threlfall EJ. Identification of plasmids by PCR-based replicon typing. J Microb Methods. 2005;63:219-28. http://dx.doi.org/10.1016/j.mimet.2005.03.018
17. Chang FY, Siu LK, Fung CP, Huang MH, Ho M. Diversity of SHV and TEM ß -Lactamases in Klebsiella pneumoniae : Gene evolution in Northern Taiwan and two novel ß -Lactamases, SHV-25 and SHV-26. Antimicrob Agents Chemother.2001;45:2407-13. http://dx.doi.org/10.1128/AAC.45.9.2407-2413.2001
18. Ma L, Chang FY, Fung CP, Chen TL, Lin JC, Lu PL, et al. Variety of TEM-, SHV-, and CTX-M-type ß -lactamases present in recent clinical isolates of Escherichia coli, Klebsiella pneumoniae, and Enterobacter cloacae from Taiwan. Microb Drug Resist. 2005;11:31-9. http://dx.doi.org/10.1089/mdr.2005.11.31
19. Eckert C, Gautier V, Arlet G. DNA sequence analysis of the genetic environment of various bla CTX-M genes. J Antimicrob Chemother.2006;57:14-23. http://dx.doi.org/10.1093/jac/dki398.
20. Di Conza, Ayala JA, Power P, Mollerach M, Gutkind G. Novel class 1 integron (InS21) carrying bla CTX-M-2 in Salmonella enterica serovar infantis. Antimicrob Agents Chemother.2002;46:2257-61. http://dx.doi.org/10.1128/AAC.46.7.2257-2261.2002
21. Poirel L, Decousser JW, Nordmann P. Insertion sequence IS Ecp1B is involved in expression and mobilization of a bla CTX-M ß -lactamase gene. Antimicrob Agents Chemother. 2003;47:293845. http://dx.doi.org/10.1128/AAC.47.9.2938-2945.2003
22. Saladin M, Cao VT, Lambert T, Donay JL, Herrmann JL, Ould-Hocine Z, et al. Diversity of CTX-M ß -lactamases and their promoter regions from Enterobacteriaceae isolated in three Parisian hospitals. FEMS Microbiol. Lett. 2002;209:161-8.
23. Sabaté M, Tarrago R, Navarro F, Miro E, Verges C, Barbe J, et al. Cloning and sequence of the gene encoding a novel cefotaxime hydrolyzing ß -lactamase (CTXM-9) from Escherichia coli in Spain. Antimicrob. Agents Chemother. 2002;44:1970-3. http://dx.doi.org/10.1128/AAC.46.8.2656-2661.2002.
24 . Araque M, Nieves B, Lauretti L, Rossolini GM. Molecular basis of extended-spectrum ß -lactamase production in nosocomial isolates of Klebsiella pneumoniae from Mérida, Venezuela. Int J Antimicrob Agents. 2000;15:37-42. http://dx.doi.org/10.1016/S0924-8579(99)00168-5
25 . González A. Gil F, Solorzano M, Cruz J, Püig, J, Nieves B. Brote por Klebsiella pneumoniae multirresistente y productora de ß -lactamasa de espectro extendido en una unidad de alto riesgo neonatal. Rev Chil Infectol. 2011;28:28-34. http://dx.doi.org/10.4067/S0716-10182011000100005
26. Diestra K, Juan C, Curiao T, Moya B, Miró E, Oteo J, et al. Characterization of plasmids encoding bla ESBL and surrounding genes in Spanish clinical isolates of Escherichia coli and Klebsiella pneumoniae. J Antimicrob Chemother. 2009;63:60-6. http://dx.doi.org/10.1093/jac/dkn453275
27. Marcadé G, Deschamps C, Boyd A, Gautier V, Picard B, Branger C, et al. Replicon typing of plasmids in Escherichia coli producing extended-spectrum ß -lactamases. J Antimicrob Chemother. 2009;63:67-71. http://dx.doi.org/10.1093/jac/dkn428
28. Moodley A, Guardabassi L. Transmission of IncN plasmids carrying bla CTX-M-1 between commensal Escherichia coli in pigs and farm workers. Antimicrob. Agents Chemother. 2009;53:1709-11. http://dx.doi.org/10.1128/AAC.01014-08
29. García-Fernández A, Villa L, Moodley A, Hasman H, Miriagou V, Guardabassi L, et al. Multilocus sequence typing of IncN plasmids. J. Antimicrob. Chemother. 2011;66:1987-991. http://dx.doi.org/10.1093/jac/dkr225
30. Villa L, García A, Fortini D, Carattoli A. Replicon sequence typing of IncF plasmids carrying virulence and resistance determinants. J Antimicrob Chemother. 2010;65:2518-29. http://dx.doi.org/10.1093/jac/dkq347
31. Celis YA, Pulido IY, Valenzuela EM, Reguero MT, Mantilla JR. Ambiente genético del gen bla CTX-M-12 en aislamientos hospitalarios de Klebsiella pneumoniae. Rev Colomb Biotecnol. 2009;11:48-58.
32. Su Z, Dai X, Chen J, Kong F, Wang H, Li Y. The bla CTX-M-1 gene located in a novel complex class I integron bearing an IS CR1 element in Escherichia coli isolates from Zhenjiang, China. J Antimicrob Chemother 2008;62:1150-64. http://dx.doi.org/10.1093/jac/dkn300