Caracterización inmunológica de un grupo familiar colombiano con infección por SARS-CoV-2
Resumen
Introducción. Se han descrito diferentes marcadores inmunológicos durante la COVID-19, los cuales persisten incluso después de la convalecencia y se asocian con los estadios clínicos de la infección. Sin embargo, aún son pocos los estudios orientados al análisis exhaustivo de las alteraciones del sistema inmunológico en el curso de la infección.
Objetivo. Evaluar la producción de citocinas proinflamatorias, la reacción de anticuerpos, y el fenotipo y la función de las células NK y los linfocitos T en una familia colombiana con infección por SARS-CoV-2.
Materiales y métodos. Se evaluaron las citocinas proinflamatorias mediante RT-PCR y ELISA; la frecuencia, el fenotipo y la función de las células NK (en cocultivos con células K562) y linfocitos T CD8+ (estimulados con péptidos spike/RdRp) mediante citometría de flujo, y los anticuerpos anti-SARS-CoV-2, mediante inmunofluorescencia indirecta y prueba de neutralización por reducción de placa.
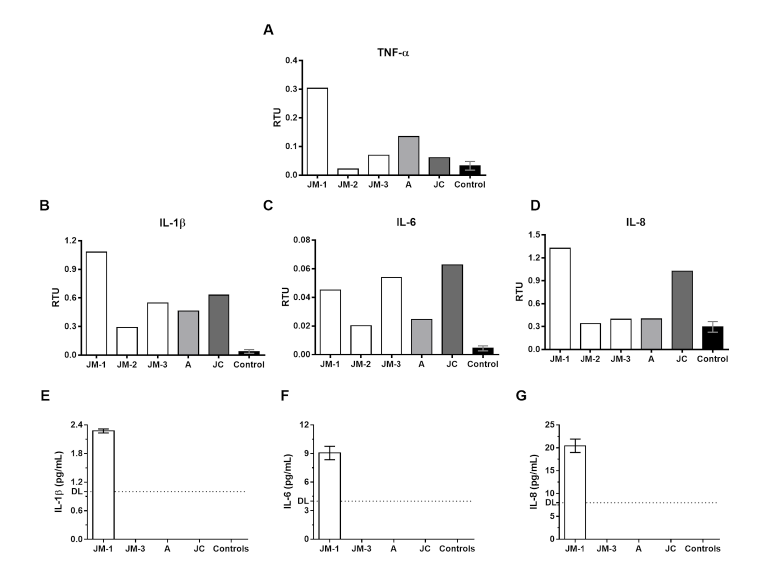
Resultados. Durante la COVID-19 hubo una producción elevada de citocinas proinflamatorias, con disminución de las células NK CD56bright y reacción citotóxica. Comparados con los controles sanos, los individuos infectados presentaron con gran frecuencia linfocitos T CD8+ disfuncionales CD38+HLA-DR-. Además, en los linfocitos T CD8+ estimulados con péptidos virales, predominó una reacción monofuncional con gran producción de IL-10 durante la fase aguda y una reacción bifuncional caracterizada por la coexpresión de CD107a y granzima B o perforina durante la convalecencia.
Conclusión. Aunque la reacción inflamatoria caracteriza la infección por SARS-CoV-2, hay otras alteraciones fenotípicas y funcionales en células NK y linfocitos T CD8+ que podrían asociarse con la progresión de la infección. Se requieren estudios adicionales para entender estas alteraciones y guiar futuras estrategias de inmunoterapia.
Descargas
Referencias bibliográficas
Li Q, Guan X, Wu P, Wang X, Zhou L, Tong Y, et al. Early transmission dynamics in Wuhan, China, of novel coronavirus-infected pneumonia. N Engl J Med. 2020;382:1199-207. https://doi.org/10.1056/NEJMoa2001316
Chen Y, Liu Q, Guo D. Emerging coronaviruses: Genome structure, replication, and pathogenesis. J Med Virol. 2020;92:418-23. https://doi.org/10.1002/jmv.25681
Ortiz ME, Thurman A, Pezzulo AA, Leidinger MR, Klesney-Tait JA, Karp PH, et al. Heterogeneous expression of the SARS-Coronavirus-2 receptor ACE2 in the human respiratory tract. EBioMedicine. 2020;60:102976. https://doi.org/10.1016/j.ebiom.2020.102976
Chu H, Chan JFW, Wang Y, Yuen TT, Chai Y, Hou Y, et al. Comparative replication and immune activation profiles of SARS-CoV-2 and SARS-CoV in human lungs: An ex vivo study with implications for the pathogenesis of COVID-19. Clin Infect Dis. 2020;71:1400-9. https://doi.org/10.1093/cid/ciaa410
Huang C, Wang Y, Li X, Ren L, Zhao J, Hu Y, et al. Clinical features of patients infected with 2019 novel coronavirus in Wuhan, China. Lancet. 2020;395:497-506. https://doi.org/10.1016/S0140-6736(20)30183-5
Yuan X, Tong X, Wang Y, Wang H, Wang L, Xu X. Coagulopathy in elderly patients with coronavirus disease 2019. AGING Med. 2020;3:260-5. https://doi.org/10.1002/agm2.12133
Ponti G, Maccaferri M, Ruini C, Tomasi A, Ozben T. Biomarkers associated with COVID-19 disease progression. Crit Rev Clin Lab Sci. 2020;57:1-11. https://doi.org/10.1080/10408363.2020.1770685
Taborda NA, Hernández JC, Montoya CJ, Rugeles MT. Natural killer cells and their role in the immune response during human immunodeficiency virus type-1 infection. Inmunologia. 2014;33:11-20. https://doi.org/10.1016/j.inmuno.2013.11.002
Alrubayyi A. NK cells in COVID-19: Protectors or opponents? Nat Rev Immunol. 2020;20:520. https://doi.org/10.1038/s41577-020-0408-0
Della Chiesa M, Sivori S, Carlomagno S, Moretta L, Moretta A. Activating KIRs and NKG2C in viral infections: Toward NK cell memory? Front Immunol. 2015;6:1-8. https://doi.org/10.3389/fimmu.2015.00573
Schlums H, Cichocki F, Tesi B, Theorell J, Beziat V, Holmes TD, et al. Cytomegalovirus infection drives adaptive epigenetic diversification of NK cells with altered signaling and effector function. Immunity. 2015;42:443-56. https://doi.org/10.1016/j.immuni.2015.02.008
Flórez-Álvarez L, Blanquiceth Y, Contreras K, Ossa-Giraldo AC, Velilla PA, Hernández JC, et al. NK cell activity and CD57+/NKG2Chigh phenotype are increased in men who have sex with men at high risk for HIV. Front Immunol. 2020;11:1-14. https://doi.org/10.3389/fimmu.2020.537044
Zheng M, Gao Y, Wang G, Song G, Liu S, Sun D, et al. Functional exhaustion of antiviral lymphocytes in COVID-19 patients. Cell Mol Immunol. 2020;17:533-5. https://doi.org/10.1038/s41423-020-0402-2
Sun B, Feng Y, Mo X, Zheng P, Wang Q, Li P, et al. Kinetics of SARS-CoV-2 specific IgM and IgG responses in COVID-19 patients. Emerg Microbes Infect. 2020;9:940-8. https://doi.org/10.1080/22221751.2020.1762515
Wang Y, Zhang L, Sang L, Ye F, Ruan S, Zhong B, et al. Kinetics of viral load and antibody response in relation to COVID-19 severity. J Clin Invest. 2020;130:5235-44. https://doi.org/10.1172/JCI138759
Huang I, Pranata R. Lymphopenia in severe coronavirus disease-2019 (COVID-19): Systematic review and meta-analysis. J Intensive Care. 2020;8:1-10. https://doi.org/10.1186/s40560-020-00453-4
Centers for Disease Control and Prevention-CDC. Real-time RT-PCR Primers and Probes for COVID-19. Accessed on: April 2, 2020. Available from: https://www.cdc.gov/coronavirus/2019-ncov/lab/rt-pcr-panel-primer-probes.html
Feria MG, Taborda NA, Hernández JC, Rugeles MT. HIV replication is associated to inflammasomes activation, IL-1β, IL-18 and caspase-1 expression in GALT and peripheral blood. PLoS ONE. 2018;13:e0192845. https://doi.org/10.1371/journal.pone.0192845
Flórez-Álvarez L, Blanquiceth Y, Ramírez K, Ossa-Giraldo AC, Velilla PA, Hernández JC, et al. NK cell activity and CD57+/NKG2Chigh phenotype are increased in men who have sex with men at high risk for HIV. Front Immunol. 2020;11:1-14. https://doi.org/10.3389/fimmu.2020.537044
Díaz FJ, Aguilar-Jiménez W, Flórez-Álvarez L, Valencia G, Laiton-Donato K, Franco-Muñoz C, et al. Aislamiento y caracterización de una cepa temprana de SARS-CoV-2 durante la epidemia de 2020 en Medellín, Colombia. Biomédica. 2020;40(Supl.2):148-58. https://doi.org/10.7705/biomedica.5834
Cai Y, Zhang J, Xiao T, Peng H, Sterling SM, Walsh RM, et al. Distinct conformational states of SARS-CoV-2 spike protein. Science. 2020;369:1586-92. https://doi.org/10.1126/science.abd4251
Gao Y, Yan L, Huang Y, Liu F, Zhao Y, Cao L, et al. Structure of the RNA-dependent RNA polymerase from COVID-19 virus. Science. 2020;368:779-82. https://doi.org/10.1126/science.abb7498
Luna OF, Gómez J, Cárdenas C, Albericio F, Marshall SH, Guzmán F. Deprotection reagents in Fmoc solid phase peptide synthesis: Moving away from piperidine? Molecules. 2016;21:1-12. https://doi.org/10.3390/molecules21111542
Perdomo-Celis F, Velilla PA, Taborda NA, Rugeles MT. An altered cytotoxic program of CD8 + T-cells in HIV-infected patients despite HAART-induced viral suppression. PLoS ONE. 2019;14:1-24. https://doi.org/10.1371/journal.pone.0210540
Roederer M, Nozzi JL, Nason MC. SPICE: Exploration and analysis of post-cytometric complex multivariate datasets. Cytom Part A. 2011;79:167-74. https://doi.org/10.1002/cyto.a.21015
Shi Y, Wang G, Cai X, Deng J, Zheng L, Zhu H, et al. An overview of COVID-19. J Zhejiang Univ Sci B. 2020;21:343-60. https://doi.org/10.1631/jzus.B2000083
Ge H, Wang X, Yuan X, Xiao G, Wang C, Deng T, et al. The epidemiology and clinical information about COVID-19. Eur J Clin Microbiol Infect Dis. 2020;39:1011-9. https://doi.org/10.1007/s10096-020-03874-z
García LF. Immune response, inflammation, and the clinical spectrum of COVID-19. Front Immunol. 2020;11:4-8. https://doi.org/10.3389/fimmu.2020.01441
Azkur AK, Akdis M, Azkur D, Sokolowska M, van de Veen W, Brüggen MC, et al. Immune response to SARS-CoV-2 and mechanisms of immunopathological changes in COVID-19. Allergy Eur J Allergy Clin Immunol. 2020;75:1564-81. https://doi.org/10.1111/all.14364
Ni L, Ye F, Cheng ML, Feng Y, Deng YQ, Zhao H, et al. Detection of SARS-CoV-2-specific humoral and cellular immunity in COVID-19 convalescent individuals. Immunity. 2020;52:971-7.e3. https://doi.org/10.1016/j.immuni.2020.04.023
Duan K, Liu B, Li C, Zhang H, Yu T, Qu J, et al. Effectiveness of convalescent plasma therapy in severe COVID-19 patients. Proc Natl Acad Sci USA. 2020;117:9490-6. https://doi.org/10.1073/pnas.2004168117
Wang X, Guo X, Xin Q, Pan Y, Hu Y, Li J, et al. Neutralizing antibody responses to severe acute respiratory syndrome coronavirus 2 in coronavirus disease 2019 in patients and convalescent patients. Clin Infect Dis. 2020;71:2688-94. https://doi.org/10.1093/cid/ciaa721
Long QX, Tang XJ, Shi QL, Li Q, Deng HJ, Yuan J, et al. Clinical and immunological assessment of asymptomatic SARS-CoV-2 infections. Nat Med. 2020;26:1200-4. https://doi.org/10.1038/s41591-020-0965-6
Qin C, Zhou L, Hu Z, Zhang S, Yang S, Tao Y, et al. Dysregulation of immune response in patients with Coronavirus 2019 (COVID-19) in Wuhan, China. Clin Infect Dis. 2020;71:762-8. https://doi:10.1093/cid/ciaa248
Han H, Ma Q, Li C, Liu R, Zhao L, Wang W, et al. Profiling serum cytokines in COVID-19 patients reveals IL-6 and IL-10 are disease severity predictors. Emerg Microbes Infect. 2020;9:1123-30. https://doi.org/10.1080/22221751.2020.1770129
Le Garff-Tavernier M, Béziat V, Decocq J, Siguret V, Gandjbakhch F, Pautas E, et al. Human NK cells display major phenotypic and functional changes over the life span. Aging Cell. 2010;9:527-35. https://doi.org/10.1111/j.1474-9726.2010.00584.x
Kang CK, Han GC, Kim M, Kim G, Shin HM, Song KH, et al. Aberrant hyperactivation of cytotoxic T-cell as a potential determinant of COVID-19 severity. Int J Infect Dis. 2020;97:313-21. https://doi.org/10.1016/j.ijid.2020.05.106
Payen D, Cravat M, Maadadi H, Didelot C, Prosic L, Dupuis C, et al. A longitudinal study of immune cells in severe COVID-19 patients. Front Immunol. 2020:11:580250. https://doi.org/10.3389/fimmu.2020.580250
Mazzoni A, Salvati L, Maggi L, Capone M, Vanni A, Spinicci M, et al. Impaired immune cell cytotoxicity in severe COVID-19 is IL-6 dependent. J Clin Invest. 2020:130:4694-703. https://doi:10.1172/jci138554
Zheng HY, Zhang M, Yang CX, Zhang N, Wang XC, Yang XP, et al. Elevated exhaustion levels and reduced functional diversity of T cells in peripheral blood may predict severe progression in COVID-19 patients. Cell Mol Immunol. 2020;17:541-3. https://doi.org/10.1038/s41423-020-0401-3
De Biasi S, Meschiari M, Gibellini L, Bellinazzi C, Borella R, Fidanza L, et al. Marked T cell activation, senescence, exhaustion and skewing towards TH17 in patients with COVID-19 pneumonia. Nat Commun. 2020;11:1-17. https://doi.org/10.1038/s41467-020-17292-4
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |