Genotipificación de aislamientos del complejo Mycobacterium tuberculosis mediante MIRU-VNTR, Cali, Colombia, 2013-2015
Resumen
Introducción. La tuberculosis continúa siendo uno de los problemas de salud más importantes a nivel mundial y, con la infección por el virus de la inmunodeficiencia humana (HIV), constituye la principal causa de muerte por infecciones. En el 2016, se notificaron 6,3 millones de casos nuevos de la enfermedad.
Objetivo. Describir los patrones genéticos determinados mediante la genotipificación del número variable de repeticiones en tándem de unidades repetitivas interespaciadas de micobacterias (Mycobacterial Interspersed Repetitive Units - Variable Number of Tandem Repeats, MIRU-VNTR) en la población de estudio y compararlos con los hallados en otros estudios locales e internacionales.
Materiales y métodos. Mediante MIRU-VNTR, entre el 2013 y el 2015 se hizo la genotipificación de 105 muestras de ADN extraídas del esputo o de aislamientos en cultivo de M. tuberculosis provenientes de pacientes residentes en Cali con diagnóstico de tuberculosis pulmonar. La amplificación de 24 loci MIRU-VNTR se hizo por medio de la reacción en cadena de la polimerasa (PCR). Los amplicones resultantes se visualizaron por electroforesis en geles de agarosa (2 %) teñidos con SYBR Safe™. La asignación de los alelos se hizo con un análisis gráfico con el programa GelAnalyzer 2010. Los resultados obtenidos se analizaron con el algoritmo UPGMA y se compararon con las bases de datos internacionales MIRU-VNTRplus y SITVITWEB.
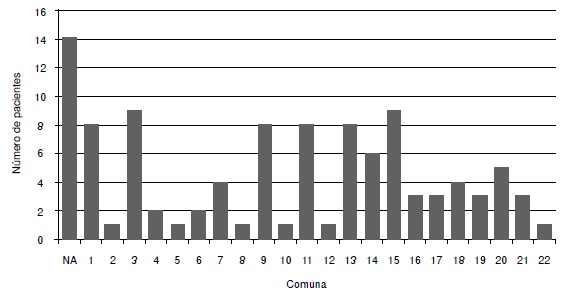
Resultados. Se genotipificaron por completo 62 de las muestras y se obtuvieron 58 perfiles diferentes de MIRU-VNTR. Al comparar con las bases de datos internacionales, su distribución por linajes fue la siguiente: 54,8 % para el LAM, 25,8 % para el Haarlem, 14,5 % para el S, 3,2 % para el Beijing y 1,6 % para el Cameroon. Los patrones MIRU-VNTR correspondieron a 20 tipos internacionales de MIRU (MIRU International Types, MIT) diferentes, y los más frecuentes fueron el MIT 190 y el MIT 110, con 22,6 y 6,5 %, respectivamente.
Conclusión. Estos resultados confirmaron hallazgos previos sobre el predominio de los linajes LAM y Haarlem en la ciudad y la presencia de los MIT encontrados en otra ciudad de Colombia.
Descargas
Referencias bibliográficas
World Health Organization. Global tuberculosis control: WHO Report 2017. Geneva: World Health Organization; 2017. p. 224.
Organización Mundial de la Salud. Preguntas y respuestas: tuberculosis ultrarresistente (TB-XR). Fecha de consulta: 27 de mayo de 2018. Disponible en: http://www.who.int/features/qa/extensively-resistant-tuberculosis/es/
Pedraza L, García C, Muñoz A. Caracterización de pacientes con tuberculosis y tuberculosis resistente a múltiples medicamentos en instituciones de tercer nivel de Bogotá D.C. Enferm Glob. 2012;25:129-38 https://doi.org/10.4321/S1695-61412012000100008
Instituto Nacional de Salud. Informe del evento tuberculosis, Colombia, 2017. Fecha de consulta: 27 de mayo de 2018. Disponible en: http://www.ins.gov.co/buscador-eventos/Informesdeevento/TUBERCULOSIS%202017.pdf
Secretaría Departamental del Salud del Valle. Boletín epidemiológico semanal. Semana epidemiológica número 52 de 2015. Fecha de consulta: 10 de mayo de 2015. Disponible en: http://www.valledelcauca.gov.co/salud/descargar.php?id=17080
Secretaría Departamental del Salud del Valle. Informe anual, 2016: Vigilancia en salud pública. Fecha de consulta: 27 de mayo de 2017. Disponible en: http://www.valledelcauca.gov.co/salud/descargar.php?id=19934
Mathema B, Kurepina N, Bifani P, Kreiswirth B. Molecular epidemiology of tuberculosis: Current insights. Clin Microbiol Rev. 2006;19:658-85. https://doi.org/10.1128/CMR.00061-05
Kato-Maeda M, Small P. How molecular epidemiology has changed what we know about tuberculosis. West J Med. 2000;172:256-9.
van Soolingen D, De Haas W, Kremer K. Restriction fragment length polymorphism (RFLP) typing of micobacteria. Methods Mol Med. 2001;54:165-203. https://doi.org/10.1385/1-59259-147-7:165
Chaoui I, Zozio T, Lahlou O, Sabouni R, Abid M, El Aouad R, et al. Contribution of spoligotyping and MIRU-VNTRs to characterize prevalent Mycobacterium tuberculosis genotypes infecting tuberculosis patients in Morocco. Infect Genet Evol. 2014;21:463-71. https://doi.org/10.1016/j.meegid.2013.05.023
Cowan L, Mosher L, Diem L, Massey J, Crawford J. Variable-number tandem repeat typing of Mycobacterium tuberculosis isolates with low copy numbers of IS6110 by using mycobacterial interspersed repetitive units. J. Clin Microbiol. 2002;40:1592-602. https://doi.org/10.1128/JCM.40.5.1592-1602.2002
Pitondo-Silva A, Santos A, Jolley K, Leite C, Darini A. Comparison of three molecular typing methods to assess genetic diversity for Mycobacterium tuberculosis. J Microbiol Methods. 2013;93:42-8. https://doi.org/10.1016/j.mimet.2013.01.020
Barnes P, Cave M. Molecular epidemiology of tuberculosis. N Engl J Med. 2003;349:1149-56. https://doi.org/10.1056/NEJMra021964
van Embden J, Cave M, Crawford J, Dale J, Eisenach K, Gicquel B, et al. Strain identification of Mycobacterium tuberculosis by DNA fingerprinting: Recommendations for a standardized methodology. J Clin Microbiol. 1993;31:406-9.
Rozo J, Ribón W. Molecular tools for Mycobacterium tuberculosis genotyping. Rev Salud Pública (Bogotá). 2010;12:510-21.
Cowan L, Diem L, Monson T, Wand P, Temporado D, Oemig T, et al. Evaluation of a two-step approach for large-scale, prospective genotyping of Mycobacterium tuberculosis isolates in the United States. J Clin Microbiol. 2005;43:688-95. https://doi.org/10.1128/JCM.43.2.688-695.2005
Kamerbeek J, Schouls L, Kolk A, van Agterveld M, van Soolingen D. Simultaneous detection and strain differentiation of Mycobacterium tuberculosis for diagnosis and epidemiology. J Clin Microbiol. 1997;35:907-14.
Supply P, Allix C, Lesjean S, Cardoso-Oelemann M, Rüsch-Gerdes S, Willery E. Proposal for standardization of optimized mycobacterial interspersed repetitive unit-variable-number tandem repeat typing of Mycobacterium tuberculosis. J Clin Microbiol. 2006;44:4498-510. https://doi.org/10.1128/JCM.01392-06
Allix-Beguec C, Harmsen D, Weniger T, Supply P, Niemann S. Evaluation and strategy for use of MIRU-VNTRplus, a multifunctional database for online analysis of genotyping data and phylogenetic identification of Mycobacterium tuberculosis complex isolates. J Clin Microbiol. 2008;46:2692-9. https://doi.org/10.1128/JCM.00540-08
Demay C, Liens B, Burguière T, Hill V, Couvin D, Millet J, et al. SITVITWEB – A publicly available international multimarker database for studying Mycobacterium tuberculosis genetic diversity and molecular epidemiology. Infect Genet Evol. 2012;12:755-66. https://doi.org/10.1016/j.meegid.2012.02.004
Shabbeer A, Ozcaglar C, Yener B, Bennett K. Web tools for molecular epidemiology of tuberculosis. Infect Genet Evol. 2012;12:767-81. https://doi.org/ 10.1016/j.meegid.2011.08.019
García de Viedma D, Mokrousov I, Rastogi N. Innovations in the molecular epidemiology of tuberculosis. Enferm Infecc Microbiol Clin. 2011;29:8-13. https://doi.org/10.1016/S0213-005X(11)70012-X
Organización Panamericana de la Salud. Manual para el diagnóstico bacteriológico de la tuberculosis. Parte 2: Cultivo. Fecha de consulta: 10 de febrero de 2015. Disponible en: http://www1.paho.org/Spanish/AD/DPC/CD/tb-labs-cultivo.pdf
Sneath P, Sokal R. Numerical Taxonomy: The principles and practice of numerical classification. San Francisco, CA: Freeman and Company; 1973. p. 549.
Rodríguez N, Martínez M, Herranz M, Sánchez M, Barroso P, Bouza E, et al. Evaluation of the new advanced 15-loci MIRU-VNTR genotyping tool in Mycobacterium tuberculosis epidemiology studies. BMC Microbiol. 2008;8:34. https://doi.org/10.1186/1471-2180-8-34
Cerezo I, Jimenez Y, Hernández J, Zozio T, Murcia M, Rastogi N. First insight on the population structure of Mycobacterium tuberculosis complex as studied by spoligotyping and MIRU-VNTRs in Bogotá, Colombia. Infect Genet Evol. 2012;12:657-63. https://doi.org/10.1016/j.meegid.2011.07.006
Realpe T, Correa N, Rozo J, Ferro B, Gómez V, Zapata E, et al. Population structure among Mycobacterium tuberculosis isolates from pulmonary tuberculosis patients in Colombia. PLoS One. 2014;9:1-12. https://doi.org/10.1371/journal.pone.0093848
Beltrán MY. Genotipificación de Mycobacterium tuberculosis en aislados clínicos obtenidos de pacientes VIH positivos de los hospitales Simón Bolívar y Santa Clara de Bogotá (tesis). Bogotá: Universidad Nacional de Colombia; 2016. p. 106.
Castro C, Ricardo A, Zabaleta A, Llerena C, Puerto G. Caracterización de aislamientos clínicos de Mycobacterium tuberculosis obtenidos de individuos positivos para HIV en Colombia, 2012. Biomédica. 2017;37:86-95. https://doi.org/10.7705/biomedica.v37i1.3112
Nieto L, Ferro B, Villegas S, Mehaffy C, Forero L, Moreira C, et al. Characterization of extensively drug-resistant tuberculosis cases from Valle del Cauca, Colombia. J Clin Microbiol. 2012;50:4185-7. https://doi.org/10.1128/JCM.01946-12
Wirth T, Hildebrand F, Allix C, Wölbeling F, Kubica T, Kremer K, et al. Origin, spread and demography of the Mycobacterium tuberculosis complex. PLoS Pathog. 2008;4:1-10. https://doi.org/10.1371/journal.ppat.1000160
Barletta F, Otero L, Collante J, Asto B, de Jong B, Seas C, et al. Genetic variability of Mycobacterium tuberculosis complex in patients with no known risk factors for MDR-TB in the North-Eastern of Lima, Perú . BMC Infect Dis. 2013;13:397. https://doi.org/10.1186/1471-2334-13-397
Lu W, Lu B, Liu Q, Dong H, Shao Y, Jiang Y, et al. Genotypes of Mycobacterium tuberculosis isolates in rural China: Using MIRU-VNTR and spoligotyping methods. Scand J Infect Dis. 2014;26:98-106. https://doi.org/10.3109/00365548.2013.858182
Bidovec-Stojkovič U, Seme K, Žolnir-Dovč M, Supply P. Prospective genotyping of Mycobacterium tuberculosis from fresh clinical samples. PLoS One. 2014;9:e109547. https://doi.org/10.1371/journal.pone.0109547
Algunos artículos similares:
- Claudia Llerena, Santiago Elías Fadul, María Consuelo Garzón, Graciela Mejía, Dora Leticia Orjuela, Luz Mary García, Hilda Beatriz Álvarez, Fernando Javier Ruiz, Resistencia de Mycobacterium tuberculosis a los fármacos antituberculosos en menores de 15 años en Colombia , Biomédica: Vol. 30 Núm. 3 (2010)
- Angie Zabaleta, Claudia Llerena, Serie de casos: tuberculosis extremadamente resistente a drogas en Colombia, 2006-2016 , Biomédica: Vol. 39 Núm. 4 (2019)
- Juan Gabriel Bueno-Sánchez, Jairo René Martínez-Morales, Elena E. Stashenko, Wellman Ribón, Actividad antituberculosa de plantas colombianas , Biomédica: Vol. 29 Núm. 1 (2009)
- María Consuelo Garzón, Dailyn Yorledy Angée, Claudia Llerena, Dora Leticia Orjuela, Jorge Ernesto Victoria, Vigilancia de la resistencia del Mycobacterium tuberculosis a los fármacos antituberculosos, Colombia 2004-2005 , Biomédica: Vol. 28 Núm. 3 (2008)
- Wellman Ribón, Claudia Castro, Liliana González, Juan Carlos Rozo, Gloria Puerto, Evaluación de la bioseguridad del protocolo de extracción de ADN para especies del complejo Mycobacterium tuberculosis implementado en el Instituto Nacional de Salud , Biomédica: Vol. 29 Núm. 4 (2009)
- Leandro Galvis, Ángel Y. Sánchez, Leonardo F. Jurado, Martha I. Murcia, Tuberculosis asociada a antagonistas del factor de necrosis tumoral alfa, presentación de un caso y análisis de los casos reportados en Colombia , Biomédica: Vol. 38 Núm. 1 (2018)
- Diego Chaves, Andrea Sandoval, Luis Rodríguez, Juan C. García, Silvia Restrepo, María Mercedes Zambrano, Análisis comparativo de seis genomas del complejo Mycobacterium tuberculosis , Biomédica: Vol. 30 Núm. 1 (2010)
- Flor de María Cáceres, Luis Carlos Orozco, Incidencia y factores asociados al abandono del tratamiento antituberculoso , Biomédica: Vol. 27 Núm. 4 (2007)
- Diana Castaño, Mauricio Rojas, Alteraciones en el reclutamiento y activación de proteínas Rab durante la infección micobacteriana , Biomédica: Vol. 30 Núm. 2 (2010)
- Dora Orjuela, Gloria Puerto, Graciela Mejía, Claudia Castro, María Consuelo Garzón, Luz Mary García, Elkin Hernández, Wellman Ribón, Gerzaín Rodríguez, Tuberculosis cutánea por mesoterapia, estudio de seis casos , Biomédica: Vol. 30 Núm. 3 (2010)
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |