Correlation between the number of false positive variants and the quality of results using Ion Torrent PGM™ sequencing to screen BRCA genes
Abstract
Introduction: Next Generation Sequencing (NGS) is cost-effective and a faster method to study genes, but its protocol is challenging.
Objective: To analyze different adjustments to the protocol for screening the BRCA genes using Ion Torrent PGM sequencing and correlate the results with the number of false positive (FP) variants.
Material and methods: We conducted a library preparation process and analyzed the number of FP InDels, the library concentration, the number of cycles in the target
amplification step, the purity of the nucleic acid, the input, and the number of samples/Ion 314 chips in association with the results obtained by NGS.
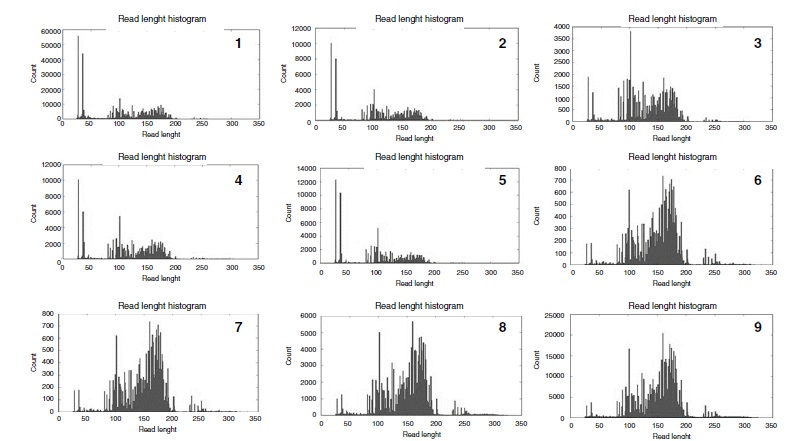
Results: We carried out 51 reactions and nine adjustments of protocols and observed eight FP InDels in homopolymer regions. No FP Single-Nucleotide Polymorphism variant was observed; 67.5% of protocol variables were jointly associated with the quality of the results obtained (p<0.05). The number of FP InDels decreased when the quality of results increased.
Conclusion: The Ion AmpliSeq BRCA1/BRCA2 Community Panel had a better performance using four samples per Ion-314 chip instead of eight and the optimum number of cycles in the amplification step, even when using high-quality DNA, was 23. We observed better results with the manual equalization process and not using the Ion Library Equalizer kit. These adjustments provided a higher coverage of the variants and fewer artifacts (6.7-fold). Laboratories must perform internal validation because FP InDel variants can vary according to the quality of results while the NGS assay should be validated with Sanger.
Downloads
References
Niell BL, Rennert G, Bonner JD, Almog R, Tomsho LP, Gruber SB. BRCA1 and BRCA2 founder mutations and the risk of colorectal cancer. J Natl Cancer Inst. 2004;96:15-21. https://doi.org/10.1093/jnci/djh008
Garber JE, Syngal S. One less thing to worry about: The shrinking spectrum of tumors in BRCA founder mutation carriers. J Natl Cancer Inst. 2004;96:2-310. https://doi.org/1093/jnci/djh021
Wallace AJ. New challenges for BRCA testing: A view from the diagnostic laboratory. Eur J Hum Genet. 2016;24(Suppl.1):S10-18. https://doi.org/10.1038/ejhg.2016.94
Loeffelholz M, Fofanov Y. The main challenges that remain in applying high-throughput sequencing to clinical diagnostics. Expert Rev Mol Diagn. 2015;15:1405-8. https://doi.org/10.1586/14737159.2015.1088385
Germani A, Libi F, Maggi S, Stanzani G, Lombardi A, Pellegrini P, et al. Rapid detection of copy number variations and point mutations in BRCA1/2 genes using a single workflow by ion semiconductor sequencing pipeline. Oncotarget. 2018;9:33648-55. https://doi.org/10.18632/oncotarget.26000
Zanella I, Merola F, Biasiotto G, Archetti S, Spinelli E, Di Lorenzo D. Evaluation of the Ion Torrent PGM sequencing workflow for the routine rapid detection of BRCA1 and BRCA2 germline mutations. Exp Mol Pathol. 2017;102:314-20. https://doi.org/10.1016/j.yexmp.2017.03.001
Oosterwijk JC, de Vries J, Mourits MJ, de Bock GH. Genetic testing and familial implications in breast-ovarian cancer families. Maturitas. 2014;78:252-7. https://doi.org/10.1016/j.maturitas.2014.05.002
Claus EB, Schildkraut JM, Thompson D, Risch NJ.The genetic attributable risk of breast and ovarian cancer. Cancer. 1996;77:2318-24. https://doi.org/10.1002/(SICI)1097-0142(19960601)77:11<2318::AID-CNCR21>3.0.CO;2-Z
Lih CJ, Harrington RD, Sims DJ, Harper KN, Bouk CH, Datta V, et al. Analytical validation of the next-generation sequencing assay for a nationwide signal-finding clinical trial: Molecular analysis for therapy choice clinical trial. J Mol Diagn. 2017;19:313-27. https://doi.org/10.1016/j.jmoldx.2016.10.007
Maistro S, Teixeira N, Encinas G, Katayama ML, Niewiadonski VD, Cabral LG, et al. Germline mutations in BRCA1 and BRCA2 in epithelial ovarian cancer patients in Brazil. BMC Cancer. 2016;16:934. https://doi.org/10.1186/s12885-016-2966-x
Bragg LM, Stone G, Butler MK, Hugenholtz P, Tyson GW. Shining a light on dark sequencing: Characterising errors in Ion Torrent PGM data. PLoS Comput Biol. 2013;9:e1003031. https://doi.org/10.1371/journal.pcbi.1003031
Kang HP, Maguire JR, Chu CS, Haque IS, Lai H, Mar-Heyming R, et al. Design and validation of a next generation sequencing assay for hereditary BRCA1 and BRCA2 mutation testing. Peer J. 2016;4:e2162. https://doi.org/10.7717/peerj.2162
Park J, Jang W, Chae H, Kim Y, Chi HY, Kim M. Comparison of targeted next-generation and Sanger sequencing for the BRCA1 and BRCA2 mutation screening. Ann Lab Med. 2016;36:197-201. https://doi.org/201603197
Mehta A, Vasudevan S, Sharma SK, Kumar D, Panigrahi M, Suryavanshi M, et al. Germline BRCA1 and BRCA2 deleterious mutations and variants of unknown clinical significance associated with breast/ovarian cancer: A report from North India. Cancer Manag Res. 2018;10:6505-16. https://doi.org/10.2147/CMAR.S186563
Rehm HL, Bale SJ, Bayrak-Toydemir P, Berg JS, Brown KK, Deignan JL, et al. ACMG clinical laboratory standards for next-generation sequencing. Genet Med. 2013;15:733-47. https://doi.org/10.1038/gim.2013.92
Trujillano D, Weiss ME, Schneider J, Koster J, Papachristos EB, Saviouk V, et al. Nextgeneration sequencing of the BRCA1 and BRCA2 genes for the genetic diagnostics of hereditary breast and/or ovarian cancer. J Mol Diagn. 2015;17:162-70. https://doi.org/10.1016/j.jmoldx.2014.11.004
Sanger F, Nicklen S, Coulson AR. DNA sequencing with chain-terminating inhibitors. Proc Natl Acad Sci USA. 1977;74:5463-7. https://doi.org/10.1073/pnas.74.12.5463
Vendrell JA, Vilquin P, Larrieux M, van Goethem C, Solassol J. Benchmarking of ampliconbased next-generation sequencing panels combined with bioinformatics solutions for germline BRCA1 and BRCA2 alteration detection. J Mol Diagn. 2018;20:754-64. https://doi.org/S1525-1578(18)30072-2 19
Jennings LJ, Arcila ME, Corless C, Kamel-Reid S, Lubin IM, Pfeifer J, et al. Guidelines for validation of next-generation sequencing-based oncology panels: A joint consensus recommendation of the Association for Molecular Pathology and College of American Pathologists. J Mol Diagn. 2017;19:341-65. https://doi.org/S1525-1578(17)30025-9
Beck TF, Mullikin JC, Program NCS, Biesecker LG. Systematic evaluation of Sanger validation of next-generation sequencing variants. Clin Chem. 2016;62:647-54. https://doi.org/10.1373/clinchem.2015.249623
Buzolin AL, Moreira CM, Sacramento PR, Oku AY, Fornari A, Antonio DSM, et al. Development and validation of a variant detection workflow for BRCA1 and BRCA2 genes and its clinical application based on the Ion Torrent technology. Hum Genomics. 2017;11:14. https://doi.org/10.1186/s40246-017-0110-x
Harismendy O, Ng PC, Strausberg RL, Wang X, Stockwell TB, Beeson KY, et al. Evaluation of next generation sequencing platforms for population targeted sequencing studies. Genome Biol. 2009;10:R32. https://doi.org/10.1186/gb-2009-10-3-r32
Strom CM, Rivera S, Elzinga C, Angeloni T, Rosenthal SH, Goos-Root D, et al. Development and validation of a next-generation sequencing assay for BRCA1 and BRCA2 variants for the clinical laboratory. PLoS ONE. 2015;10:e0136419. https://doi.org/10.1371/journal.pone.0136419
Bijwaard K, Dickey JS, Kelm K, Tezak Z. The first FDA marketing authorizations of next generation sequencing technology and tests: Challenges, solutions and impact for future assays. Expert Rev Mol Diagn. 2015;15:33-40. https://doi.org/10.1586/14737159.2015.979795
Gargis AS, Kalman L, Berry MW, Bick DP, Dimmock DP, Hambuch T, et al. Assuring the quality of next-generation sequencing in clinical laboratory practice. Nat Biotechnol. 2012;30:1033-6. https://doi.org/10.1038/nbt.2403
Some similar items:
- Ingrid Yamile Pulido, José Ramón Mantilla, Emilia María Valenzuela, María Teresa Reguero, Elsa Beatriz González, Distribution of extended spectrum β-lactamases-codifying genes in Klebsiella pneumoniae isolates from hospitals of Bogota, D.C., Colombia , Biomedica: Vol. 31 No. 1 (2011)
- Wellman Ribón, Claudia Castro, Liliana González, Juan Carlos Rozo, Gloria Puerto, Biosafety evaluation of the DNA extraction protocol for Mycobacterium tuberculosis complex species, as implemented at the Instituto Nacional de Salud, Colombia , Biomedica: Vol. 29 No. 4 (2009)
- Brian Alejandro Suárez, Claudia Liliana Cuervo, Concepción Judith Puerta, The intergenic region of the histone h2a gene supports two major lineages of Trypanosoma rangeli , Biomedica: Vol. 27 No. 3 (2007)
- Rafael José Vivero, Maria Angélica Contreras-Gutiérrez, Eduar Elías Bejarano, Analysis of the primary and secondary structure of the mitochondrial serine transfer RNA in seven species of Lutzomyia , Biomedica: Vol. 27 No. 3 (2007)
- Diana Carolina López, Carlos Jaramillo, Felipe Guhl, Population structure and genetic variability of Rhodnius prolixus (Hemiptera: Reduviidae) from different geographic areas of Colombia. , Biomedica: Vol. 27 No. 1esp (2007): Enfermedad de Chagas
- Sandy Milena Caldera, María Cristina Jaramillo, Suljey Cochero, Alveiro Pérez-Doria, Eduar Elías Bejarano, Genetic differences between populations of Aedes aegypti from municipalities in northern Colombia, with high and low dengue incidence , Biomedica: Vol. 33 (2013): Suplemento 1, Fiebres hemorrágicas
- Leonardo F. Jurado, Martha I. Murcia, Patricia Hidalgo, John E. Leguizamón, Lorena R. González, Phenotypic and genotypic diagnosis of bone and miliary tuberculosis in an HIV+ patient in Bogotá, Colombia , Biomedica: Vol. 35 No. 1 (2015)
- Johana Rueda, Teresa Realpe, Gloria Mejía, Elsa Zapata, Jaime Robledo, GenoType MTBDRplus 1.0® for the detection of cross-resistance between isoniazide and ethionamide in isolates of multidrug-resistant Mycobacterium tuberculosis , Biomedica: Vol. 35 No. 4 (2015)
- Andrea Casas-Vargas, Liza M. Romero, William Usaquén, Sara Zea, Margarita Silva, Ignacio Briceño, Alberto Gómez, José Vicente Rodríguez, Mitochondrial DNA diversity in prehispanic bone remains on the eastern Colombian Andes , Biomedica: Vol. 37 No. 4 (2017)
- Adrián Peñata, Richard Salazar, Tatiana Castaño, Julián Bustamante, Sigifredo Ospina, Molecular diagnosis of extrapulmonary tuberculosis and sensitivity to rifampicin with an automated real-time method , Biomedica: Vol. 36 (2016): Suplemento 1, Microbiología médica
| Article metrics | |
|---|---|
| Abstract views | |
| Galley vies | |
| PDF Views | |
| HTML views | |
| Other views | |