Filogenia y resistencia de cepas de Escherichia coli productoras de betalactamasas de espectro extendido a los antibióticos en pacientes con cáncer hospitalizados en Perú
Resumen
Introducción. Las infecciones asociadas con la atención en salud constituyen un problema de salud pública porque aumentan la morbimortalidad de los pacientes, sobre todo de aquellos con factores de riesgo, como la inmunosupresión debida a enfermedades oncológicas. Es importante conocer la diversidad genética de los
principales microorganimos causantes de infecciones hospitalarias mediante la vigilancia epidemiológica tradicional y la epidemiología molecular, para hacer un mejor seguimiento y detectar brotes tempranamente.
Objetivo. Determinar el grupo filogenético y la resistencia a antibióticos de las cepas de Escherichia coli aisladas de pacientes con cáncer hospitalizados.
Materiales y métodos. Se hizo un estudio de tipo transversal que incluyó 67 cepas de Escherichia coli productoras de betalactamasas de espectro extendido (BLEE). Se determinó el grupo filogenético, el perfil de resistencia a los antibióticos, los genes de resistencia a betalactámicos, el tipo de las muestras y los servicios de hospitalización de donde fueron recuperadas.
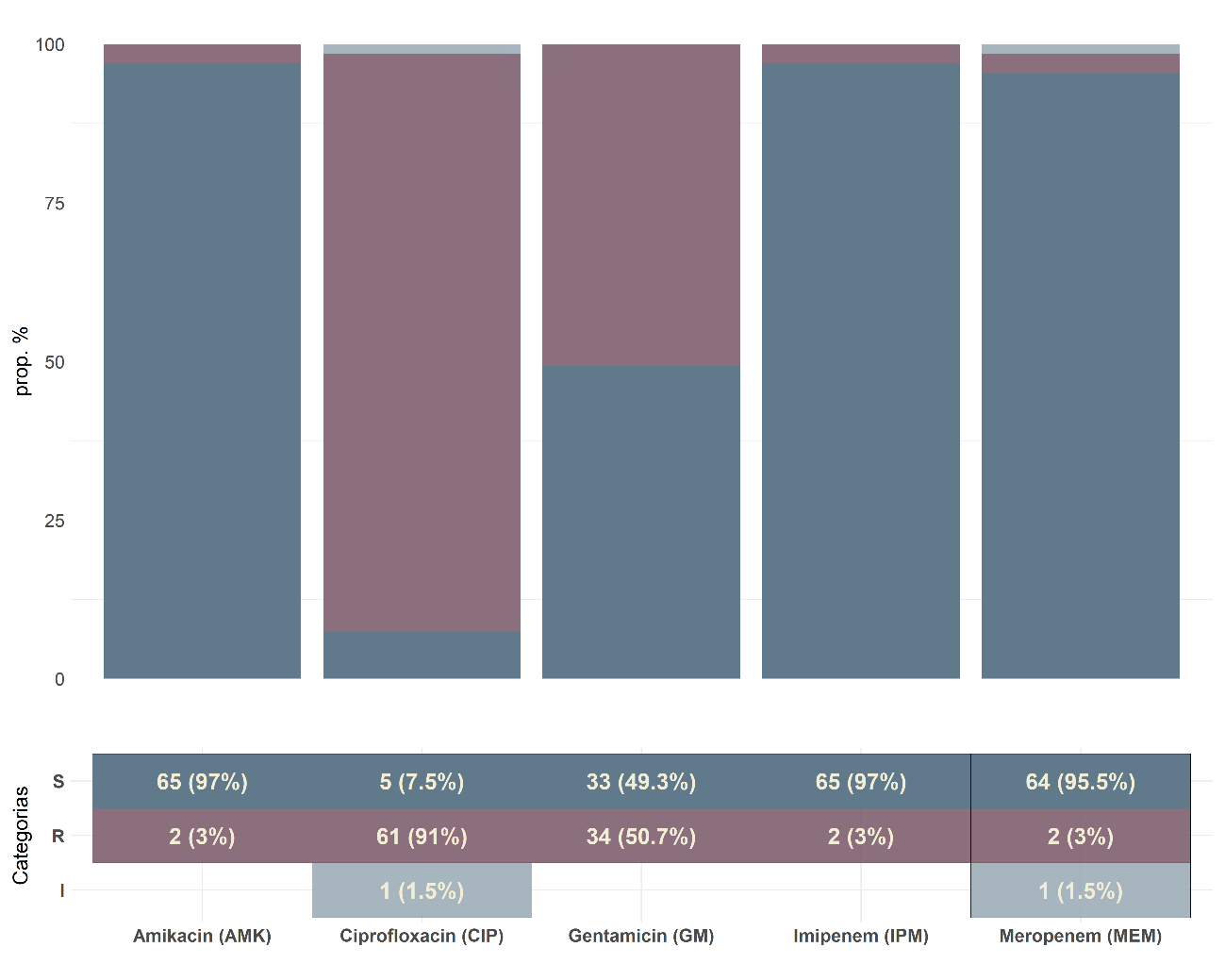
Resultados. El grupo filogenético más frecuente fue el B2 (36 %). El 57 % de las cepas B2 fueron aisladas de muestras de orina y el 33 % provenía del servicio de urología. La resistencia a ciprofloxacino y gentamicina fue de 91 y 53 %, respectivamente, y el 79 % de las cepas tenía el gen blaCTX-M. Se encontró una relación significativa (p<0,05) entre los grupos filogenéticos y la resistencia a ciprofloxacina, así como a la edad del paciente.
Conclusión. El filogrupo de E. coli predominante fue el B2. Se evidenció una gran resistencia a ciprofloxacina y gentamicina, una proporción elevada de cepas BLEE con el blaCTX-M, y una relación entre el grupo filogenético y la resistencia a ciprofloxacino.
Descargas
Referencias bibliográficas
Organización Panamericana de la Salud. Vigilancia epidemiológica de las infecciones asociadas a la atención de la salud en neonatología. Módulo IV - PAHO; 2013. Fecha de consulta: 18 de febrero de 2022. Disponible en: https://iris.paho.org/handle/10665.2/31361
Yagui-Moscoso M, Vidal-Anzardo M, Rojas-Mezarina L, Sanabria-Rojas H. Prevención de infecciones asociadas a la atención de salud: conocimientos y prácticas en médicos residentes. An Fac Med. 2021;82:131-9. https://doi.org/10.15381/anales.v82i2.19839
Johnson JR, Russo TA. Molecular epidemiology of extraintestinal pathogenic Escherichia coli. EcoSal Plus. 2018;8:1-32. https://doi.org/10.1128/ecosalplus.ESP-0004-2017
Bonilla-Marciales AP, Chávez-Cañas WO, Hernández-Mogollón RA, Ramón-Jaimes NA. Estrategias de prevención y control de las infecciones en pacientes oncológicos. MedUNAB. 2019;22:356-68.
Velázquez-Brizuela IE, Aranda-Gama J, Camacho-Cortés JL, Ortiz GG. Epidemiología de infecciones nosocomiales en el Instituto Jalisciense de Cancerología. Rev Cuba Salud Pública. 2013;39:19-31.
Clermont O, Christenson JK, Denamur E, Gordon DM. The Clermont Escherichia coli phylo-typing method revisited: Improvement of specificity and detection of new phylo-groups. Environ Microbiol Rep. 2013;5:58-65. https://doi.org/10.1111/1758-2229.12019
Gonzáles-Rodríguez AO, Pastor HJ, Villafuerte CA, Barrón YL de, Miranda DV de, Cunza SS. Clasificación filogenética de Escherichia coli uropatógena y respuesta inmunometabólica en adultos mayores con infección urinaria en casas de reposo. Arch Med Col. 2019;19:238-46. https://doi.org/10.30554/archmed.19.2.3334.2019
Nicolas-Chanoine MH, Bertrand X, Madec JY. Escherichia coli ST131, an intriguing clonal group. Clin Microbiol Rev. 2014;27:543-74. https://doi.org/10.1128/CMR.00125-13
CLSI. M100Ed27 - Performance standards for antimicrobial susceptibility testing. 32nd edition. Fecha de consulta: 21 de febrero de 2022. Disponible en: https://clsi.org/standards/products/microbiology/documents/m100/
Ghasemi Y, Archin T, Kargar M, Mohkam M. A simple multiplex PCR for assessing prevalence of extended-spectrum β-lactamases producing Klebsiella pneumoniae in Intensive Care Units of a referral hospital in Shiraz, Iran. Asian Pac J Trop Med. 2013;6:703-8. https://doi.org/10.1016/S1995-7645(13)60122-4
Chávez-Hidalgo DC. Frecuencia y subtipos del gen blaCTX-M en enterobacterias productoras de BLEE aisladas de urocultivos en el Instituto Nacional de Enfermedades Neoplásicas de enero a diciembre del 2017 [tesis]. Lima: Universidad Nacional Mayor San Marcos; 2019.
Tejada-Llacsa PJ, Huarcaya JM, Melgarejo GC, Gonzáles LF, Cahuana J, Pari RM, et al. Caracterización de infecciones por bacterias productoras de BLEE en un hospital de referencia nacional. An Fac Med. 2015;76:161-6. https://doi.org/10.15381/anales.v76i2.11143
Sader HS, Jones RN, Andrade-Baiocchi S, Biedenbach DJ, SENTRY Participants Group (Latin America). Four-year evaluation of frequency of occurrence and antimicrobial susceptibility patterns of bacteria from bloodstream infections in Latin American medical centers. Diagn Microbiol Infect Dis. 2002;44:273-80. https://doi.org/10.1016/s0732-8893(02)00469-8
Koirala S, Khadka S, Sapkota S, Sharma S, Khanal S, Thapa A, et al. Prevalence of CTX-M β-lactamases producing multidrug resistant Escherichia coli and Klebsiella pneumoniae among patients attending Bir Hospital, Nepal. BioMed Res Int. 2021:9958294. https://doi.org/10.1155/2021/9958294
Dirar MH, Bilal NE, Ibrahim ME, Hamid ME. Prevalence of extended-spectrum β-lactamase (ESBL) and molecular detection of blaTEM, blaSHV and blaCTX-M genotypes among Enterobacteriaceae isolates from patients in Khartoum, Sudan. Pan Afr Med J. 2020;37:213. https://doi.org/10.11604/pamj.2020.37.213.24988
Demirci M, Ünlü Ö, İstanbullu Tosun A. Detection of O25b-ST131 clone, CTX-M-1 and CTX-M-15 genes via real-time PCR in Escherichia coli strains in patients with UTIs obtained from a university hospital in Istanbul. J Infect Public Health. 2019;12:640-4. https://doi.org/10.1016/j.jiph.2019.02.017
Hernández E, Araque M, Millán Y, Millán B. Prevalencia de b-lactamasa CTX-M-15 en grupos filogenéticos de Escherichia coli uropatógena aisladas en pacientes de la comunidad en Mérida, Venezuela. Invest Clin. 2014;55:13.
Galindo-Méndez M. Caracterización molecular y patrón de susceptibilidad antimicrobiana de Escherichia coli productora de β-lactamasas de espectro extendido en infección del tracto urinario adquirida en la comunidad. Rev Chil Infectol. 2018;35:29-35. https://doi.org/10.4067/s0716-10182018000100029
Gonzáles E, Patiño L, Ore E, Martínez V, Moreno S, Cruzado NB, et al. β-lactamasas de espectro extendido tipo CTX-M en aislamientos clínicos de Escherichia coli y Klebsiella pneumoniae en el Instituto Nacional de Salud del Niño-Breña, Lima, Perú. Rev Médica Hered. 2019;30:242-8. https://doi.org/10.20453/rmh.v30i4.3659
Alsharapy SA, Yanat B, Lopez-Cerero L, Nasher SS, Díaz-De-Alba P, Pascual Á, et al. Prevalence of ST131 clone producing both ESBL CTX-M-15 and AAC(6’)Ib-cr among ciprofloxacin-resistant Escherichia coli isolates from Yemen. Microb Drug Resist Larchmt N. 2018;24:1537-42. https://doi.org/10.1089/mdr.2018.0024
Millán Y, Araque M, Ramírez A. Distribución de grupos filogenéticos, factores de virulencia y susceptibilidad antimicrobiana en cepas de Escherichia coli uropatógena. Rev Chil Infectol. 2020;37:117-23. https://doi.org/10.4067/s0716-10182020000200117
Cole BK, Ilikj M, McCloskey CB, Chávez-Bueno S. Antibiotic resistance and molecular characterization of bacteremia Escherichia coli isolates from newborns in the United States. PLoS ONE. 2019;14:e0219352. https://doi.org/10.1371/journal.pone.0219352
Ranjbar R, Nazari S, Farahani O. Phylogenetic analysis and antimicrobial resistance profiles of Escherichia coli strains isolated from UTI-suspected patients. Iran J Public Health. 2020;49:1743-9. https://doi.org/10.18502/ijph.v49i9.4094
Tenaillon O, Skurnik D, Picard B, Denamur E. The population genetics of commensal Escherichia coli. Nat Rev Microbiol. 2010;8:207-17. https://doi.org/10.1038/nrmicro2298
Tsai W-L, Hung C-H, Chen H-A, Wang J-L, Huang I-F, Chiou Y-H, et al. Extended-spectrum β-lactamase-producing Escherichia coli bacteremia: Comparison of pediatric and adult populations. J Microbiol Immunol Infect. 2018;51:723-31. https://doi.org/10.1016/j.jmii.2017.08.005
Algunos artículos similares:
- Yasmin Varela, Beatriz Millán, María Araque, Diversidad genética de cepas extraintestinales de Escherichia coli productoras de las betalactamasas TEM, SHV y CTX-M asociadas a la atención en salud , Biomédica: Vol. 37 Núm. 2 (2017)
- María Fernanda Gutiérrez, Sandra Moreno, Mónica Viviana Alvarado, Andrea Bermúdez, Análisis filogenético de las cepas de rotavirus y virus de la hepatitis A encontradas en agua de consumo en el municipio de Quibdó, Chocó , Biomédica: Vol. 29 Núm. 2 (2009)
- Brian Alejandro Suárez, Claudia Liliana Cuervo, Concepción Judith Puerta, La región intergénica del gen H2A apoya las subpoblaciones KP1(-) y KP1(+) de Trypanosoma rangeli , Biomédica: Vol. 27 Núm. 3 (2007)
- Nadia Yadira Castañeda, Jacqueline Chaparro-Olaya, Jaime E. Castellanos, Producción y caracterización de un anticuerpo policlonal dirigido contra la fosfoproteína del virus de la rabia , Biomédica: Vol. 27 Núm. 2 (2007)
- Manuel Muñoz, Juan Carlos Navarro, Virus Mayaro: un arbovirus reemergente en Venezuela y Latinoamérica , Biomédica: Vol. 32 Núm. 2 (2012)
- Andrés Páez, Constanza Nuñez, Clemencia García, Jorge Boshell, Epidemiología molecular de epizootias de rabia en Colombia, 1994-2002: evidencia de rabia humana y canina asociada a quirópteros. , Biomédica: Vol. 23 Núm. 1 (2003)
- Matilde Anaya, Eduardo Barbará, Jesús Padrón, Sofía F. Borrego, Oderlaise Valdés, Alian Molina, Influencia del campo magnético sobre el crecimiento de microorganismos patógenos ambientales aislados en el Archivo Nacional de la República de Cuba , Biomédica: Vol. 35 Núm. 3 (2015)
- Elvia Michelli, Adriana Millán, Hectorina Rodulfo, Mirian Michelli, Jesús Luiggi, Numirin Carreño, Marcos de Donato, Identificación de Escherichia coli enteropatógena en niños con síndrome diarreico agudo del Estado Sucre, Venezue , Biomédica: Vol. 36 (2016): Suplemento 1, Microbiología médica
- Paula Andrea Báez, Carlos Mario Jaramillo, Lina Arismendi, Julio Cesar Rendón, Fabián Cortés-Mancera, Dioselina Peláez, María Mercedes González, Francisco Molina, María Cristina Navas, Evidencia de circulación del virus de la hepatitis A, subgenotipo IA, en muestras ambientales en Antioquia, Colombia , Biomédica: Vol. 36 (2016): Suplemento 2, Enfermedades virales
- Mónica Tatiana Herrera, Jhon Jhamilton Artunduaga, Claudia Cristina Ortiz, Rodrigo Gonzalo Torres, Síntesis de nanopartículas de ácido poli-láctico cargadas con antibióticos y su actividad antibacteriana contra Escherichia coli O157:H7 y Staphylococcus aureus resistente a meticilina , Biomédica: Vol. 37 Núm. 1 (2017)
Derechos de autor 2022 Biomédica

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |