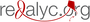Caracterización de β-lactamasas de espectro extendido en aislamientos clínicos colombianos de Salmonella enterica no tifoidea de 1997 a 2022
Resumen
Introducción. Salmonella spp. es un agente patógeno zoonótico transmitido al humano por el agua o los alimentos contaminados. La presencia de β-lactamasas de espectro extendido es un creciente problema para la salud pública debido a que estas enzimas confieren resistencia contra las cefalosporinas de tercera y cuarta generación.
Objetivo. Caracterizar las β-lactamasas de espectro extendido en aislamientos de Salmonella spp. recibidos por el programa de vigilancia de enfermedad diarreica aguda o enfermedad transmitida por alimentos del Grupo de Microbiología del Instituto Nacional de Salud.
Materiales y métodos. Entre enero de 1997 y junio de 2022, se recibieron 444 aislamientos de Salmonella spp. resistentes, por lo menos, a una de las cefalosporinas de tercera generación. El fenotipo de las β-lactamasas de espectro extendido se identificó con la prueba de doble disco. El ADN se extrajo por ebullición y mediante PCR se amplificaron los genes blaCTX-M, blaSHV y blaTEM.
Resultados. Todos los aislamientos fueron positivos para la prueba de β-lactamasas de espectro extendido. Los resultados de la amplificación por PCR fueron: blaCTX-M + blaTEM (n=200), blaCTX-M (n=177), blaSHV (n=16), blaSHV + blaCTX-M (n=6), blaTEM (n=13) y blaSHV + blaCTX-M + blaTEM (n=3). Del total, 26 aislamientos fueron negativos para los genes evaluados. Los aislamientos positivos para β-lactamasas de espectro extendido se identificaron en Bogotá y en 21 departamentos: Chocó, Magdalena, Meta, Bolívar, Casanare, Cesar, Córdoba, Quindío, Atlántico, Tolima, Cauca, Cundinamarca, Huila, Boyacá, Caldas, Norte de Santander, Risaralda, Antioquia, Nariño, Santander y Valle del Cauca.
Conclusión. La resistencia a las cefalosporinas de tercera generación en aislamientos de Salmonella spp. fue generada principalmente por blaCTX-M. El 44 % (197/444) de los aislamientos presentó resistencia a ampicilina, tetraciclina, cloranfenicol y trimetoprimsulfametoxazol Los serotipos portadores de β-lactamasas de espectro extendido más frecuentes fueron S. Typhimurium y S. Infantis.
Descargas
Referencias bibliográficas
Majowicz SE, Musto J, Scallan E, Angulo FJ, Kirk M, O'Brien SJ, et al. International collaboration on enteric disease 'Burden of Illness' studies. The global burden of nontyphoidal Salmonella gastroenteritis. Clin Infect Dis. 2010;50:882-9.https://doi.org/10.1086/650733
Balasubramanian R, Im J, Lee JS, Jeon HJ, Mogeni OD, Kim JH, et al. The global burden and epidemiology of invasive non-typhoidal Salmonella infections. Hum Vaccin Immunother. 2019;15:1421-6. https://doi.org/10.1080/21645515.2018.1504717
Tack DM, Ray L, Griffin PM, Cieslak PR, Dunn J, Rissman T, et al. Preliminary incidence and trends of infections with pathogens transmitted commonly through food - Foodborne Diseases Active Surveillance Network, 10 U.S. Sites, 2016-2019. MMWR Morb Mortal Wkly Rep. 2020;69:509-14. https://doi.org/10.15585/mmwr.mm6917a1
Gal-Mor O, Boyle EC, Grassl GA. Same species, different diseases: how and why typhoidal and non-typhoidal Salmonella enterica serovars differ. Front Microbiol. 2014;5:391. https://doi.org/10.3389/fmicb.2014.00391
Centers for Disease Control and Prevention. Antibiotic resistance threats in the United States, 2019. Fecha de consulta: 17 de noviembre de 2022. Disponible en: https://doi.org/10.15620/cdc:82532
Hohmann EL. Nontyphoidal salmonellosis. Clin Infect Dis. 2001;32:263-9. https://doi.org/10.1086/318457
Bush K. Past and present perspectives on beta-lactamases. Antimicrob Agents Chemother. 2018;62:1-20. https://doi.org/10.1128/AAC.01076-18
Franco A, Leekitcharoenphon P, Feltrin F, Alba P, Cordaro G, Lurescia M, et al. Emergence of a clonal lineage of multidrug-resistant ESBL-producing Salmonella Infantis transmitted from broilers and broiler meat to humans in Italy between 2011 and 2014. PLoS ONE. 2015;10:e0144802. https://doi.org/10.1371/journal.pone.0144802
Wei Z, Xu X, Yan M, Chang H, Li Y, Kan B, et al. Salmonella typhimurium and Salmonella enteritidis infections in sporadic diarrhea in children: source tracing and resistance to third-generation cephalosporins and ciprofloxacin. Foodborne Pathog Dis. 2019;16:244-55. https://doi.org/10.1089/fpd.2018.2557
Canton R, González-Alba JM, Galán JC. CTX-M enzymes: Origin and diffusion. Front Microbiol. 2012;3:110. https://doi.org/10.3389/fmicb.2012.00110
Brown AC, Chen JC, Watkins LKF, Campbell D, Folster JP, Tate H, et al. CTX-M-65 extended-spectrum beta-lactamase-producing Salmonella enterica serotype Infantis, United States. Emerg Infect Dis. 2018;24:2284-91. https://doi.org/10.3201/eid2412.180500
Moura Q, Fernandes MR, Silva KC, Monte DF, Esposito F, Dropa M, et al. Virulent nontyphoidal Salmonella producing CTX-M and CMY-2 beta-lactamases from livestock, food and human infection, Brazil. Virulence. 2018;9:281-6. https://doi.org/10.1080/21505594.2017.1279779
Zhang CZ, Ding XM, Lin XL, Sun RY, Lu YW, Cai RM, et al. The emergence of chromosomally located blaCTX-M-55 in Salmonella from foodborne animals in China. Front Microbiol. 2019;10:1268. https://doi.org/10.3389/fmicb.2019.01268
Okoro CK, Barquist L, Connor TR, Harris SR, Clare S, Stevens MP, et al. Signatures of adaptation in human invasive Salmonella Typhimurium ST313 populations from sub- Saharan Africa. PLoS Negl Trop Dis. 2015;9:e0003611. https://doi.org/10.1371/journal.pntd.0003611
World Health Organization. Global antimicrobial resistance surveillance system (GLASS) report: Early implementation 2020. Fecha de consulta: 27 de diciembre de 2022. Disponible en: https://apps.who.int/iris/handle/10665/332081
Collaborators GBDN-TSID. The global burden of non-typhoidal Salmonella invasive disease: A systematic analysis for the Global Burden of Disease Study 2017. Lancet Infect Dis. 2019;19:1312-24. https://doi.org/10.1016/S1473-3099(19)30418-9
Liebana E, Carattoli A, Coque TM, Hasman H, Magiorakos AP, Mevius D, et al. Public health risks of enterobacterial isolates producing extended-spectrum beta-lactamases or AmpC beta-lactamases in food and food-producing animals: An EU perspective of epidemiology, analytical methods, risk factors, and control options. Clin Infect Dis. 2013;56:1030-7. https://doi.org/10.1093/cid/cis1043
van Boeckel TP, Brower C, Gilbert M, Grenfell BT, Levin SA, Robinson TP, et al. Global trends in antimicrobial use in food animals. Proc Natl Acad Sci U S A. 2015;112:5649-54. https://doi.org/10.1073/pnas.1503141112
World Health Organization. Antigenic formulae of the Salmonella serovars. Fecha de consulta: 20 de marzo de 2022. Disponible en: https://www.pasteur.fr/sites/default/files/veng_0.pdf
Clinical and Laboratory Standards Institute. Performance standards for antimicrobial susceptibility testing. CLSI supplement M100. Wayne P: CLSI; 2020.
Monstein HJ, Ostholm-Balkhed A, Nilsson MV, Nilsson M, Dornbusch K, Nilsson LE. Multiplex PCR amplification assay for the detection of blaSHV, blaTEM and blaCTX-M genes in Enterobacteriaceae. APMIS. 2007;115:1400-8. https://doi.org/10.1111/j.1600-0463.2007.00722.x
Instituto Nacional de Salud Colombia. Vigilancia por laboratorio de Salmonella. 2021. Fecha de consulta: 4 de enero de 2023. Disponible en: https://www.ins.gov.co/BibliotecaDigital/vigilancia-por-laboratorio-de-salmonella-no-tifoidea-en-colombia-2021.pdf
European Food Safety Authority and European Centre for Disease Prevention and Control. The European Union summary report on antimicrobial resistance in zoonotic and indicator bacteria from humans, animals and food in 2018/2019. EFSA J. 2021;19:6490. Fecha de consulta: 7 de octubre de 2023. Disponible en: https://doi.org/10.2903/j.efsa.2021.6490
Livermore DM. Beta-lactamase-mediated resistance and opportunities for its control. J Antimicrob Chemother. 1998;41(Suppl.D):25-41. https://doi.org/10.1093/jac/41.suppl_4.25
Eller C, Simon S, Miller T, Frick JS, Prager R, Rabsch W, et al. Presence of beta-lactamases in extended-spectrum-cephalosporin-resistant Salmonella enterica of 30 different serovars in Germany 2005-11. J Antimicrob Chemother. 2013;68:1978-81. https://doi.org/10.1093/jac/dkt163
Kola A, Kohler C, Pfeifer Y, Schwab F, Kuhn K, Schulz K, et al. High prevalence of extendedspectrum- beta-lactamase-producing Enterobacteriaceae in organic and conventional retail chicken meat, Germany. J Antimicrob Chemother. 2012;67:2631-4. https://doi.org/10.1093/jac/dks295
Rodríguez I, Barownick W, Helmuth R, Mendoza MC, Rodicio MR, Schroeter A, et al. Extended-spectrum beta-lactamases and AmpC beta-lactamases in ceftiofur-resistant Salmonella enterica isolates from food and livestock obtained in Germany during 2003-07. J Antimicrob Chemother. 2009;64:301-9. https://doi.org/10.1093/jac/dkp195
Carattoli A. Plasmids and the spread of resistance. Int J Med Microbiol. 2013;303:298-304. https://doi.org/10.1016/j.ijmm.2013.02.001
Rozwandowicz M, Brouwer MSM, Fischer J, Wagenaar JA, González-Zorn B, Guerra B, et al. Plasmids carrying antimicrobial resistance genes in Enterobacteriaceae. J Antimicrob Chemother. 2018;73:1121-37. https://doi.org/10.1093/jac/dkx488
Coipan CE, Westrell T, van Hoek A, Alm E, Kotila S, Berbers B, et al. Genomic epidemiology of emerging ESBL-producing Salmonella Kentucky bla CTX-M-14b in Europe. Emerg Microbes Infect. 2020;9:2124-35. https://doi.org/10.1080/22221751.2020.1821582
Aviv G, Tsyba K, Steck N, Salmon-Divon M, Cornelius A, Rahav G, et al. A unique megaplasmid contributes to stress tolerance and pathogenicity of an emergent Salmonella enterica serovar Infantis strain. Environ Microbiol. 2014;16:977-94. https://doi.org/10.1111/1462-2920.12351
Hancock SJ, Phan MD, Peters KM, Forde BM, Chong TM, Yin WF, et al. Identification of IncA/C plasmid replication and maintenance genes and development of a plasmid multilocus sequence typing scheme. Antimicrob Agents Chemother. 2017;61. https://doi.org/10.1128/AAC.01740-16
Tate H, Folster JP, Hsu CH, Chen J, Hoffmann M, Li C, et al. Comparative analysis of extended-spectrum-beta-lactamase CTX-M-65-producing Salmonella enterica serovar Infantis isolates from humans, food animals, and retail chickens in the United States. Antimicrob Agents Chemother. 2017;61. https://doi.org/10.1128/AAC.00488-17
Cohen E, Rahav G, Gal-Mor O. Genome sequence of an emerging Salmonella enterica serovar Infantis and genomic comparison with other S. Infantis strains. Genome Biol Evol. 2020;12:151-9. https://doi.org/10.1093/gbe/evaa048
Pietsch M, Simon S, Meinen A, Trost E, Banerji S, Pfeifer Y, et al. Third generation cephalosporin resistance in clinical non-typhoidal Salmonella enterica in Germany and emergence of blaCTX-M-harbouring pESI plasmids. Microb Genom. 2021;7. https://doi.org/10.1099/mgen.0.000698
Gymoese P, Kiil K, Torpdahl M, Osterlund MT, Sorensen G, Olsen JE, et al. WGS based study of the population structure of Salmonella enterica serovar Infantis. BMC Genomics. 2019;20:870. https://doi.org/10.1186/s12864-019-6260-6
Sun H, Wan Y, Du P, Bai L. The epidemiology of monophasic Salmonella Typhimurium. Foodborne Pathog Dis. 2020;17:87-97. https://doi.org/10.1089/fpd.2019.2676
European Food Safety Authority and European Centre for Disease Prevention and Control. The European Union One Health 2019 Zoonoses Report. EFSA J. 2021;19:6406. Fecha de consulta: 20 de diciembre de 2022. Disponible en: https://doi.org/10.2903/j.efsa.2021.6406
Bevan ER, Jones AM, Hawkey PM. Global epidemiology of CTX-M beta-lactamases: Temporal and geographical shifts in genotype. J Antimicrob Chemother. 2017;72:2145-55. https://doi.org/10.1093/jac/dkx146
Wang W, Zhao L, Hu Y, Dottorini T, Fanning S, Xu J, et al. Epidemiological study on prevalence, serovar diversity, multidrug resistance, and CTX-M-type extended-spectrum beta-lactamases of Salmonella spp. from patients with diarrhea, food of animal origin, and pets in several provinces of China. Antimicrob Agents Chemother. 2020;64. https://doi.org/10.1128/AAC.00092-20
M'Ikanatha NM, Yin X, Boktor SW, Dettinger LA, Tewari D. Integrated surveillance for antimicrobial resistance in Salmonella from clinical and retail meat sources reveals genetically related isolates harboring quinolone- and ceftriaxone-resistant determinants. Open Forum Infect Dis. 2021;8:ofab213. https://doi.org/10.1093/ofid/ofab213
Bush K, Bradford PA. Epidemiology of beta-lactamase-reducing pathogens. Clin Microbiol Rev. 2020;33. https://doi.org/10.1128/CMR.00047-19
The White House. National action plan for combating antibiotic-resistant bacteria. Fecha de consulta: 19 de noviembre de 2022. Disponible en: https://obamawhitehouse.archives.gov/sites/default/files/docs/
Karp BE, Tate H, Plumblee JR, Dessai U, Whichard JM, Thacker EL, et al. National Antimicrobial Resistance Monitoring System: Two decades of advancing public health through integrated surveillance of antimicrobial resistance. Foodborne Pathog Dis. 2017;14:545-57. https://doi.org/10.1089/fpd.2017.2283
US Food and Drug Administration. New animal drugs; cephalosporin drugs; extralabel animal drug use; order of prohibition. 21 CFR part 530 (Docket No. FDA–2008–N–0326). Federal Register. 2012;77:736-8. Fecha de consulta: 19 de diciembre de 2022. Disponible en: https://www.federalregister.gov/documents/2012/01/06/2012-35/new-animal-drugscephalosporin-drugs-extralabel-animal-drug-use-order-of-prohibition
Castellanos LR, van der Graaf-van Bloois L, Donado-Godoy P, León M, Clavijo V, Arévalo A, et al. Genomic characterization of extended-spectrum cephalosporin-resistant Salmonella enterica in the Colombian poultry chain. Front Microbiol. 2018;9:2431. https://doi.org/10.3389/fmicb.2018.02431
Shane AL, Mody RK, Crump JA, Tarr PI, Steiner TS, Kotloff K, et al. 2017 Infectious Diseases Society of America Clinical Practice Guidelines for the diagnosis and management of infectious diarrhea. Clin Infect Dis. 2017;65:e45-e80. https://doi.org/10.1093/cid/cix669
World Health Organization. Global action plan on antimicrobial resistance. Fecha de consulta: 16 de octubre de 2022. Disponible en: https://www.who.int/antimicrobialresistance/publications/globalaction-plan/en/
Algunos artículos similares:
- Ingrid Yamile Pulido, José Ramón Mantilla, Emilia María Valenzuela, María Teresa Reguero, Elsa Beatriz González, Distribución de genes codificadores de β-lactamasas de espectro extendido en aislamientos de Klebsiella pneumoniae de hospitales de Bogotá, D.C., Colombia , Biomédica: Vol. 31 Núm. 1 (2011)
- Militza Guzmán, Elsa Salazar, Vicmaris Cordero, Ana Castro, Andreína Villanueva, Hectorina Rodulfo, Marcos De Donato, Multirresistencia a medicamentos y factores de riesgo asociados con infecciones urinarias por Escherichia coli adquiridas en la comunidad, Venezuela , Biomédica: Vol. 39 Núm. Sp. 1 (2019): Suplemento 1, Microbiología médica, mayo
- Eduardo Rodríguez-Noriega, Gerardo León-Garnica, Santiago Petersen-Morfín, Héctor Raúl Pérez-Gómez, Esteban González-Díaz, Rayo Morfín-Otero, La evolución de la resistencia bacteriana en México, 1973-2013 , Biomédica: Vol. 34 (2014): Abril, Suplemento 1, Resistencia bacteriana
- Ana María Perilla, Camilo González, Sandra Liliana Valderrama, Natasha Vanegas, Bibiana Chavarro, Luis Carlos Triana, José Roberto Támara, Carlos Arturo Álvarez, Neumonía necrosante por Staphylococcus aureus extrahospitalario resistente a la meticilina:reporte de dos casos en Colombia , Biomédica: Vol. 29 Núm. 4 (2009)
- David Felipe Briceño, Adriana Correa, Carlos Valencia, Julián Andrés Torres, Robinson Pacheco, María Camila Montealegre, Diego Ospina, María Virginia Villegas, Grupo de Resistencia Bacteriana Nosocomial, Actualización de la resistencia a antimicrobianos de bacilos Gram negativos aislados en hospitales de nivel III de Colombia: años 2006, 2007 y 2008 , Biomédica: Vol. 30 Núm. 3 (2010)
- José Ramón Mantilla, María Teresa Reguero, Elsa Beatriz González, Ibonne Ayde García, Aura Lucía Leal, Paula Andrea Espinal, Celia Alpuche, Ismael Alberto Valderrama, Martha Isabel Garzón, Narda María Olarte, Caracterización molecular de un brote por Klebsiella pneumoniae productora de CTX-M-12 en la unidad de cuidado intensivo neonatal de un hospital colombiano , Biomédica: Vol. 26 Núm. 3 (2006)
- Beatriz Millan, David Castro, Maria Araque, Bárbara Ghiglione, Gabriel Gutkind, ISCR1 asociado con genes blaCTX-M-1 y blaCTX-M-2 en plásmidos IncN e IncFIIA aislados en Klebsiella pneumoniae de origen hospitalario en Mérida, Venezuela , Biomédica: Vol. 33 Núm. 2 (2013)
- Edna Catering Rodríguez, Sandra Yamile Saavedra, Aura Lucía Leal, Carlos Álvarez, Narda Olarte, Alberto Valderrama, Sonia Isabel Cuervo, Javier Escobar, Diseminación de Klebsiella pneumoniae productoras de KPC-3 en hospitales de Bogotá durante un periodo de tres años , Biomédica: Vol. 34 (2014): Abril, Suplemento 1, Resistencia bacteriana
- Nancy Yaneth Flórez , Stefany Alejandra Arévalo , Edna Catering Rodríguez , Jaime Guerrero , Kelly Paola Valverde , Paula Lucía Díaz , Lucy Angeline Montaño, Doris Mabel Gartner , Carolina Duarte , Jaime Enrique Moreno, Brote de Salmonella enterica subsp. enterica serovar Give asociado con enfermedad transmitida por alimentos en Vichada, Colombia, 2015 , Biomédica: Vol. 41 Núm. 1 (2021)
- Johnny Durango, Germán Arrieta, Salim Mattar, Presencia de Salmonella spp. en un área del Caribe colombiano: un riesgo para la salud pública. , Biomédica: Vol. 24 Núm. 1 (2004)
Derechos de autor 2023 Biomédica

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |