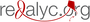Perfiles de expresión de los genes ERG11, MDR1 y AFR1 en Cryptococcus neoformans var. grubii aislados de pacientes con VIH
Resumen
Introducción. El fluconazol es el antifúngico más utilizado para la prevención y el tratamiento de infecciones causadas por el género Cryptococcus, agente etiológico de la criptococosis. La resistencia al fluconazol en los aislamientos de Cryptoccocus neoformans puede hacer fracasar el tratamiento y generar recaídas de la infección.
Objetivo. Evaluar los perfiles de expresión de los genes AFR1, MDR1 y ERG11 en aislamientos clínicos de C. neoformans var. grubii, durante la respuesta in vitro a la inducción con fluconazol.
Materiales y métodos. Se estudiaron 14 aislamientos de C. neoformans var. grubii provenientes de pacientes con HIV, de los cuales 6 eran sensibles al fluconazol y 8 presentaban sensibilidad disminuida. Los niveles de expresión de los genes ERG11, AFR1 y MDR1 se determinaron mediante PCR en tiempo real.
Resultados. Los aislamientos resistentes al fluconazol mostraron sobreexpresión de los genes AFR1 y MDR1, mientras que la expresión de los fenotipos de resistencia evaluados se mantuvo homogénea en ERG11, en todos los aislamientos de C. neoformans var. grubii.
Conclusiones. La sobreexpresión de los genes AFR1 y MDR1 que codifican las bombas de eflujo, contribuye a la resistencia al fluconazol en los aislamientos estudiados. Sin embargo, los patrones de resistencia que se registran en este hongo, sumado a los casos de recaídas en pacientes con HIV, no pueden atribuirse únicamente a los casos de resistencia por exposición al fármaco. Otros mecanismos podrían también estar involucrados en este fenómeno, como la resistencia emergente (resistencia mediante otros genes ERG) y la heterorresistencia, los cuales deben ser estudiados en estos aislamientos.
Descargas
Referencias bibliográficas
Perfect JR, Dismukes WE, Dromer F, Goldman DL, Graybill JR, Hamill RJ, et al. Clinical practice guidelines for the management of cryptococcal disease: 2010 update by the infectious diseases society of America. Clin Infect Dis. 2010;50:291-322. https://doi.org/10.1086/649858
Rajasingham R, Smith RM, Park BJ, Jarvis JN, Govender NP, Chiller TM, et al. Global burden of disease of HIV-associated cryptococcal meningitis: An updated analysis. Lancet Infect Dis. 2017;17:873-81. https://doi.org/10.1016/S1473-3099(17)30243-8
Escandón P, Lizarazo J, Agudelo CI, Castañeda E. Cryptococcosis in Colombia: Compilation and analysis of data from laboratory-based surveillance. J Fungi (Basel). 2018;4(1). https://doi.org/10.3390/jof4010032
Kwon-Chung KJ, Bennett JE, Wickes BL, Meyer W, Cuomo CA, Wollenburg KR, et al. The case for adopting the “Species Complex” nomenclature for the etiologic agents of cryptococcosis. mSphere. 2017;2:e00357-16. https://doi.org/10.1128/mSphere.00357-16
Hagen F, Khayhan K, Theelen B, Kolecka A, Polacheck I, Sionov E, et al. Recognition of seven species in the Cryptococcus gattii/Cryptococcus neoformans species complex. Fungal Genet Biol. 2015;78:16-48. https://doi.org/10.1016/j.fgb.2015.02.009
Idnurm A, Lin X. Rising to the challenge of multiple Cryptococcus species and the diseases they cause. Fungal Genet Biol. 2015;78:1-6. https://doi.org/10.1016/j.fgb.2015.05.002
Serna-Espinosa BN, Guzmán-Sanabria D, Forero-Castro M, Escandón P, Sánchez-Quitian ZA. Environmental Status of Cryptococcus neoformans and Cryptococcus gattii in Colombia. J Fungi (Basel). 2021;7. https://doi.org/10.3390/jof7060410
Gushiken AC, Saharia KK, Baddley JW. Cryptococcosis. Infect Dis Clin North Am. 2021;35:493-514. https://doi.org/10.1016/j.idc.2021.03.012
World Health Organization. Guidelines for the diagnosis, prevention and management of cryptococcal disease in HIV-infected adults, adolescents and ahildren: Supplement to the 2016 Consolidated guidelines on the use of antiretroviral drugs for treating and preventing HIV infection. Geneva: World Health Organization; 2018. p. 51.
Bicanic T, Harrison T, Niepieklo A, Dyakopu N, Meintjes G. Symptomatic relapse of HIV-associated cryptococcal meningitis after initial fluconazole monotherapy: The role of fluconazole resistance and immune reconstitution. Clin Infect Dis. 2006;43:1069-70. https://doi.org/10.1086/507895
Maziarz EK, Perfect JR. Cryptococcosis. Infect Dis Clin North Am. 2016;30:179-206. https://doi.org/10.1016/j.idc.2015.10.006
Agudelo CA, Muñoz C, Ramírez A, Tobón AM, de Bedout Bact C, Cano LE, et al. Response to therapy in patients with cryptococcosis and AIDS: Association with in vitro susceptibility to fluconazole. Rev Iberoam Micol. 2015;32:214-20. https://doi.org/10.1016/j.riam.2014.07.006
Cheong JWS, McCormack J. Fluconazole resistance in cryptococcal disease: Emerging or intrinsic? Med Mycol. 2013;51:261-9. https://doi.org/10.3109/13693786.2012.715763
Basso LR, Gast CE, Bruzual I, Wong B. Identification and properties of plasma membrane azole efflux pumps from the pathogenic fungi Cryptococcus gattii and Cryptococcus neoformans. J Antimicrob Chemother. 2015;70:1396-407. https://doi.org/10.1093/jac/dku554
Venkateswarlu K, Taylor M, Manning NJ, Rinaldi MG, Kelly SL. Fluconazole tolerance in clinical isolates of Cryptococcus neoformans. Antimicrob Agents Chemother. 1997;41:748-51. https://doi.org/10.1128/AAC.41.4.748
Thornewell SJ, Peery RB, Skatrud PL. Cloning and characterization of CneMDR1: A Cryptococcus neoformans gene encoding a protein related to multidrug resistance proteins. Gene. 1997;201:21-9. https://doi.org/10.1016/s0378-1119(97)00421-6
Posteraro B, Sanguinetti M, Sanglard D, La Sorda M, Boccia S, Romano L, et al. Identification and characterization of a Cryptococcus neoformans ATP binding cassette (ABC) transporter-encoding gene, CnAFR1, involved in the resistance to fluconazole. Mol Microbiol. 2003;47:357-71. https://doi.org/10.1046/j.1365-2958.2003.03281.x
Sanguinetti M, Posteraro B, La Sorda M, Torelli R, Fiori B, Santangelo R, et al. Role of AFR1, an ABC transporter-encoding gene, in the in vivo response to fluconazole and virulence of Cryptococcus neoformans. Infect Immun. 2006;74:1352-9. https://doi.org/10.1128/IAI.74.2.1352-1359.2006
Espinel-Ingroff A, Aller AI, Canton E, Castañón-Olivares LR, Chowdhary A, Cordoba S, et al. Cryptococcus neoformans-Cryptococcus gattii species complex: An international study of wild-type susceptibility endpoint distributions and epidemiological cutoff values for fluconazole, itraconazole, posaconazole, and voriconazole. Antimicrob Agents Chemother. 2012;56:5898–906. https://doi.org/10.1128/AAC.01115-12
Clinical and Laboratory Standards Institute. Values TDIE. Epidemiological cutoff values for antifungal susceptibility testing. Fecha de consulta: 5 de octubre de 2022. Disponible en: https://clsi.org/media/1934/m59ed2_sample-updated.pdf
Procop GW, Alexander BD, Dufresne PJ, Fuller J, Ghannoum MA, Hanson KE, et al. Method for antifungal disk diffusion susceptibility testing of yeasts. Fecha de consulta: 5 de octubre de 2022. Disponible en: https://clsi.org/media/2634/m44ed3_sample.pdf
Livak KJ, Schmittgen TD. Analysis of relative gene expression data using real-time quantitative PCR and the 2(-Delta Delta C(T)) method. Methods. 2001;25:402-8. https://doi.org/10.1006/meth.2001.1262
Gast CE, Basso LR, Bruzual I, Wong B. Azole resistance in Cryptococcus gattii from the Pacific Northwest: Investigation of the role of ERG11. Antimicrob Agents Chemother. 2013;57:5478-85. https://doi.org/10.1128/AAC.02287-12
Cannon RD, Lamping E, Holmes AR, Niimi K, Baret PV, Keniya MV, et al. Efflux-mediated antifungal drug resistance. Clin Microbiol Rev. 2009;22:291-321. https://doi.org/10.1128/CMR.00051-08
Li M, Liao Y, Chen M, Pan W, Weng L. Antifungal susceptibilities of Cryptococcus species complex isolates from AIDS and non-AIDS patients in Southeast China. Braz J Infect Dis. 2012;16:175-9. https://doi.org/10.1016/s1413-8670(12)70301-x
Wu SY, Kang M, Liu Y, Chen ZX, Xiao YL, He C, et al. Molecular epidemiology and antifungal susceptibilities of Cryptococcus species isolates from HIV and non-HIV patients in Southwest China. Eur J Clin Microbiol Infect Dis. 2021;40:287-95. https://doi.org/10.1007/s10096-020-04013-4
Bongomin F, Oladele RO, Gago S, Moore CB, Richardson MD. A systematic review of fluconazole resistance in clinical isolates of Cryptococcus species. Mycoses. 2018;61:290-7. https://doi.org/10.1111/myc.12747
Liu JY, Shi C, Wang Y, Li WJ, Zhao Y, Xiang MJ. Mechanisms of azole resistance in Candida albicans clinical isolates from Shanghai, China. Res Microbiol. 2015;166:153-61. https://doi.org/10.1016/j.resmic.2015.02.009
Florio AR, Ferrari S, De Carolis E, Torelli R, Fadda G, Sanguinetti M, et al. Genome-wide expression profiling of the response to short-term exposure to fluconazole in Cryptococcus neoformans serotype A. BMC Microbiol. 2011;11:97. https://doi.org/10.1186/1471-2180-11-97
Chang M, Sionov E, Khanal LA, Kwon-Chung KJ, Chang YC. Roles of Three Cryptococcus neoformans and Cryptococcus gattii efflux pump-coding genes in response to drug treatment. Antimicrob Agents Chemother. 2018;62:e01751-17. https://doi.org/10.1128/AAC.01751-17
Sionov E, Chang YC, Garraffo HM, Kwon-Chung KJ. Heteroresistance to fluconazole in Cryptococcus neoformans is intrinsic and associated with virulence. Antimicrob Agents Chemother. 2009;53:2804–15. https://doi.org/10.1128/AAC.00295-09
Algunos artículos similares:
- Jairo Lizarazo, Melva Linares, Catalina de Bedout, Ángela Restrepo, Clara Inés Agudelo, Elizabeth Castañeda, Grupo Colombiano para el Estudio de la Criptococosis, Estudio clínico y epidemiológico de la criptococosis en Colombia: resultados de nueve años de la encuesta nacional, 1997-2005 , Biomédica: Vol. 27 Núm. 1 (2007)
- Efraín Guillermo Sánchez , David Acosta , Juan Álvarez, Gabriela Sánchez , Julio García-Casallas, Criptococosis diseminada por terapia biológica, se debe gestionar el riesgo , Biomédica: Vol. 42 Núm. 2 (2022)
- Sunny Sánchez, Dolores Zambrano, Maylen García, César Bedoya, Carlos Fernández, María Teresa Illnait-Zaragozí, Caracterización molecular de los aislamientos de Cryptococcus neoformans de pacientes con HIV, Guayaquil, Ecuador , Biomédica: Vol. 37 Núm. 3 (2017)
- Patricia Escandón, Popchai Ngamskulrungroj, Wieland Meyer, Elizabeth Castañeda, Determinación in vitro de la pareja sexual en aislamientos del complejo Cryptococcus neoformans , Biomédica: Vol. 27 Núm. 2 (2007)
- Carmelo José Espinosa, Jorge Alberto Cortés, Juan Sebastián Castillo, Aura Lucía Leal, Revisión sistemática de la resistencia antimicrobiana en cocos Gram positivos intrahospitalarios en Colombia , Biomédica: Vol. 31 Núm. 1 (2011)
- Mercedes Figueroa, Armando Cortés, Álvaro Pazos, Luis Eduardo Bravo, Sensibilidad in vitro a amoxicilina y claritromicina de Helicobacter pylori obtenido de biopsias gástricas de pacientes en zona de bajo riesgo para cáncer gástrico , Biomédica: Vol. 32 Núm. 1 (2012)
- Alexandra Castañeda, Sandra Huérfano, Maria Claudia Rodríguez, Elizabeth Castañeda, Recuperación de Cryptococcus neoformans var. gattii serotipo C a partir de detritos de almendros , Biomédica: Vol. 21 Núm. 1 (2001)
- Alexandra Castañeda, Elizabeth Castañeda, Aislamiento de especies de Cryptococcus asociadas con Eucalyptus en un parque de Bogotá , Biomédica: Vol. 21 Núm. 1 (2001)
- Elsa De La Cadena, Christian José Pallares, Juan Carlos García-Betancur, Jessica A. Porras, María Virginia Villegas, Actualización sobre la resistencia antimicrobiana en instituciones de salud de nivel III y IV en Colombia entre enero del 2018 y diciembre del 2021 , Biomédica: Vol. 43 Núm. 4 (2023)
- Claudia Parra, John Mario González, Elizabeth Castañeda, Susana Fiorentino, Producción de anticuerpos IgG1 anti-glucuronoxilomanana en ratones resistentes a la criptococosis , Biomédica: Vol. 25 Núm. 1 (2005)
Derechos de autor 2022 Biomédica

Esta obra está bajo una licencia internacional Creative Commons Atribución 4.0.
| Estadísticas de artículo | |
|---|---|
| Vistas de resúmenes | |
| Vistas de PDF | |
| Descargas de PDF | |
| Vistas de HTML | |
| Otras vistas | |