Monkeypox virus genome sequence from an imported human case in Colombia
Abstract
Introduction: Monkeypox virus (MPXV) is an enveloped double-stranded DNA virus with a genome of approximately 197.209 bp. The current classification divides MPXV into three clades: Clade I (Central African or Congo Basin clade) and clades IIa and IIb (West African clades).
Objective: To report the complete genome and phylogenetic analysis of a human monkeypox case detected in Colombia.
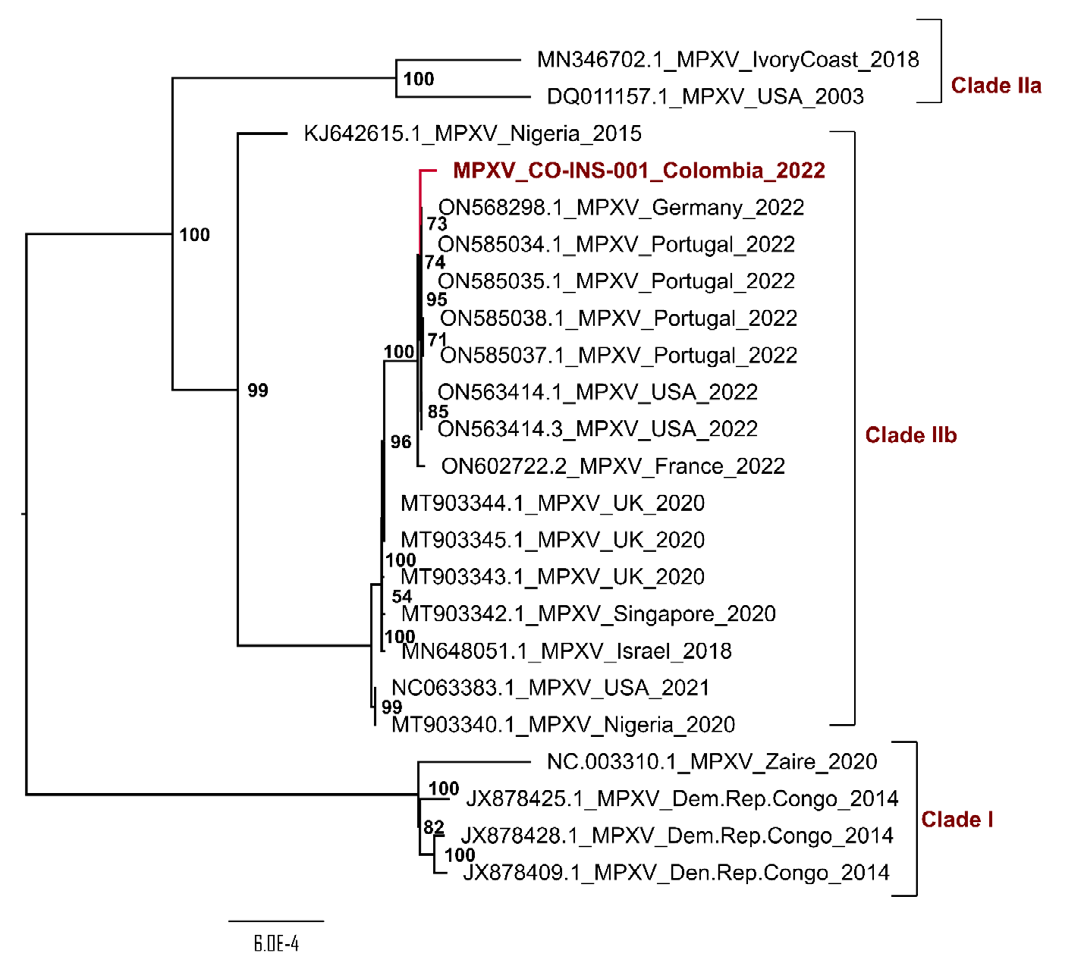
Materials and methods: Exudate from vesicular lesions was obtained from a male patient with recent travel history to Spain. A direct genomic approach was implemented in which total DNA from the sample was purified through a column-based method, followed by sequencing on the Nanopore GridION. Reads were aligned against the MPXV reference genome using minimap2 v.2.24 and phylogenetic inference was performed using maximum likelihood estimation.
Results: A total of 11.951 reads mapped directly to a reference genome with 96.8% of coverage (190.898 bp).
Conclusion: Phylogenetic analysis of the MPXV circulating in Colombia demonstrated its close relationship to clade IIb responsible for the multi-country outbreak in 2022.
Downloads
References
Yinka-Ogunleye A, Aruna O, Dalhat M, Dalhat M, Ogoina D, McCollum A, et al . Outbreak of human monkeypox in Nigeria in 2017-18: A clinical and epidemiological report. Lancet Infect Dis. 2019;19:872-9. https://doi.org/10.1016/S1473-3099(19)30294-4
World Health Organization. Multi-country monkeypox outbreak in non-endemic countries. Geneva; WHO; 2022. Accessed: June 28, 2022. Available from: https://www.who.int/emergencies/disease-outbreak-news/item/2022-DON385
Happi C, Adetifa I, Mbala P, Njouom R, Nakoune E, Happi A, et al. Urgent need for a non-discriminatory and non-stigmatizing nomenclature for monkeypox virus. PLoS Biol. 2022;20:e3001769. https://doi.org/10.1371/journal.pbio.3001769
Likos AM, Sammons SA, Olson VA, Frace AM, Li Y, Olsen-Rasmussen M, et al. A tale of two clades: Monkeypox viruses. J Gen Virol. 2005;86:2661-72. https://doi.org/10.1099/vir.0.81215-0
Centers for Disease Control and Prevention. Monkeypox Outbreak Global Map USA: Atlanta, GA:CDC;2022. Accessed: July 5, 2022. Available from: https://www.cdc.gov/poxvirus/monkeypox/response/2022/world-map.html
Li H. Minimap2: Pairwise alignment for nucleotide sequences. Bioinformatics. 2018;34:3094-100. https://doi.org/10.1093/bioinformatics/bty191
Nguyen LT, Schmidt HA, von Haeseler A, Minh BQ. IQ-TREE: A fast and effective stochastic algorithm for estimating maximum-likelihood phylogenies. Mol Biol Evol. 2015;32:268-74. https://doi.org/10.1093/molbev/msu3
Li H, Handsaker B, Wysoker A, Fennell T, Ruan J, Homer N, et al. The Sequence Alignment/Map format and SAMtools. Bioinformatics. 2009;25:2078-9. https://doi.org/10.1093/bioinformatics/btp352
Kugelman JR, Johnston SC, Mulembakani PM, Kisalu N, Lee MS, Koroleva G, et al. Genomic variability of monkeypox virus among humans, Democratic Republic of the Congo. Emerg Infect Dis. 2014;20:232-9. https://doi.org/10.3201/eid2002.130118
Patrono LV, Pléh K, Samuni L, Ulrich M, Röthemeier C, Sachse A, et al. Monkeypox virus emergence in wild chimpanzees reveals distinct clinical outcomes and viral diversity. Nat Microbiol. 2020;5:955-65. https://doi.org/10.1038/s41564-020-0706-0
Likos AM, Sammons SA, Olson VA, Frace AM, Li Y, Olsen-Rasmussen M, et al. A tale of two clades: Monkeypox viruses. J Gen Virol. 2005;86:2661-72. https://doi.org/10.1099/vir.0.81215-0
Nakazawa Y, Mauldin MR, Emerson GL, Reynolds MG, Lash RR, Gao J, et al. A phylogeographic investigation of African monkeypox. Viruses. 2015;7:2168-84. https://doi.org/10.3390/v7042168
Antwerpen M, Lang D, Zange S, Walter M, Woelfel R. First German genome sequence of Monkeypox virus associated to multi-country outbreak in May 2022. Virological.org. 2022.
Accessed: July 12, 2022. Available from: https://virological.org/t/first-german-genomesequence-of-monkeypox-virus-associated-to-multi-country-outbreak-in-may-2022/812
Isidro J, Borges V, Pinto M, Sobral D, Santos JD, Nunes A, et al. Phylogenomic characterization and signs of microevolution in the 2022 multi-country outbreak of monkeypox virus. Nat Med. 2022;28:1569-72. https://doi.org/10.1038/s41591-022-01907-y
Gigante CM, Korber B, Seabolt MH, Wilkins K, Davidson W, Rao AK, et al. Multiple lineages of Monkeypox virus detected in the United States, 2021-2022. bioRxiv. 2022. https://doi.org/10.1101/2022.06.10.495526
Croville G, Walch M, Guérin J, Mansuy J, Pasquier C, Izopet J. First French draft genome sequence of Monkeypox virus, May 2022. Virological.org. 2022. Accessed: July 15, 2022. Availble from: https://virological.org/t/first-french-draft-genome-sequence-of-monkeypoxvirus-may-2022/819
Mauldin MR, McCollum AM, Nakazawa YJ, Mandra A, Whitehouse ER, Davidson W, et al. Exportation of monkeypox virus from the African continent. J Infect Dis. 2022;225:1367-76. https://doi.org/10.1093/infdis/jiaa559
Cohen-Gihon I, Israeli O, Shifman O, Erez N, Melamed S, Paran N, et al. Identification and whole-genome sequencing of a Monkeypox virus strain isolated in Israel. Microbiol Resour Announc. 2020;9:e01524-19. https://doi.org/10.1128/MRA.01524-19
Shchelkunov SN, Totmenin AV, Babkin IV, Safronov PF, Ryazankina OI, Petrov NA, et al. Human monkeypox and smallpox viruses: Genomic comparison. FEBS Lett. 2021;509:66-70. https://doi.org/10.1016/s0014-5793(01)03144-1
Claro IM, Romano CM, Candido DD, Lima EL, Lindoso JAL, Ramundo MS, et al. Shotgun metagenomic sequencing of the first case of monkeypox virus in Brazil, 2022. Rev Inst Med Trop Sao Paulo. 2022;64:e48. https://doi.org/10.1590/S1678-9946202264048
Isidro J, Borges V, Pinto M, Sobral D, Santos JD, Nunes A, et al. Phylogenomic characterization and signs of microevolution in the 2022 multi-country outbreak of monkeypox virus. Nat Med. 2022;28:1569-72. https://doi.org/10.1038/s41591-022-01907-y
Some similar items:
- Daniela Arturo-Terranova, Sebastián Giraldo-Ocampo, Andrés Castillo , Molecular characterization of Epstein-Barr virus variants detected in the oral cavity of adolescents in Cali, Colombia , Biomedica: Vol. 40 No. Supl. 1 (2020): Mayo, Infecciones en el trópico
- Andrés Gómez-Palacio, Juan Suaza-Vasco, Sandra Castaño, Omar Triana, Sandra Uribe, Aedes albopictus (Skuse, 1894) infected with the American-Asian genotype of dengue type 2 virus in Medellín suggests its possible role as vector of dengue fever in Colombia , Biomedica: Vol. 37 No. Sup. 2 (2017): Suplemento 2, Entomología médica, 2017
- Katherine Laiton-Donato, José A. Usme-Ciro, Angélica Rico, Lissethe Pardo, Camilo Martínez, Daniela Salas, Susanne Ardila, Andrés Páez, Phylogenetic analysis of Chikungunya virus in Colombia: Evidence of purifying selection in the E1 gene , Biomedica: Vol. 36 (2016): Suplemento 2, Enfermedades virales
- Luis Eduardo Echeverría, Clara Saldarriaga, Sebastián Campbell-Quintero, Lisbeth Natalia Morales-Rodríguez, Juan David López-Ponce de León, Andrés Felipe Buitrago, Erika Martínez-Carreño, Jorge Alberto Sandoval-Luna, Alexis Llamas, Gustavo Adolfo Moreno-Silgado, Julián Vanegas-Eljach, Nelson Eduardo Murillo-Benítez, Ricardo Gómez-Paláu, Alex Arnulfo Rivera-Toquica, Juan Esteban Gómez-Mesa, RECOLFACA research group, Diabetes mellitus in patients with heart failure and effect modification of risk factors for short-term mortality: An observational study from the Registro Colombiano de Falla Cardíaca (RECOLFACA) , Biomedica: Vol. 44 No. Sp. 1 (2024): Enfermedades crónicas no transmisibles
- Raúl Murillo, Ricardo Cendales, Carolina Wiesner, Marion Piñeros, Sandra Tovar, Effectiveness of cytology-based cervical cancer screening in the Colombian health system , Biomedica: Vol. 29 No. 3 (2009)
- Sandra Lorena Girón, Julio César Mateus, Fabián Méndez, Impact of an open waste disposal site on the occurrence of respiratory symptoms and on health care costs of children , Biomedica: Vol. 29 No. 3 (2009)
- José Joaquín Carvajal, Ligia Inés Moncada, Mauricio Humberto Rodríguez, Ligia del Pilar Pérez, Víctor Alberto Olano, Characterization of Aedes albopictus (Skuse, 1894) (Diptera:Culicidae) larval habitats near the Amazon River in Colombia , Biomedica: Vol. 29 No. 3 (2009)
- Andrés Páez, Gloria Rey, Carlos Agudelo, Alvaro Dulce, Edgar Parra, Hernando Díaz-Granados, Damaris Heredia, Luis Polo, Outbreak of urban rabies transmitted by dogs in Santa Marta, northern Colombia , Biomedica: Vol. 29 No. 3 (2009)
- Patricia Escobar, Katherine Paola Luna, Indira Paola Hernández, César Mauricio Rueda, María Magdalena Zorro, Simon L. Croft, In vitro susceptibility of Trypanosoma cruzi strains from Santander, Colombia, to hexadecylphosphocholine (miltefosine), nifurtimox and benznidazole , Biomedica: Vol. 29 No. 3 (2009)
- María Fernanda Gutiérrez, Sandra Moreno, Mónica Viviana Alvarado, Andrea Bermúdez, DNA sequence analysis indicates human origin of rotavirus and hepatitis A virus strains from western Colombia , Biomedica: Vol. 29 No. 2 (2009)
Copyright (c) 2022 Biomedica

This work is licensed under a Creative Commons Attribution 4.0 International License.
| Article metrics | |
|---|---|
| Abstract views | |
| Galley vies | |
| PDF Views | |
| HTML views | |
| Other views | |