Molecular characterization of multiresistant enterobacteria in two departments of the Peruvian jungle
Abstract
Introduction. The emergence of multiresistant enterobacteria producing extendedspectrum beta-lactamase (ESBL) in outpatients with urinary tract infections represents a public health problem in Perú.
Objectives. To characterize multiresistant enterobacteria isolated from patients diagnosed with urinary tract infection in two Peruvian jungle departments using molecular techniques.
Materials and methods. We conducted a descriptive, observational, and retrospective study of 61 urine culture isolates from two departments in the Peruvian jungle during 2017-2018. Resistance profiles were identified using the MicroScan™ automated system and a conventional polymerase chain reaction (PCR) was used for the detection of blaCTX-M, blaTEM and blaSHV genes.
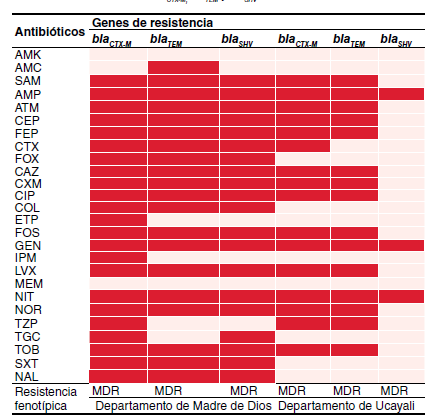
Results. The most common positive ESBL enterobacteria for each department were Escherichia coli in Madre de Dios (10/40; 25%) and Ucayali (16/21; 76.2%). Gene blaCTX-M was the most prevalent in both departments (25/61; 41%), followed by blaTEM (15/61; 24.6%), and blaSHV (10/61; 16.4%). As for the antimicrobial susceptibility profile, we detected resistance levels of 72.6% for ampicillin, 82.3% for cephalothin, and 88.7% for nitrofurantoin.
Conclusions. BLEE-producing multi-resistant enterobacteria strains in both departments were 57.4% and blaCTX-M was the most common gene.
Downloads
References
Organización Mundial de la Salud. Resistencia a los antibióticos 2021. Fecha de consulta: 3 de mayo del 2021. Disponible en: https://bit.ly/3h0V2Ch
Yábar MN, Curi-Pesantes B, Torres CA, Calderón-Anyosa R, Riveros M, Ochoa TJ. Multiresistance and factors associated with the presence of extended-spectrum betalactamases in Escherichia coli strains isolated from urine culture. Rev Perú Med Exp Salud Pública. 2017;34:660-5. https://doi.org/10.17843/rpmesp.2017.344.2922
Sbiti M, Lahmadi K, Louzi L. Profil épidémiologique des entérobactéries uropathogènes productrices de bêta-lactamases à spectre élargi. Pan Afr Med J. 2017;28:29. https://doi.org/10.11604/pamj.2017.28.29.11402
Rada AM, Hernández GC, Restrepo E, Villegas MV. Distribución y caracterización molecular de betalactamasas en bacterias Gram negativas en Colombia, 2001-2016. Biomédica. 2019;39:199-220. https://doi.org/10.7705/biomedica.v39i3.4351
Díaz MJ, Amar PW, Angulo LM, BS. Prevalencia de Escherichia coli productor de betalactamasas de espectro extendido (BLEE) y otras resistencias en urocultivos en un hospital general de Ica, Perú. Revista Médica Panacea. 2015;5:20-4. https://doi.org/10.35563/rmp.v5i1.68
Marcos-Carbajal P, Salvatierra G, Yareta J, Pino J, Vásquez N, Díaz P, et al. Caracterización microbiológica y molecular de la resistencia antimicrobiana de Escherichia coli uropatógenas de hospitales públicos peruanos. Rev Peru Med Exp Salud Pública. 2021;38:119-23. https://doi.org/10.17843/ rpmesp.2021.381.6182
Red Latinoamericana de Vigilancia de la Resistencia a los Antimicrobianos (ReLAVRA). Magnitud y tendencias de la resistencia a los antimicrobianos en Latinoamérica. RELAVRA 2014, 2015, 2016. Fecha de consulta: 3 de mayo del 2021. Disponible en: https://bit.ly/2R4PqfT
Mostajo AG, Díaz CA, Morón PR. Frecuencia de infección del tracto urinario intrahospitalaria por enterobacterias productoras de betalactamasas de espectro extendido y factores asociados en un hospital nacional. Revista de la Sociedad Peruana de Medicina Interna. 2015;28:113-20.
Olarte LT, Cáceres GD, Cortés JA. Nuevas cefalosporinas. Rev Chil Infectol. 2018;35:465-75. https://doi.org/10.4067/s0716-10182018000500465
Instituto Geográfico Nacional, 1989. Atlas del Perú. Lima-Perú. Fecha de consulta: 3 de mayo del 2021. Disponible en: https://bit.ly/3y8M2kA
Astete S, Madrid L, Fukuda F, Buckley A, Meritens D, Menchola JV. Sensibilidad antibiótica de los gérmenes causantes de infecciones urinarias en pacientes ambulatorios en el Hospital Nacional Arzobispo Loayza. Rev Soc Per Med Inter. 2004;17:5-8.
Beckman C. MicroScan. Manual de procedimiento para microorganismos gramnegativos, LabPro multirregional ≥ V4.42. U.S.A. August 2019. Fecha de consulta: 3 de mayo de 2021. Disponible en: https://bit.ly/307u5Tq
Clinical & Laboratory Standards Institute. M100: Performance Standards for Antimicrobial Susceptibility Testing, 30th edition. Clinical & Laboratory Standards Institute. Fecha de consulta: 1 de mayo de 2021. Disponible en: https://clsi.org/standards/products/microbiology/documents/m100/
Kiratisin P, Apisarnthanarak A, Laesripa C, Saifon P. Molecular characterization and epidemiology of extended-spectrum-beta-lactamase-producing Escherichia coli and Klebsiella pneumoniae isolates causing health care-associated infection in Thailand, where the CTX-M family is endemic. Antimicrob Agents Chemother. 2008;52:2818-24. https://doi.org/10.1128/AAC.00171-08
Arce Z, Núñez JL, Clavo RF, Valverde DF. Detección del gen CTX-M en cepas de Escherichia coli productoras de β-lactamasas de espectro extendido procedentes del Hospital Regional de Lambayeque; Chiclayo-Perú: noviembre 2012-julio 2013. Revista Del Cuerpo Médico Hospital Nacional Almanzor Aguinaga Asenjo. 2013;6:12-5.
Morejón-García M. Betalactamasas de espectro extendido. Revista Cubana de Medicina. 2013;52:272-80.
García MH, Valdez CA, Porta TV. Detección de los genes de β-lactamasas blaTEM, blaSHV y blaCTX-M en aislamientos de Escherichia coli comunitarios. Revista Científica de la Facultad de Ciencias Químicas y Farmacia. 2019;28:45-56.
Galván F, Agapito J, Bravo N, Lagos J, Tamariz J. Caracterización fenotípica y molecular de Escherichia coli productoras de β-Lactamasas de espectro extendido en pacientes ambulatorios de Lima, Perú. Revista Médica Herediana. 2016;27:22-9.
Alarcón NC, González JF, Sarabia RL, Sánchez JS, Rosas MR. Caracterización de β-lactamasas de espectro extendido producidas por Escherichia coli de infecciones del tracto urinario adquiridas en la comunidad de Chilpancingo. Guerrero, México. 2014. Fecha de consulta: 1 de mayo de 2021. Disponible en: http://tlamati.uagro.mx/t51/t512.pdf
Rodríguez-Villalobos H, Bogaerts P, Berhin C, Bauraing C, Deplano A, Montesinos I, et al. Trends in production of extended-spectrum β-lactamases among Enterobacteriaceae of clinical interest: Results of a nationwide survey in Belgian hospitals. J Antimicrob Chemother. 2011;66:37-47. https://doi.org/10.1093/jac/dkq388
Suárez-Trueba B, Milián-Samper Y, Espinosa-Rivera F, Hart-Casares M, Llanes-Rodríguez N, Martínez-Batista ML. Susceptibilidad antimicrobiana y mecanismos de resistencia de Escherichia coli aisladas a partir de urocultivos en un hospital de tercer nivel. Revista Cubana de Medicina. 2014;53:3-13.
Some similar items:
- María Carolina Sanabria, Gerardo Muñoz, Clara Inés Vargas, Mutations in the BRCA1 gene (185delAG and 5382insC) are not present in any of the 30 breast cancer patients analyzed from eastern Colombia , Biomedica: Vol. 29 No. 1 (2009)
- Brian Alejandro Suárez, Claudia Liliana Cuervo, Concepción Judith Puerta, The intergenic region of the histone h2a gene supports two major lineages of Trypanosoma rangeli , Biomedica: Vol. 27 No. 3 (2007)
- Patricia Escandón, Popchai Ngamskulrungroj, Wieland Meyer, Elizabeth Castañeda, In vitro mating of Colombian isolates of the Cryptococcus neoformans species complex , Biomedica: Vol. 27 No. 2 (2007)
- Isabel Mayorga-Fayad, Gloria I. Lafaurie, Adolfo Contreras, Diana M. Castillo, Alexandra Barón, María del Rosario Aya, Subgingival microbiota in chronic and aggressive periodontitis in Bogotá, Colombia: an epidemiological approach , Biomedica: Vol. 27 No. 1 (2007)
- Winston Rojas, Maria Antonieta Caro, Juan Guillermo Lopera, Omar Triana, Juan Carlos Dib, Gabriel Bedoya, Analysis of polymorphisms in the trypanothione reductase and cruzipain genes in Colombian strains of Trypanosoma cruzi , Biomedica: Vol. 27 No. 1esp (2007): Enfermedad de Chagas
- Juan Carlos Herrera, Luis Fernando Isaza, José Luis Ramírez, Gonzalo Vásquez, Carlos Mario Muñetón, Detection of chromosome 17 aneuplody and TP53 gene deletion in a broad variety of solid tumors by dual-color fluorescence in situ hybridization (FISH) , Biomedica: Vol. 30 No. 3 (2010)
- Jessika Consuegra, Sonia Jakeline Gutiérrez, Adriana Jaramillo, Ignacio Sanz, Gilberto Olave, Jorge Enrique Soto, Carlos Valencia, Adolfo Contreras, Enteric Gram negative rods and unfermented of glucose bacteria in patients with peri-implant disease , Biomedica: Vol. 31 No. 1 (2011)
- Martha Lucía Serrano, Juan José Yunis, Identification of three new mutations in the RB1 gene in patients with sporadic retinoblastoma in Colombia , Biomedica: Vol. 33 No. 1 (2013)
- Andrea Gómez, Gustavo Salguero, Herbert García, Fabio Aristizábal, Oscar Gutiérrez, Luis Alberto Angel, Jorge Padrón, Carlos Martínez, Humberto Martínez, Omar Malaver, Rosa Barvo, Alejandro Giraldo, Detection mutations in the DNA mismatch repair genes of hMLH1 and hMSH2 genes in Colombian families with suspicion of hereditary non-polyposis colorectal carcinoma (Lynch syndrome). , Biomedica: Vol. 25 No. 3 (2005)
- Leidy González, Jorge Alberto Cortés, Systematic review of antimicrobial resistance in Enterobacteriaceae isolates from Colombian hospitals , Biomedica: Vol. 34 No. 2 (2014)
| Article metrics | |
|---|---|
| Abstract views | |
| Galley vies | |
| PDF Views | |
| HTML views | |
| Other views | |